(なぜローザ)清成さんの名前が出たので、少し寄り道しておきましょうかね。
>>
理研CDBの研究室主宰者22名、連名でSTAP論文問題に対する声明を発表
2014年3月14日 19時12分 マイナビニュース
理化学研究所 発生・再生科学総合研究センター(理研CDB)の研究室主宰者22名は3月14日、連名で英科学雑誌「Nature」に掲載されたSTAP細胞に関する論文に関連して生じているさまざまな問題に対する声明を発表した。
今回の声明を出したのは以下の22名の研究室主宰者(声明文記載順)。
・戎家美紀・永樂元次・榎本秀樹・藤原裕展・古田泰秀・花嶋かりな・平谷伊智朗・今井猛・猪股秀彦・北島智也・清成寛・清末優子・倉永英里奈・Raj Ladher・森本充・森下喜弘・西村隆史・Guojun Sheng・柴田達夫・高橋政代・上田泰己・Yu-Chiun Wang
なお、声明の原文は以下のとおり。
今回Nature誌に掲載されたSTAP論文に関連して生じている様々な問題に対し、下記に名を連ねる者は、同じ理研CDBの研究室主宰者として大変深刻に受け止め、憂慮しております。わたしたちは同じ研究者として科学の公正性を回復、担保するためのあらゆる努力を払う所存です。また、理化学研究所における研究活動が社会の信頼無くしては成り立たないことを十分に自覚しております。我々は、社会及び研究者コミュニティーに対して最大限誠実な行動を取ることをお約束すると共に、高い規範の下に研究活動に励み、その成果を社会に還元すべく不断の努力を続けることをここに表明いたします。清成寛さんは、若山さんが協力を断ったために、小保方さんの再現実験でキメラ胚に移植する実験を代わりにしてくれた人です。

八細胞期胚へのES細胞のインジェクション写真ですね。彼はすべてを分かってると思いますね。彼はES細胞も、そして小保方さんが作ったSTAP酸浴細胞も両方ともインジェクトしたことがあるんです。
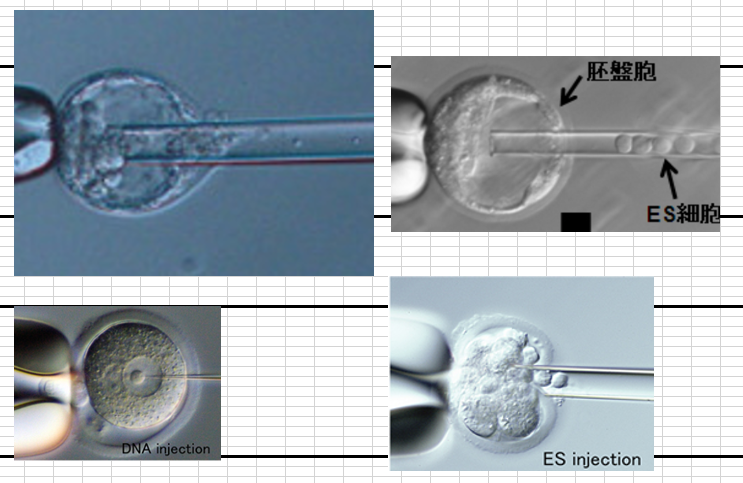
左下のマイクロインジェクションの針は1~2マイクロメーターという細さです。注射する対象物の大きさは移植者が常に意識していることで、ESを混ぜられて分からなかったなどということはありません。笹井さんもそうおっしゃっておられたとおりです。清成さんにES細胞が混ぜられていても分からなかったと思いますかと聞いたら彼はとても困ったでしょうね。記者は誰も聞かなかった。左上の写真はまさに若山さんが小保方さんから渡された細胞をナイフカットしてインジェクトしている当の写真です。細胞塊の中にES細胞が見えますか? 胚盤胞の一番左奥にあるのはリシピエントのインナーセルマスです。ほぼES細胞と同じ大きさですが、そんなものが混じっていて若山さんが気づかないなんてことはありません。この実験は264個の胚盤胞を使って行われているんですよ。清成さんが再現実験で行ったインジェクション操作も244回です。若山さんは64匹のキメラマウスを得たが、清成さんは生まれたマウスの一匹もGFP確認できなかった。
若山さんの作ったキメラマウスは小保方さんの酸浴細胞をナイフカットして移植したキメラ胚から作られたものではありません。彼が小保方酸浴細胞の核を移植したクローン胚から作ったntES由来のキメラなんです。その意味で小保方さんは無実です。キメラは実際にできています。なぜできたのかを説明できなければなりません。小保方さんが出所不明の太田ESと学生にもらったGOF ESとFLSのコントロールとして若山さんから貰っていた「僕のマウス」ESを使って、若山さんを騙してキメラマウスを作らせたというのが桂報告書とBCA報告書の結論です。
出来ているキメラ、幹細胞、テラトーマ=太田ES、GOF ES、「僕のマウス」ESこれが両報告書の結論です。この結論が間違っているのです。重大な間違いなのでこのまま放置してもらっては困る。従ってOoboeさんのパートナー氏が検察に調査依頼をされているのです。GLという名の幹細胞が2011/11/25に培養開始されて樹立されている。この細胞は調べられていませんが、できているのですから、両報告書の論理では学生のntESを混ぜて小保方さんが若山さんに渡したからできたということになる。後のGLSは学生のGOF ESを使ったと言ってるのですから、この時も学生のGOF ESを使っていないとおかしい。ところが2011/12/27に移植したテラトーマからはアクロシンCAGが出たので、両報告書は太田ESが使われていると結論した。テラトーマはGOFマウスがドナーになっている。小保方さんがESを使うなら学生のGOF ESを使うはずなのに、出てきたのは太田ESだという矛盾に両報告書は真摯に向き合わなければならない。
更に、AC129とFLS-Tシリーズの幹細胞から「僕のマウス」ESが出たことは、小保方さんの捏造ではないということが確認されてしまうと、今度は誰が何のために行ったのかという、新たに深刻な問題を発生させる。
①日本細胞生物学会から竹市所長に送られたメールの連名者達
②大隅理事長
③11Jigen
④世界展望
⑤東北大学黒田教授
⑥放医研の協力者
⑦京都大学太田氏
⑧遠藤氏
⑨吉村氏
⑩岡部氏
⑪日経サイエンス記者
⑫毎新聞社記者
⑬理研から小保方さんの実験ノートをNHKに全コピー違法流出させた者
⑭細胞リストを毎日新聞に違法流出させた者
⑮ノフラー氏
⑯李氏
⑰感想氏
⑱小保方バッシングを行い続けてきた書き込み者達(日経サイエンス記者の知り合いと自分で言ってるものもいる)
等々、たくさんの人々が調査対象者になるでしょうね。
最初に小保方さんの高校時代の個人的な悪意ある噂話を流布した週刊誌があったが、誰に頼まれたのかを調査すべきでしょうね。吉村さんはネットに流出していたと称しながらネイチャー査読文を更に拡散した犯人で、しかもTCR再構成の有り方に関して素人に向けてスピン解説をしている。加えて後に遠藤氏論文の中で謝辞の対象者になっている。若山・遠藤・吉村というラインがありますね。一方でNHKには若山さん自らが番組出演している。データを流したのは彼自身ですね。番組にはなぜか山梨大の若山研に大日向氏が出演してPCRの小芝居をしている。いつ山梨に来たのか。若山さんは共同著者なので仮に小保方さんが捏造したのだったら共著者間と理研内で話し合って解決に向かわせないといけない立場です。何をしているのでしょうかね。
一つには理研が大々的に記者会見をして世間に宣伝してしまったということがある。こんな大成功の後ですから引っ込みがつかないというようなこともあるかもしれませんが、そもそも若山さんはただ騙されただけなんでしょう。だったら理研に行って、理研上層部のいる前で、自分は小保方さんに騙されたんだと言えばいいことです。罪は無いではありませんか。そして小保方さんが認めたら、理研はあれは間違いだったと世間に謝ったら済むことです。ところが彼はそうしなかった。マスコミを巻き込みながら小保方さんに騙されたと騒いだのです。そして当の小保方さんは今に至るまで、自分はESを若山さんに渡したことは無いと主張しているままです。どうしてこんなことになったのか。若山さんが理研に行って、皆の居る前で堂々と小保方さんと対峙しなかったからではありませんか。最初の成功時に渡したマウスは何だったのか、「僕のマウス」ではなかったじゃないか。どうしてFLSの時の話から始めなければならないんだい。半分しかGFPが来なかったなんて話は不要でしょ。最初はGFPはヘテロじゃないか。皆の居る前で捏造したと言ってる相手と対峙すべきでしょ。ナイフカットで胚盤胞注入した時にどうしてわからなかったのと笹井さんに聞かれて困るようなことでもあるのか。なんでも本当のことを言って説明すればいいだけじゃないか。君は一体何をしているんだい。
理研も又どうして警察を入れなかったのか。公務員は犯罪を知ったら必ず警察に届けるということになってる。研究倫理の問題ではない。NHKに小保方さんの実験ノートの全コピーが流出した時点で既に犯罪です。公務員は誰でもまず警察に届けなければならない。理研はみなし公務員の義務を怠っている。マスコミ業も惨状のようですね。
お前たちは何をやってるんだということですよ。一般国民は飽きれてるぜ。自動車や工作機械や薬品や発電機やコンピューターを作っている製造業に従事している社員も、ダムやビルや住宅を作っている土建業の人々も、世界各地から物質を調達している商社マンたちも、金融機関に勤めている人々も、卸でも小売りでも流通産業に従事しているものや、JRやANAや宅急便の運輸業に従事している人々も、医者や看護師や弁護士や公認会計や、有りとあらゆる大人の日本国民はお前たちのユルフンに呆れている。無能だね。永遠にぬるま湯に浸かってせいぜいナガイキセイヤ。
(さて、戻って、なぜローザ)
若山研で使われたことのある129マウスは太田氏が論文で使った129/Sv-ter、そして129/Sv-X1、更にロックフェラー大学で作ったB6のGFPホモマウスからGFPだけを移し替えた129/Sv-X1GFPホモがある。
太田氏は129/Sv-terと岡部マウスとのF1を使って2005年の論文の実験を行った。論文の掲載経緯は以下です。
Biology of Reproduction, Volume 73, Issue 3, 1 September 2005, Pages 390–395, https://doi.org/10.1095/biolreprod.105.041673
Published:
1-Sep-05
Article history
Received:
8-Mar-05
Revision Received:
29-Mar-05
Accepted:
26-Apr-05問題になったFES1とFES2の凍結日は2005/12/7です。論文は3月に提出されていますから実験は当然もう終わっています。ntESG2の凍結日は2005/1/20です。番号の若いntESG1の凍結日は2007/8/3ですが、これは2008年の論文があって、ここで2005年時の凍結細胞を解凍してもう一度使っている。番号はこの時に振り直されているのではないかと推定される。2008年論文の掲載経緯は以下です。
Biology of Reproduction, Volume 79, Issue 3, 1 September 2008, Pages 486–492, https://doi.org/10.1095/biolreprod.107.067116
Published:
1-Sep-08
Article history
Received:
12-Dec-07
Revision Received:
9-Jan-08
Accepted:
25-Apr-08この太田氏の二つの論文で作られたntESは129/Sv-ter x 岡部B6なんです。論文にちゃんと書いてあります。岡部B6というのは無論問題のアクロシンGFPの共挿入マウスですね。
ところが、若山氏が京都大学の太田氏から入手して理研にも最終的に提出されたはずのサンプルは論文と同じくラベルも129/Sv-ter x 岡部B6だっんですが、なんと調査した結果は親の雌雄が逆になっていた。つまり岡部B6 x 129/Sv-ter になっていた。
加えて驚くべきことに桂調査チームはこの事実をスルーしたのです。提出されたサンプルの中身が論文とラベルと異なっているにも関わらず、中身の入れ替えを問題にすることなく、同時提出されているFES1とFES2を証拠採用しているのです。
>>
他方、同じ Acr-GFP/CAG-GFP の挿入を持つ ES 細胞 ntESG1、および ntESG2 の X 染色体は C57BL/6 であることが判明したことから、調 査対象の STAP 幹細胞 FLS3、FI 幹細胞 CTS1 と性染色体の構成が異なるため、これ らは比較解析の対照から除外された。 (5P)頭大丈夫か?ユルフンかい?他方、ntESG2は実験終了後早々の2005/1/20には凍結されていて、その後論文の提出作業に入る。FES1とFES2は約1年後の2005/12/7の凍結です。何の目的もなく作ったと太田さんは日経サイエンスに証言している。実験には使っていないから作ってすぐに凍結しているはずです。ところがこの129はterではなく、X1なんです。岡部マウスとのF1の受精卵ESなんです。今まで使っていて飼育しているF1ではなく、又一から交配し直して受精卵ESを作ったという。ntESではありません。何のためにこんなものをつくったのか。本人は何の目的もなく作ったと言っていて、しかも日経サイエンスにはterで作ったと記憶していると語っている。そちらの方が普通でしょう。X1で作るときは交配からやりなおさないといけない。
>>
太田氏によると、このES細胞2株は、核移植ES細胞を作ったのと同じころ、たまたま作ったES細胞だという。結局研究に使うこともなく、2010年3月に他大学に転出する際にすべて持って出たと思っていた。
太田氏の記憶では母マウスはクレア社の茶色い129+Terマウスだったが、実際には129X1の白マウスだった。(53P)GLSの遺伝子異常の件から我々は既に若山さんのサンプルの入れ替えを立証しています。このFESでもサンプルは入れ替えられているのです。薄茶のマウスと白毛のマウスの違いを覚えてないいうことはあり得ないことです。受精卵ESを作ったことは覚えているので、論文の実験では薄茶を使っていたことは明確で、その後新たに白毛を交配させて受精卵ESを作ったのなら覚えていますよ。その記憶がないというのは以前の実験で使っていた維持飼育していたF1を使ったということです。若山さんはそれを取り寄せて、中身をFLSに取り換えた。
BCA報告のSNPs解析結果です。FES1の中身は入れ替えられていてFLS3ですから一致は少しも不思議でない。129GFP ESはそもそも小保方さんが使っていたFLSですからこれも当たり前の結果です。ただし、このサンプルに関して小保方さんも含めて全員が知らないと言ったという証言が確認されていますが、小保方さんに対してどういう聞き方をしたかですね。後にサンプルの中身に関して答えているところでは129GFP ESはFLSのことになります。それを知らないと答えたのはいつの時点での話なのか、又どういう聞き方をしたのかによる。小保方さんは自分がどういう目に遭っているのかを知らないのです。そしてラボに迷惑もかけられないと考えている。しかも桂調査チームもまさかntES化実験だった可能性なんかには気づいてもいないし、若山さんの嘘の動機にも気づいていない。
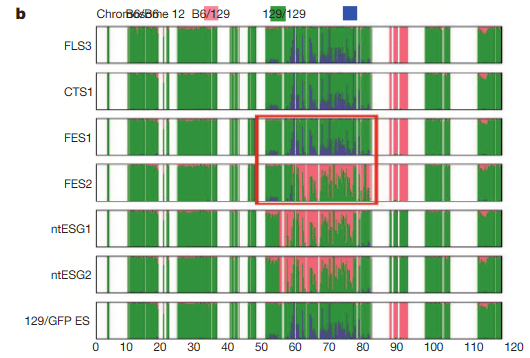
ntESG1とG2はラベルと中身が違っているものです。そのntESとFES2の一部のSNPs分布が重なっている部分がある。中身の入れ替え時にきれいに洗いだされていないのではないかとも疑われるところです。和モガさんが既に指摘していますね。ただ、この場合は両方が混じるので又違うパターンになるのではないかとも疑義されます。
いずれにせよ、FES1も2もX1ですから太田さんの作ったものではない。またntESG1もG2も太田さんの二本の論文の細胞とは違います。親の雌雄が違っている。むしろラベルの記載が論文通りなのが中身の入れ替えを証していると疑義される。
BCA報告書にはあからさまな間違いが書いてあります。
>>
These STAP cell lines were then compared with four ES cell lines—FES1, FES2, and
two nuclear transfer ES lines (ntESG1 and ntESG2) (ref. 9)—established from crossing the Acr/cag-GFP mouse strain with 129 mice in the Wakayama laboratory in 2005 (Extended Data Fig. 1a and Extended Data Table 1).
(ref. 9)というのは以下ですね。
>>
9. Ohta, H. & Wakayama,T.
Generation of normal progeny by intracytoplasmic sperm injection following grafting of testicular tissue from cloned mice that died postnatally. Biol. Reprod. 73, 390–395 (2005).
この論文で使われているF1はB6N SLC♀/ 129+Ter CLEA♂ではありません。親の雌雄が逆でチューブのラベルに書いてある通りの、ntESG1:129B6F1G1、ntESG2: 129B6F1G2 です。BCA報告は論文もちゃんと読んでない。しかも、ラベルと中身が違ってるのにそれをスルーした。こんな解析データをつけて何の意味があるのでしょうかね。中身は太田氏の細胞ではない、にも関わらず太田氏の細胞だと言ってる。どうしたのだ。アンポンタンなのか。ユルフンなのか?この論文には明確にメスが129だと書いてありますが、今現在コピペできないようにされていますね。以前は山梨大の若山研文献にもありましたが今外されている。このこと自体が若山さん犯人を証明していると言ってもいいでしょう。論文はOoboeさんとパートナー氏、そして相沢さんも持っている。無論、私もです。
日本語訳なら大丈夫かな。
>>
マウス
未成熟雄B6C3F1、成体雌ICRおよび成体雌BDF1マウスは、静岡研究所動物センター(Hamamatsu、Japan)から購入した。メス129 / Sv-terおよびオスICRヌードマウスは、CLEA Japan、Inc.およびCharles River Japan、Inc.からそれぞれ購入した。アクロシン/ eGFP(Acr3-EGFP)[34]およびpCX-eGFP [35]トランスジーン(C57BL / 6TgN(acro / act-EGFP)OsbC3-N01-FJ002 [36])の両方を有する緑色蛍光タンパク質(GFP)トランスジェニックマウスは、 )は、大阪大学(Osaka、Osaka、Japan;すべての菌株の名称は元の研究の名称である)のOkabe博士によって親切に提供された。 GFP導入遺伝子を有する129B6F1マウスを作製するために、メス129 / Sv-terマウスをC57BL / 6 GFPトランスジェニックマウスの雄と交配させ、核移植のためのドナーとしてこれらの交配の子孫を使用した。すべての動物実験は実験動物のケアと使用のガイドラインに準拠しており、理研神戸研究所の実験動物実験機関委員会の承認を受けています。はは。
(驚くべき虚偽)以下は桂報告書のスライド版にあるSNPs解析表です。
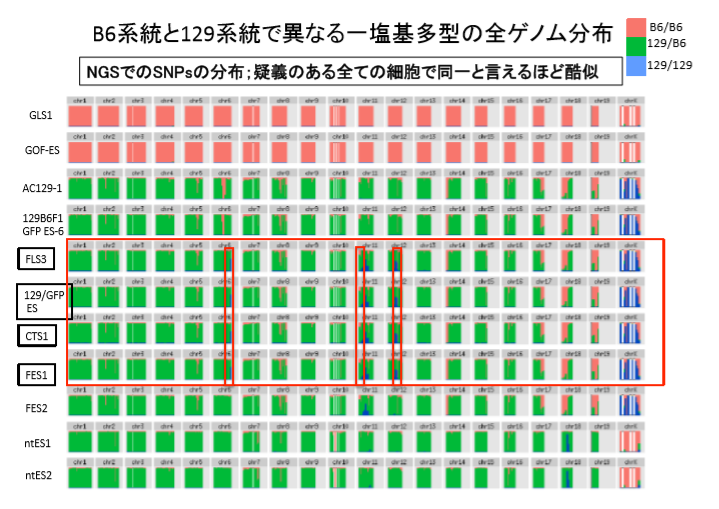
こちらはBCA報告に添付されているSNPs解析表です。
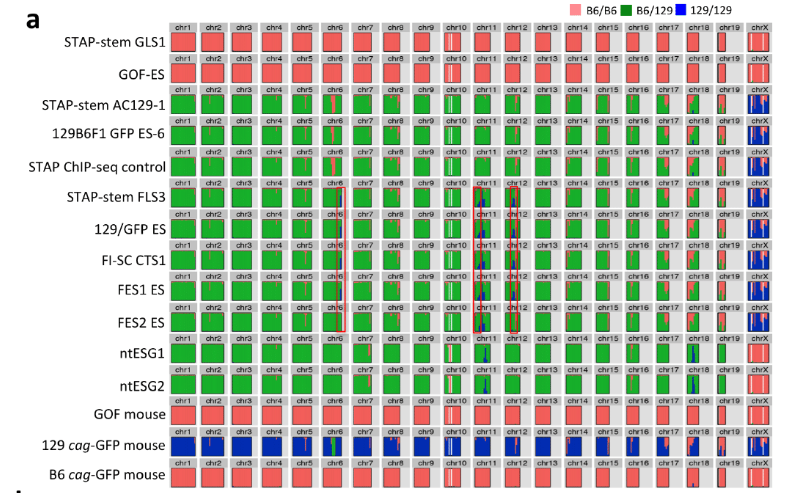
どこが違うか確認されてください。
上部のGOFマウス関係は別として一番下に129 cag-GFP mouseとcag-GFP mouseが置かれている。これは言うまでもありませんが「僕のマウス」の両親です。AC129を巡る問題1で説明したとおり、若山さんはロックフェラー大学でB6のCAGホモマウスを作った。後の調査で18番染色体にGFPが入っていることが分かっています。その後彼は理研に移籍してからこのB6cagホモマウスのGFPをそのまま129/Sv-X1に移し替える実験を長期にわたって行った。一度B6GFPホモマウスと市販の129を交配させてF1を作る。F1ですから今度はGFPがヘテロに入っている。このF1に市販の129をかけ戻す。戻し交配という。この時常にGFPの光っている子供に対してのみ戻し交配を続ける。これを20継代続けるとGFPのみが最初のB6の遺伝子座に残る129の近交系マウスに戻る。その後にヘテロ同士の兄妹交配からホモの子供を作るんです。何度掛け合わせてもすべての子供からGFPが出るとホモになっていることが確認できる。20継代は大体4年位かかる。これでGFP以外は完全に近交系マウスになる。しかし、直前のグラフを見ると129は近交系になってませんよね。
近交系になってないという言い方は誤解が有るかもしれません。129特異的SNPsというのが分かっていて、こういう分析をすると全部ブルーになるマウスがあるというのが前提です。ピンクが混じっていてはおかしいわけです。上の表の若山さんの129はピンクが混じっています。でもこのままで20継代続けたら近交系マウスになります。ただし129/Svマウスとは言えないということです。これは近交系マウスになっていたとしても若山さんマウスということです。ただ、この1件だけでは近交系マウスとして固定しているかどうかは分からない。たくさん抜き出して全部同じパターンになっていたら若山129マウスとして近交系になっているということですが、どうも他の交配結果からして、近交系にもなりきっていませんね。遺伝子配列にヘテロのままになってるいろんなマウスか混じっているようです。つまりマウスコンタミして20継代まで経過していないということです。ここに例示されているパターンのものだけでないということですね。他のパターンのマウスが混じっている。だからこそ129B6129BF1ESも1と6の二種のSNPsパターンに分かれたということです。たまたま使われたマウスが2種だけで実際にはもっと違ったパターンのマウスも入っている可能性がある。
BCA報告は129の全部ブルーの市販のマウスのSNPsパターンを示していません。なぜでしょうね。純粋に青とピンクが半々になったらグリーンで示されることになる。ここにピンクが入っているということは全部ブルーであるはずの129側にピンクが入っているということです。若山さんの129のブルーの中にピンクが入っているパターンがそのまま他の細胞のグリーンの中にピンクで残っているものを表にしてみましょう。
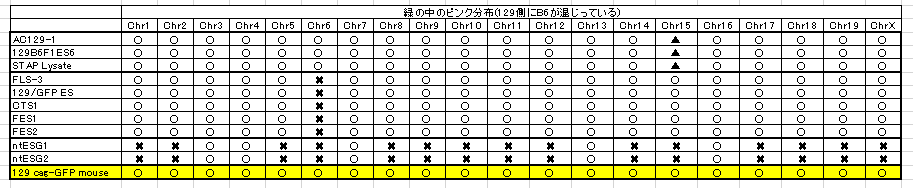
〇は若山マウスと同じB6混入があるという意味です。✖は若山さんのB6混入パターンと違うという意味で、▲は又✖とも違うパターンという意味です。〇という表示はそれが正しいパターンだという意味に取られやすいのですが、若山さんの129は全然正しくないものです。本当は正しいブルーだけのパターンのものをコントロールとしてつけるべきでしょうが、既述しているようにそれがつけられていませんから、代わりにntESG1G2を基準にしてそれを〇表示してみましょう。ntESG1G2は太田さんの論文とチューブラベル表示のものではなくて中身は親の雌雄の違う別の細胞です。でも使われている129+terは市販のもので、若山129マウスではありませんね。報告書が確認しています。こちらは緑の中にほとんどピンクが無いということを確認してください。129+terにはB6のコンタミがほとんどないんです。少しだけあるのはもともとのジャクソン研究所でのコンタミではないかと疑われる。というのもこちらにあるピンクはX1にもありますからね。共通している。以下にntESを〇にした表を示します。
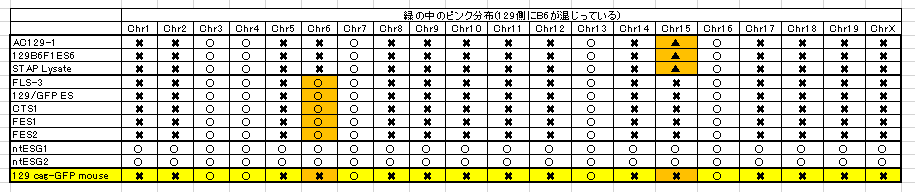
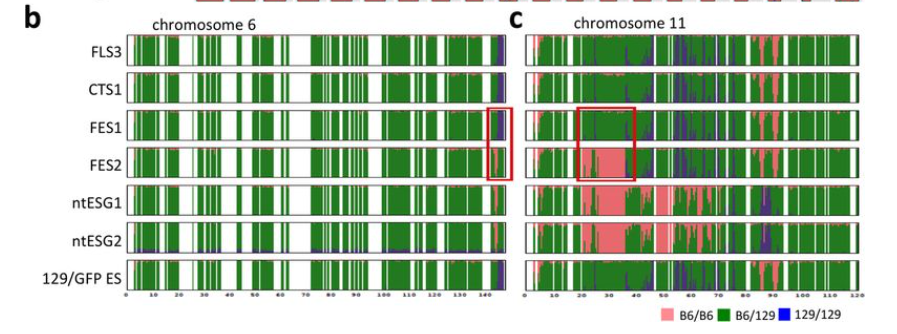
6番と15番染色体以外の染色体に関するB6のコンタミパターンは全部若山129マウスと同じだということに気づけます。市販の129X1/Svにはこういうコンタミは無いはずですね。むしろ129+ter/Svと同じはずです。というのも129X1/Svと129+ter/SvのSNPsパターンがそんなに違っているのなら、こういう比較そのものが意味をなさなくなる。報告書は129とB6のSNPsパターンを比較しているだけです。129の中の亜種で違うSNPsなんて使うわけがありませんね。
上の表は他のF1細胞の129がすべて若山マウスであった可能性を暗示しています。つまり記者会見の情報から推測するに18番に1コピーのCAGが入っているということです。このことは放医研が最初GFP位置を間違えたことに端を発してうやむやになったままで調べられていませんね。
6番と15番のコンタミパターンが違っているというのは若山129マウスの中が不均一な遺伝子構成を持ったマウスでコロニー形成されていた。つまり近交系マウス化できていなかったという原因に求められるのではないでしょうか。
問題はGFPです。若山129マウスはCAG-GFPホモです。F1マウスの3番にヘテロでAcr-CAGが、18番にヘテロでCAGが入っていなければならない。
この問題は一度マクズハラさんのブログでアルイミオウジ氏の指摘として論じられている。
radiation-japan.info/journal/science/stem_sell/stap-cell-case.html

彼もその原因は分かってないから警察に呼び掛けているわけです。
では129のGFPの問題に入る前に、先にB6の方を確認して置きましょう。GOFマウスとGOFESはいいですね。ほぼ全部ピンクです。若山さんがロックフェラー大学で作ったCAGホモのB6マウスもほぼ全部ピンクです。129は混じってませんね。18番にわずかに有りますけどね。ちょっと原因が分かりません。ただもっと大きな問題が6番、11番、12番、そしてこの18番にありますからね。
ここには岡部マウスのSNPsパターンがつけられていない。
①岡部マウスは大阪大学の岡部さんのところにある。
②そしてその後太田さんが大阪大学から岡部マウスを理研若山研に持ち込んだ。そしてすぐに2005年の論文でそのマウスを使いました。そのサンプルは残っていますが、報告書の分析では使われていない。
③2005年の実験1年弱後に何の目的も無く岡部マウスとの受精卵ESが太田さんによって作られた。
④報告書が分析したのは論文とラベルの記載とは異なった親の雌雄の違ったサンプルの中身でした。でも使われているB6は岡部マウスだと分かっている。この時期の岡部マウスが一つあるということです。
管理場所と時期の違った四種類の岡部マウスがあるわけです。129とB6のF1であるからには全てのSNPsパターンは緑が原則です。そこにブルーが入っているということはB6側に129が入っているということです。GOFのマウスとロックフェラー大のCAGホモB6はほぼ全部ピンクでした。緑の中にブルーがはっきり出ている細胞のB6は岡部マウスなんです。これをどうやって調べるか。
まずntESG1,G2の129+terにはB6のコンタミがほとんどありませんでしたね。ほぼ全部ブルーです。ここにその129+terと岡部マウスが掛け合わされた結果がある。④の結果ですね。緑の中にブルーのある部分は④の岡部マウスB6にコンタミしている129のSNPsなんです。なぜなら、129+terが全部ブルーだと分かっているからです。なぜ全部ブルーだと分かったかというと、ntESG1,G2は全部グリーンになっていてB6の混入が無いからです。でもブルーの混入はある。これが④の岡部マウスに混入した129のSNPsなんです。それを基準にしてみましょう。
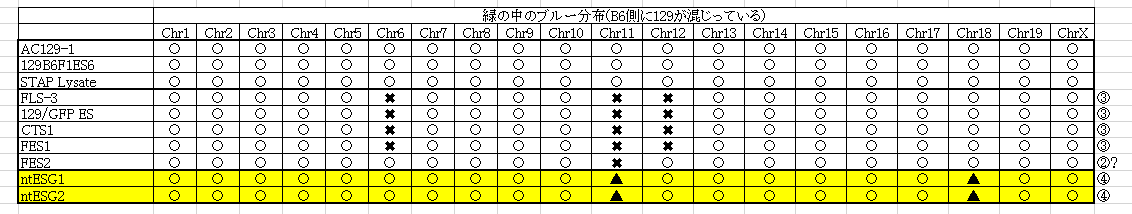
黄色の場所の▲が岡部B6マウスを若山研で自家飼育している過程で入り込んだ129です。AC129-1、129B6F1ES6、STAP Lysateの三つはいいですね。相手のB6はロックフェラー大で作った若山さんのCAGホモマウスで、解析をうけていますね。129のコンタミはありません。問題になっているのはあくまでも岡部マウスなんです。再掲しましょう。
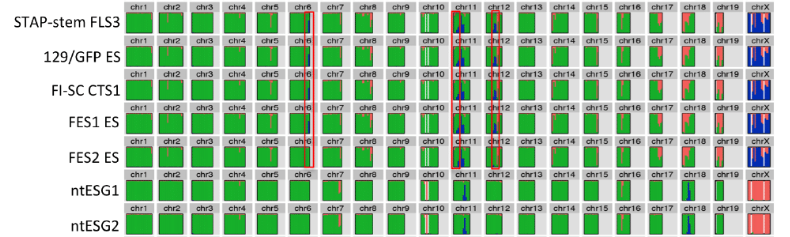
まずntESG1,G2のコンタミ場所は他のいずれとも共通していないということを確認しましょう。この岡部マウスが一つあるということです。④の分ですね。太田さんの論文のものではありませんよ。どこかから出てきたものです。今、Ooboe さんのパートナー氏が検察に調査依頼されています。
次にFLS-3、129/GFP ES、CTS1、FES1の4つの細胞株作成時に使われた岡部マウスがある。この4つは同じ場所に同じパターンのコンタミがあって、同じ岡部マウスからの作製のようですね。そして上記ntESG1,G2とは全く違う場所にあるということを確認しましょう。
そして、最後にFES2に使われた岡部マウスがあって、これは6番、12番にはなくて、11番のみFES1と共通している。時期的にはFES2の岡部マウスが使われたあと更に6番、12番のコンタミが加わっているので、作られた順番はFES2が先で、その後にFES1が作られていることになる。そしてntESG1,G2の岡部マウスは飼育コロニーの違うものとしか考えられませんね。
129も岡部マウスのB6も自家飼育している間にマウスコンタミが発生している。これが遺伝子調査結果にどう反映しているのかはとてもむつかしい問題です。逆にマウスコンタミが無ければおよそ遺伝子解析なんてできなかったでしょうね。でもあったからいろいろと問題が分かったが、そのありようがどうであったか。とても錯綜しています。
そもそも小保方多能性細胞の探求にとって若山研のマウス管理は適合的ではありませんでしたね。Oct4遺伝子を発見するのにGFPを使って大丈夫だなんて酸浴させて死にそうになってる細胞で適合的な実験方法でしょうかね。ジャームライントランスミッション実験でGFPを確認するのにそれだけマウスコンタミのあったコロニーの中で交配するなんて適合的ではないでしょう。でも、それは若山研の責任ではありませんね。若山さんのクローン胚の探求にとって、マウスコンタミは大した問題ではなかったはずですよね。こういうことになったのは偶然に過ぎない。バカンティ氏側がキメラを作ってくれと頼んできた流れです。小保方さんの細胞研究をするためのラボではありません。でもこのラボでOct4-GFPが大量に光った。誰が悪いのでしょうかね。
リクルートの話とはこれは別なんですね。多分科学的にはこちらが問題だった。
(そもそもAC129の問題とは何なのか)AC129は129-CAG-GFPマウスから作られたと言われているのにこの幹細胞からは「僕のマウス」ESが出てきた。これは中身の入れ替えで説明できる。でもSTAP lysateはどうなのか。これも中身は「僕のマウス」ESだった。桂報告書はサンプルを調整してGRASに提出したのは小保方さんだったと主張している。でも一つのことを忘れている。若山さんはリトラクション理由に何と書いているか。
>>
(4) In Fig. 4b of the Letter, STAP cell and ES cell are wrongly labelled in a reverse manner.
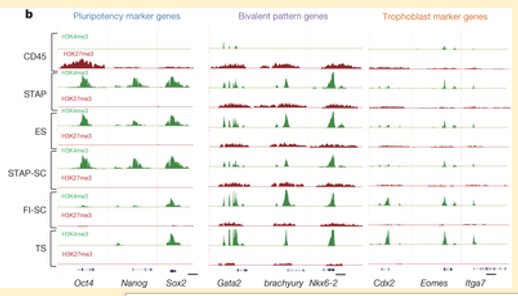 これが本当ならStap Lysateと書かれているサンプルはESなんですね。こちらを分析して「僕のマウス」ESが出るのは当たり前です。まさかそんなことしてないですよね。
これが本当ならStap Lysateと書かれているサンプルはESなんですね。こちらを分析して「僕のマウス」ESが出るのは当たり前です。まさかそんなことしてないですよね。
大丈夫か?
>>
3)STAP 細胞由来 ChIP-seq (input)サンプルは 129B6 F1ES1 から取得された
(調査結果) 小保方氏が CDB ゲノム資源解析ユニット(以下「GRAS」という)に持参し残されてい た STAP 細胞由来 ChIP-seq (input)サンプルを再度 NGS 解析した結果、STAP 細胞由来と される ChIP-seq input データは CAG-GFP の挿入を持つ 129xB6 へテロ系統由来の細胞か ら取得されたものと判明した。さらに SNPs の解析、特異的な欠失変異の解析により(2 -3-1-1(d)参照のこと)CAG-GFP が挿入された 129B6 F1ES1 とほぼ同一細胞由来の データであることが明らかとなった。 ES Lysateの方は調べたのか? リトラクション日付は (03 July 2014)よ。両方調べて初めて若山さんのリトラクション理由が本当か嘘かが分かる。又両方の結果を記載して初めて報告書がどちらのサンプルが「僕のマウス」ESだったとしているのかが分かる。このままだと、どちらが「僕のマウス」ESだったのかが分からない。その可能性として4つある。
①STAP LysateとラベルされているデータとES Lysateとラベルされているデータのどちらも「僕のマウス」ESだった。
②STAP Lysateとラベルされているデータだけを調べてそれが「僕のマウス」ESだった。
③リトラクション理由書に取り違えがあると書かれていたのでES Lysateと記載されている方がSTAP Lysateだと見做してそれのみを分析したら「僕のマウス」ESだった。
④そもそもSTAP Lysateとラベルされているデータしかなかった。
①ならそう書くことによって129/Sv-CAG-GFPのデータは無いので Fig.ure 4-b は捏造であると報告しなければならない。
②の場合はリトラクション理由書に書かれている通りES Lysateが129/Sv-CAG-GFPである可能性があるので、それを調べないままにSTAP Lysateが「僕のマウス」ESだと書いた桂報告書自体が捏造だとされなければならない。
③の場合は、ではSTAP Lysate を調べてないことによってリトラクション理由書が虚偽記載されているということを確認しないまま報告書を書いたということになり、これも捏造報告書だということになる。
④の場合はリトラクション理由書に取り違えがあると書かれていることに気づいていない杜撰な報告書だということになる。
調査報告に瑕疵があるということは明白ですね。取り下げてもらうか、訂正してもらわないといけない。我々は若山さんが犯人だと考えているので、リトラクション理由には強い疑義を抱いている。どうしてあんなに似ている Figure 4-b の取り違えに気づけるのか。手伝った人が居るのではないかと。また、取り違えが本当なら、リトラクション理由書はむしろ桂調査の結果を否定することになるのはどうしてかと。
リトラクション理由書が先に提出されている。その後にGRASに残されていたデータが再解析された。若山さんはデータが残されていたことは予期してなかったと思われる。しかし、STAP Lysateを記載通りに調べたら、中身がESであったということになると、取り違えは本当であったということになる。ところが、ではどうして調査チームはES表記されている方を調べなかったのか。そもそも我々は試料が残されていたということ自体を疑うもので、調査チームの中に犯罪者が居るのではないかと疑義している。資料が残されていたということは記者会見の席上で伊藤氏が証言している。その時の彼の表情については既述しているとおりである。
AC129を巡る問題1で述べたことを再掲しましょう。
**********
>>
(4) In Fig. 4b of the Letter, STAP cell and ES cell are wrongly labelled in a reverse manner.
Letter Figure 4-bは以下です。よくこの程度の粗雑な簡略図でSTAPとESの取り違えに気づけたものです。誰かラボ内に手伝った人でもいて、元データを持っていた人がいたことが推察されます。

**********
2年前の生データがSTAP細胞だけ冷凍されて残っているというのも変で、残っているならこの時の試料の全てのはずですが、記者会見では伊藤氏が口頭で記者の質問に答えて、この時の分析の試料がGRASにたまたま残っていたのだと証言しただけで、他の試料の有無に関しての更なる追及も無かった。又その保管に関して文書で追加説明があったということもない。伊藤さんはBCA報告の著者の一人です。
>>
Daijiro Konno, Takeya Kasukawa, Kosuke Hashimoto, Takehiko Itoh, Taeko Suetsugu, Ikuo Miura, Shigeharu Wakana, Piero Carninci & Fumio Matsuzaki
桂会見での古田女史の質問に対する答えは以下でした。言い終わった後の彼のしぐさと表情は精神分析学の研究対象に好例かもしれない。(*ttps://www.youtube.com/watch?v=M9oJGioHvIQ 1:15:00から)
>>
ただ、唯一やったのが、あ、GRASの方に残っていたChIP seq のinput 、しかもSTAP細胞のインプットはDNAとして残っていたので、それが10ページバワーポイントの、えーと、ところに書いてある、3番のアイテマイズして書いてあるところですけれども、これに関しては、えーと、30倍になるだけ、他のゲノムと同量となるまで読んでいます。そして、読んだ結果、この、同一であるということを、ま、認定したということで、それ以外に、それ以外に関しては、ま、残っていませんのでやってない、やってません。
なぜ、たくさんある中の、唯一、STAP ChIP lysateだけが残されていたのか、どういう形で唯一残されていたのか、誰かの恣意で残されていたのか、それともGRASには残し方に関する規則は無いのか。裁判だったらまず最初に検討されるもので、ここにちゃんとした答えが無いと証拠採用はされない。
**********
①唯一やったのが
②STAP細胞のインプットはDNAとして残っていた
③読んだ結果、この、同一であるということを、ま、認定したということ
④それ以外に、それ以外に関しては、ま、残っていませんのでやってない、やってません。
つまり、STAP Lysateと書いてあるDNAのインプット試料だけがあったということです。そしてこの記載事項は2014/7/3に提出さけているリトラクション理由書の記載が本当なら、小保方さんが論文の図表として採用するときに間違えたか、そもそもGRASに提出するときに既に記載を間違えているかのどちらかです。ただ、リトラクション理由書は試料が残っていたということを知らなかった可能性が高い。若山さんは何をもって取り違えに気づいたのか。あのレター論文の図だけを見て取り違えに気づけるというのは難しいのではないか。何か、別の根拠があるはずです。
図だけを見て気づけるのはNanogの発現量です。ただこれがどちらがどうといえるためには元の解析データを若山さんが持っているのでなければならない。この場合は元のデータには提出時の記載事項通りに結果が知らされていることになる。若山さんは小保方さんの図表とGRASの結果を比較してNanogの発現量から図表は結果を取り違えていると主張したことになる。この場合、GRASのSTAP Lysateと書かれているデータが図表のESだということになる。Nanogの発現量の小さい方です。これが「僕のマウス」ESだったということになる。
もう一つの可能性はそもそも小保方さん自身がSTAP細胞を作ったのちにGRASにES細胞と一緒に提出するときに記載を間違えた場合です。この場合はGRASは記載されたとおりにデータを渡すので、そもそものデータが逆になっていることになるが、若山さんが図表とGRASの間での取り違えに気づくことも無いでしょう。
明確なのは小保方さんがSTAP細胞と記載してGRASに提出した細胞は「僕のマウス」ESだということです。少なくともSTAP細胞は小保方さんが作るので、これが「僕のマウス」ESであるためには提出記載の取り違えか、或は、渡されたマウスが「僕のマウス」だったかしかありません。取り違えはES記載された試料が残ってないので確認できない。渡されたマウスが「僕のマウス」だったのなら129/Sv-CAG-GFPマウスの実験なんて無かったということになる。
我々は若山さんが嘘をついていることを知っている。AC129の問題は彼がここで何をしたかを解明することです。
取り敢えず、 Fig.ure 4-bに関して登録されているデータを書きに提示しましょう。
ChIP-Seq
①SRR1171553 小保方 CD45 positive Cells:ChIPSeq.H3K27me3 derived from spleen Oct3/4::gfp C57BL/6 GOF
②SRR1171554 小保方 CD45 positive Cells:ChIPSeq.H3K4me3 derived from spleen Oct3/4::gfp C57BL/6 GOF
③SRR1171555 小保方 CD45 positive Cells:ChIPSeq.input derived from spleen Oct3/4::gfp C57BL/6 GOF
④SRR1171562 若山 Embryonic Stem Cells:ChIPSeq.H3K27me3 C57BL/6x129/Sv
⑤SRR1171563 若山 Embryonic Stem Cells:ChIPSeq.H3K4me3 C57BL/6x129/Sv
⑥SRR1171564 若山 Embryonic Stem Cells:ChIPSeq.input C57BL/6x129/Sv
⑦SRR1171567 若山 FI-SC(Fgf Induced Stem Cells):ChIPSeq.H3K27me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-Acr-CAG
⑧SRR1171568 若山 FI-SC(Fgf Induced Stem Cells):ChIPSeq.H3K4me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-Acr-CAG
⑨SRR1171569 若山 FI-SC(Fgf Induced Stem Cells):ChIPSeq.input Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-Acr-CAG
⑩SRR1171582 小保方 Low pH treated CD45 positive Cells:ChIPSeq.H3K27me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑪SRR1171583 小保方 Low pH treated CD45 positive Cells:ChIPSeq.H3K4me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑫SRR1171584 小保方 Low pH treated CD45 positive Cells:ChIPSeq.input Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑬SRR1171587 若山 STAP-SC(STAP derived Stem Cells):ChIPSeq.H3K27me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑭SRR1171588 若山 STAP-SC(STAP derived Stem Cells):ChIPSeq.H3K4me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑮SRR1171589 若山 STAP-SC(STAP derived Stem Cells):ChIPSeq.input Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑯SRR1171592 丹羽研 Trophoblast Stem Cells:ChIPSeq.H3K27me3 CD1
⑰SRR1171593 丹羽研 Trophoblast Stem Cells:ChIPSeq.H3K4me3 CD1
⑱SRR1171594 丹羽研 Trophoblast Stem Cells:ChIPSeq.input CD1
伊藤さんが試料そのものが残っていたと言っているのは⑫のことです。生試料があったと言ってる。その他は残って無かったと。
>>
ただ、唯一やったのが、あ、GRASの方に残っていたChIP seq のinput 、しかもSTAP細胞のインプットはDNAとして残っていたので、それが10ページバワーポイントの、えーと、ところに書いてある、3番のアイテマイズして書いてあるところですけれども、これに関しては、えーと、30倍になるだけ、他のゲノムと同量となるまで読んでいます。そして、読んだ結果、この、同一であるということを、ま、認定したということで、それ以外に、それ以外に関しては、ま、残っていませんのでやってない、やってません。
本当に残ってなかったかな。私の直感は以下のようなものでしたけどね。今でも見れますね。
>>
桂会見での古田女史の質問に対する答えは以下でした。言い終わった後の彼のしぐさと表情は精神分析学の研究対象に好例かもしれない。(*ttps://www.youtube.com/watch?v=M9oJGioHvIQ 1:15:00から)
若山さんがリトラクション理由に挙げているのは⑤と⑪が逆になっているということです。そしてレターの図を見て誰にでも違いの気づけるところはNanogのグラフですが、これはTs.Marker さんが解析図を作ってくれています。
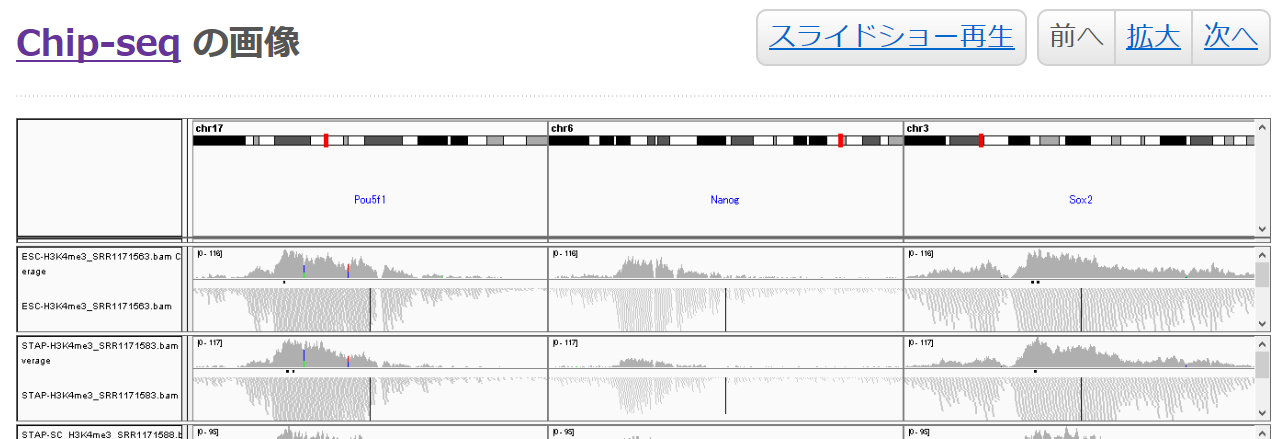
上が⑤で下が⑪です。これを見ると確かに小保方さんはグラフを間違えていますね。Nanogの多く発現している方がESです。これは論文のグラフを作ったときの間違いです。そしてリトラクション理由書の指摘事実は正しいということです。でもこれを以てリトラクトする必要はありませんよね。間違いは訂正したらいいだけです。共同著者の若山さんの行為はそういうものではない。論文そのものを全否定しているんです。自分で行った実験であるにも関わらず。他の共著者には説明も無い。話し合いもせずに一方的にリトラクトだと一人で騒いだ。何をしたのだということですね。そして小保方さんに騙されてES細胞でキメラを作らされてしまったのだというストーリーをマスコミを通して拡散した。そんなことマスコミを通すことか。共著者間で批判しあうべきことでしょう。共著者間の話し合いのテーブルにつけない理由があるわけです。我々は既にその理由を知ってるということです。
でも、今我々が解明しようとしているのはそのことではない。以下のように公共データ登録されている細胞⑪が「僕のマウス」ESであったという事実です。
>>
SRR1171583 Low pH treated CD45 positive Cells:ChIPSeq.H3K4me3 SAMN02393447 SRS559100 C57BL/6x129/Sv Oct3/4 expressing cells 2013/11/5 H.Obokata PRJNA238286 RIKEN 2014/2/13
小保方さんは2013/11/5に理研にデータを提出していて、理研はそれを2014/2/13に NCBI に登録したんです。その時の事務の担当者が誰かは分かっていません。
ここで小保方さんはこの細胞のマウス背景をC57BL/6x129/Svと書いています。まず最初に気づかなければならないことはこの細胞はAC129を作成したSTAP細胞ではないということです。雌雄の書き分けは小保方さんの間違いなのか事務担当者の間違いなのかは分かりませんが、AC129の実験で使われたマウスは「僕のマウス」の片親である129X1/Sv-CAG-GFPホモマウスだと報告書に書かれている。でも小保方さんが登録したのはF1マウスのSTAP細胞です。小保方さんはこのマウスをAC129作成時のSTAP細胞だなんて書いていませんよね。それどころかF1のSTAP細胞だと書いている。
そして検査の結果事実F1のマウス背景結果が出た。小保方さんがF1と書いているものからF1の結論が出て何がおかしいのだ。
報告書はAC129の背景を調べたらF1だったと分かったが、このAC129は129マウスで行ったものなのだからここからF1結果が出てきたらおかしいと言ってるわけです。AC129を作ったのは若山さんでしょ。自分で白毛の129/Svを小保方さんに渡してそのSTAP細胞からAC129を作ったんでしょ。そしたら中身がF1だったから小保方さんが自分にESを渡したのだと騒いでいるわけですけど、自分で中身をESに詰め替えたんでしょ。そうでないという証明が何もないじゃないか。何を一方的な嘘をついているんだよ。
それに関しては桂報告書も以下のような嘘を書いている。
>>
STAP 幹細胞 AC129 とされる細胞は 2012 年 8 月 13 日に作製されていることから、この 細胞はこれ以降に実施された実験に用いられたと判断した。公開データ再解析の結果に よれば、論文に記載された実験の中では Letter Fig.4 に使われた可能性が高く、また Letter 論文 Fig. 2i にも使われた可能性がある。しかし実験記録の不備から使用実験を 特定するには至らなかった。なお、Article のメソッドに、129/Sv carrying Rosa26-gfp からキメラ寄与能を有する STAP 幹細胞が樹立された、との記述があるが、129/Sv carrying Rosa26-gfp マウスは理研 CDB に導入された記録や飼育記録はないことから、 これは誤記と考えられ、若山氏の説明によればここで言及された STAP 幹細胞は AC129 であった可能性が高い。 「論文に記載された実験の中では Letter Fig.4 に使われた可能性が高く」などという推測は小保方さんが提出してる記載事実を見たらあり得ないと分かるでしょうに。小保方さんはF1だと書いているではないか。
これが本当に2012年の8月に提出されているSTAP細胞だったら必ず若山さんはF1マウスを小保方さんに渡しています。STAP細胞は継代保存ができない。維持培養期間は出来てからせいぜい1週間程度です。昔の細胞を提出するなんてことはできません。ChIPSeqに出した日付はGRASに残っているでしょう。生試料まであったというのだから、分析日付くらい分かっているはずだ。この頃小保方さんはたくさんのSTAP細胞を作らされたと証言していることを伊藤さんが記者会見の席で説明しましたね。他の人の実験のために使われたと言ってると。この細胞はAC129の実験のものだけではないでしょうよ。F1でも作らされている。それを小保方さんは提出しているんです。若山さんは実験ノートをつけていませんよね。よくもまあ、小保方さんのことが言えたものです。伊藤さんは小保方さんがかき集めたSTAP細胞が129/Svだったとでも思っていたんですかね。その実験は一回で終わってるでしょうに。
AC129作成時のSTAP細胞が「 Letter Fig.4 に使われた可能性」なんて全くありません。
- 2019/10/31(木) 22:32:20|
- AC129
-
-
| コメント:3


















