さて、次は桂報告書の証拠吟味ですね。ここで一応今後の参考のために小保方さんの纏まった抗弁を書き出しておきましょうかね。"スペルム"は日本語に置き換えて読んでください。ここでは禁止ワードになっていますので書けません。
手記209Pからの小保方さんによるアリバイ自己検証
第二次調査委員会によって、STAP細胞から作製されたはずの「成功したキメラ」は既存のES細胞から作製されたものであったと報告された。すでにくわしく書いたように、STAP細胞からのキメラ実験は、若山先生が作製方法をSTAP細胞塊をバラバラにして注入する方法から、マイクロナイフで切って入れる方法に切り替えた時に初めて成功している。もし私がES細胞を渡していたのなら、細胞塊をバラバラにして、キメラマウスを作製していた当初からキメラマウスの作製に成功していたはずである。そうではなく、実験方法を切り替えた時にES細胞を渡していたとするなら、連日行われていたキメラマウス作製実験において、若山先生が実験方法を変えるタイミングを予期し、そのタイミングに合わせてES細胞を若山研の誰にも知られずに準備し、ES細胞研究の第一人者である若山先生にばれずに渡すことが、果たして可能であっただろうか。
また、若山先生がSTAP細胞から樹立したというSTAP幹細胞も、以前に若山研で作製された既存のES細胞であったと報告された。STAP細胞は増殖性が低く、それがSTAP細胞の特徴の一つであり、若山先生も熟知されていたはずである。もし私がES細胞をSTAP細胞だと偽って渡していたのなら、もともと増殖している細胞が渡されていたことになり、若山先生が観察した、増殖能の低いSTAP細胞からの無限増殖する幹細胞への変化は起こるはずがなく、気がつかなかったはずはないのではないだろうか。さらに若山先生はキメラ実験・STAP幹細胞樹立実験を行っていた当時、129 x B6 F1のES細胞は若山研に存在していなかったと多くのメディアにご発言されている。
そして、これらの実験に使われていたES細胞は若山研で飼育されていたアクロシンGFPマウスという特殊なマウスから作製されたものであったことも調査結果として発表された。アクロシンGFPマウスは、通常のGFPマウスでは観察されない「光るスペルム」を持っているという特徴があるそうだ。STAP幹細胞からできたキメラマウスのジャームライントランスミッションの実験の際、「光るスペルム」を自身で採取し実験を行っていたにもかかわらず、若山先生は「STA幹細胞は自分の研究室にはいなかったマウスからできた細胞だった」と6月の記者会見で発表し、まるで私がマウスや細胞をすり替えられたかのような推論を社会に植え付けた。
6月の終わりの検証実験参加の打ち合わせの帰り道に、STAP幹細胞が間違いなく若山研にいたマウスに由来しており、そのマウスがアクロシンGFPキメラであることがわかったと私は連絡を受けた。連絡をくださった方に「アクロシンGFPマウスはどんなマウスなんですか?」と伺うと、「スペルムがGFPで光るという性質を持っている」と教えてくれた。私は若山先生が光るスペメムを顕微授精する実験を行っていたことを思い出し、その時の実験の写真も残っていることも思い出した。「若山先生は光るスペルムで実験をしていました」と告げると、「確信犯」と言葉が返ってきた。
- 2019/05/28(火) 14:02:02|
- STAP事件
-
-
| コメント:12
胎盤の結論を出す前に根本に戻りましたね。まず桂報告書は、どんなカモフラージュがあろうと、実質的に「小保方さんが既存ES細胞で捏造した」と主張していることになるという証明は終わりました。次は桂報告書の既存ESによる捏造だという証明が正しく立証されているかという問題に移りますよね。そしてそれがちゃんと立証されていないときは可能性の枠は広げられないといけないですね。
①既存ESコンタミである。(小保方さんが犯人)
②論文通りの作られ方をしている。(何らかの理由で若山さんが本物を隠したことになる。この場合はヴァカンティ研から訴訟を起こされるリスクがある。ただし、再現検証実験では論文通りの再現は出来ず、キメラは作られなかった。)
③別のつくられかたをしている。(この場合も若山さんが事件の犯人だということになる。我々の知識ではntES化によるキメラとしか推測できないが、共培養等の別の手段もあるのかもしれないが、分からない。)
そして、今ntES仮説で諸実験結果が矛盾なく説明し得るかという検証の大詰めの胎盤蛍光問題の途中で、確認のために①の否定理由を集成しておこうということになった。これはこのブログの"桂報告書疑義"をも整理し直す作業になるでしょう。
ティシュー論文から始まるのでしょうね。楽しみだ。登場人物さんたち頼みます。
- 2019/05/27(月) 12:09:57|
- STAP事件
-
-
| コメント:50
さてさて、ほとんど解けかかってるんですけど難所ですかね。
GOF-ESとGLSの中身の一致は
①小保方さんが犯人。
②若山さんが小保方さんのGOF-ESチューブにGLSを入れかえた。従ってそんなことをする人は犯人。
二社択一なんです。
①は我々の検討結果ありえませんね。すると②になるんだけど、キメラのできた理由として、本物であるか、ntES化実験の結果であるかのどちらかだとすると、丹羽さんたちは再現できなかったんですよね。するとntESだということになるだけど、まだ何かスキっとした納得に至らない。ははは。急ぐ旅でない。
- 2019/05/25(土) 21:20:19|
- STAP事件
-
-
| コメント:44
さて、STAP細胞はそれ自体が幹細胞だったのではなかったのか。どうして、若山さんは最初のキメラ成功と同時に培養でその細胞を増殖誘導できたとき、キメラ形成する細胞が培養増殖もするのだから幹細胞だと小保方さんに論文を書かせなかったのか。なぜ小保方さんは自分の細胞は増殖しないということにこだわったのか。彼女は若山さんが増殖させているという事実をどうとらえていたのか。笹井さんがSTAP幹細胞はSTAP細胞のデリバティブであるという論旨に追い込まれたのはなぜなのか。2011年11月の最初のキメラの胎盤蛍光などは何も言われていなかった以上、同時培養増殖した細胞はSTAP細胞そのものであるはずなのに。
- 2019/05/18(土) 20:12:36|
- STAP事件
-
-
| コメント:63
【0030】
以下に実施例を示して本発明を具体的かつ詳細に説明するが、実施例は本発明を限定するものと解してはならない。
【実施例】
【0031】
A.倍化ESマウスの出産率の比較
本実験の目的は、複数の4倍体胚を用いることによりESマウスの作成効率の向上を図ることである。そのためにまず、正常胚を用いて、倍化胚(3xおよび5x)を作成し、その発生能を解析した。その結果、3x正常胚では正常な範囲内で産仔を得ることに成功したが(50%;8/16)、5x正常胚からは産仔を得るにいたらなかった(0%;0/8)。以上の結果から、過剰な胚の数は発生能を障害することが示唆された。この結果を元に本実験では1xから3xの4倍体胚とES細胞のキメラ胚を作成し、ESマウスの出産率を比較検討した。
【0032】
B.倍化4倍体胚の細胞数の解析
ESマウスの作成の前に、まず、倍化した4倍体胚の細胞数が実際に増加しているのかを検討した。授精後96時間の1xから3xの4倍体胚をそれぞれ10個ずつPIとCdx2の2重染色を行った。以前の報告(Koizumi N, Fukuta K. Preimplantation development of tetraploid mouse embryo produced by cytochalasin B. Exp Anim. 1995;44(2):105-109)と一致して、1x4倍体胚では正常胚(1x2n)と比較して総細胞数の減少が認められた(表1および図1A-A”、D-D”)。一方で、2x4倍体胚および3x4倍体胚では、総細胞数の増加が確認された(表1および図1B-B”、C-C”)。これらの2x4倍体胚および3x4倍体胚では、TE細胞数およびICM細胞数もそれぞれ増加していることが示された(表1)。また、3x4倍体胚を抗Oct3/4抗体で免疫染色したところ、正常にICM細胞が染色された(図1E-E”)。以上の結果から、胚盤胞期の倍化4倍体胚では、予想通り細胞数が増加しており(表1)、ICMなどへの分化も正常に行われることが証明された(図1E-E”)。
(表1.倍化4倍体胚の細胞数の解析)
【表1】
【0033】
C.倍化4倍体胚を用いたESマウスの作成効率の検討
先の実験で倍化4倍体胚の細胞数の増加が確認されたので、次にESマウスの作成効率の検討を行った。使用したES細胞は、一般的なES細胞株であるE14、ntES細胞として129B6F1G1(GFP陽性)およびBDmt2、ならびに近交系由来のES細胞株としてDFC3H(合計10株をランダムに使用)を用いた。図2に示したように、GFPを発現する129B6F1G1を用いてキメラ胚を作成したところ、3x4倍体胚にES細胞が正常に取り込まれることが示された(図2D-F)。
【0034】
次に、各ES細胞と1xないし3xの4倍体胚とのキメラ胚を作成し、胚移植後の発生能の検討を行った。1xおよび2x4倍体胚をホスト胚として用いた場合では、3系統のES細胞(E14、129B6F1G1およびBDmt2)は、ESマウスの作成効率が約1-3%と非常に低頻度であった(表2)。一方、3x4倍体胚を用いた場合では、その作成効率が約2.5-8.5倍に向上した(8.6%:表2)。また、従来作成が困難と考えられていた近交系由来のESマウスでも、3x4倍体胚を用いることにより約7%の成功率で作成可能であることが示された(表2)。
【0035】
以上の結果から、2x4倍体胚では正常胚と同程度の細胞数を有するが(表1)、ESマウスの作成効率は、3x4倍体胚を使用したものが最も効率がよく(表2)、従来法の1xもしくは2x4倍体胚では不十分であることが示された。
(表2.倍化4倍体胚を用いたESマウスの作成効率の検討)
【表2】
*:E14;一般的に用いられているES細胞。129B6F1G1およびBDmt2;ntES細胞。DFC3H;近交系由来ntES細胞。
【0036】
D.倍化4倍体胚より作成された新生仔ESマウスの解析
ESマウスは奇形仔の頻度が高いことが知られているので、次に3x4倍体胚より作成されたESマウスの表現型を観察した。3x4倍体胚由来のESマウスは、体重・胎盤重量とも1xまたは2x4倍体胚由来のESマウスと同程度であった(表3)。また開眼・腹部ヘルニア・巨大産仔の出現率なども差は認められなかった(表3)。以上の結果から、3x4倍体胚を用いた場合であってもESマウスの奇形頻度は、少なくとも従来のESマウスと同程度であることが示された。
【0037】
ESマウスの最大の特徴は、体を構成しているほぼ全ての細胞がES細胞由来であるという点である。表1に示したように、3x4倍体胚では、総細胞数のみならず、体を構成しうるICM細胞も増加している。最後に、3x4倍体胚由来のESマウスが実際にES細胞由来の細胞で構成されているかを検討した。
【0038】
ホストである4倍体胚由来の細胞とES細胞を区別するため、ES細胞にはE14、ホストにはICRGFP由来の4倍体胚を使用した。このことにより、4倍体胚由来の細胞がGFPを発現するため、ESマウスにて4倍体由来の細胞を容易に確認することが可能となる。この方法を用いて、3x4倍体胚から合計15匹のESマウスを作成し、蛍光顕微鏡を用いて観察を行った。図3に示すように、得られた15匹すべてにおいて、胎盤でGFPが発現し、産仔では発現していなかった(図3A-C)。これらの結果から、3x4倍体胚を使用してもESマウスに4倍体胚由来の細胞が大きく混入する可能性は低いと考えられる。次に、より詳細な解析を行うため、フローサイトメトリーによるGFP細胞の検出を行った。1xおよび3x4倍体胚より作成したESマウスをそれぞれ2匹ずつ使用し、脳および肝臓におけるGFP陽性細胞を検出した。解析の結果、1xおよび3x4
【0039】
倍体胚由来のESマウスとも、4倍体胚由来のGFP細胞の混入率は1%未満であった。
以上の結果から、3x4倍体胚を用いたESマウスは、従来法に由来するESマウスと同様に、ほぼ全ての構成細胞がES細胞由来であることが証明された。
(表3、ESマウスの表現型の解析)
【表3】
a平均±SD(g)。
bOE:開眼、AH:腹部ヘルニア、LO:巨大産仔、OELO:開眼および巨大産仔を併発した仔、OEAH:開眼および腹部ヘルニアを併発した仔。
【産業上の利用可能性】
【0040】
本発明は、トランスジェニック動物やノックアウト動物のごとき遺伝子改変非ヒト動物を効率的に作成する方法およびこの方法により作成された遺伝子改変非ヒト動物を提供する。そのため、病理学的な研究や新治療法の開発に用いる実験動物の製造、新薬の開発をはじめとする多くの産業分野において利用可能である。
【図面の簡単な説明】
【0041】
【図1】図1は、倍化4倍体胚の細胞数の解析結果を示す。A-Eは1xから3x4倍体胚および正常胚(1x2n)をPI染色した写真、A’-D’は1xから3x4倍体胚および正常胚(1x2n)をCdx2で免疫染色した写真、A”-D”はそれぞれの写真を合成した写真である。A-A”は1x4倍体胚、B-B”は2x4倍体胚、C-C”は3x4倍体胚、D-D”は正常胚(1x2n)、E-E”は3x4倍体胚をPI(E)、Oct3/4 (E’)で染色した写真、およびその合成写真をE”として示した。スケールバー:A”-D”100μm。E”200μm。
【図2】図2は、129B6F1G1を用いた4倍体胚とのキメラ胚の作成を示す。A-Cは1x4倍体胚とES細胞(129B6F1G)のキメラ胚、D-Fは3xx4倍体胚とES細胞(129B6F1G)のキメラ胚を示す。A、Dは可視光写真、B、Eは蛍光写真、C、Fはそれぞれの合成写真である。スケールバー:200μm。
【図3】図3は、3x4倍体胚から作出されたESマウスの解析である。(A-C)GFPを発現する3x4倍体胚とE14 ES細胞を用いたESマウスである。(A)可視光写真を示す。(B)蛍光写真を示す。(C)AとBの合成写真を示す。胎盤においてのみ4倍体胚由来(GFP陽性)の細胞が確認される。(D、E)フローサイトメトリーによる解析結果である。1x4倍体胚由来のESマウス(D)および3x4倍体胚由来のESマウス(E)の脳(左パネル)および肝臓(右パネル)におけるGFP陽性細胞の存在頻度を解析した。1xおよび3x4倍体胚由来のESマウスともGFP陽性細胞の存在頻度は1%未満であった。
【出願人】
【識別番号】503359821
【氏名又は名称】独立行政法人理化学研究所
【出願日】
平成20年1月4日(2008.1.4)
【代理人】
【識別番号】100081422
【弁理士】
【氏名又は名称】田中 光雄
【識別番号】100084146
【弁理士】
【氏名又は名称】山崎 宏
【識別番号】100116311
【弁理士】
【氏名又は名称】元山 忠行
【識別番号】100122301
【弁理士】
【氏名又は名称】冨田 憲史
【公開番号】
特開2009-159878(P2009-159878A)
【公開日】
平成21年7月23日(2009.7.23)
【出願番号】
特願2008-89(P2008-89)
- 2019/05/14(火) 11:31:47|
- 非ヒトES動物
-
-
| コメント:0
発明の名称】
非ヒトES動物の新規作成方法
【発明者】
【氏名】大田 浩
【氏名】坂出 祐子
【氏名】山縣 一夫
【氏名】若山 照彦
【課題】4倍体胚をホストとする非ヒトES動物の作成方法において、効率的に非ヒトES動物を得る方法を開発する。
【解決手段】本発明は、非ヒトES動物を作成する方法であって、3個または4個の4倍体胚にES細胞を移入してキメラ胚を作成し、次いでキメラ胚を偽妊娠非ヒト動物に移植することを含む方法を提供する。
【特許請求の範囲】
【請求項1】
非ヒトES動物を作成する方法であって、下記工程:
(a)3個または4個の4倍体胚にES細胞を移入してキメラ胚を作成すること;次いで
(b)該キメラ胚を偽妊娠非ヒト動物に移植すること
を含む方法。
【請求項2】
該4倍体胚が3個である、請求項1に記載の方法。
【請求項3】
該4倍体胚の発生時期が2細胞期または4細胞期である、請求項1または2に記載の方法。
【請求項4】
該ES細胞が近交系のES細胞である、請求項1~3のいずれか1項に記載の方法。
【請求項5】
請求項1~4のいずれか1項に記載の方法により得られうる、非ヒトES動物。
【発明の詳細な説明】【技術分野】
【0001】
本発明は、4倍体胚をホストとして用いる非ヒトES動物の作成方法およびその方法により作成された動物に関する。
【背景技術】
【0002】
遺伝子改変動物の作成による遺伝子機能の解析は、基礎生物学および医学分野の発展に大きな貢献を果たしてきた。遺伝子改変は主にES細胞において行われ、遺伝子改変動物を作成するためには、遺伝子改変後のES細胞を生殖細胞に分化させる必要がある。一般的に、ES細胞の生殖細胞への分化は、キメラ動物を作成することにより行われている。すなわち、ES細胞(ドナー)を初期胚(ホスト)へ注入させることによりキメラ動物を作出し、キメラ個体内でドナー由来の生殖細胞分化を誘導するという方法である(非特許文献1)。しかしながら、この方法ではホスト由来の生殖細胞も数多く存在するため、ES細胞由来の子孫を得るために多数の交配が必要な場合が多く、遺伝子改変動物の作出において大きな障壁となっていた。
【0003】
この問題を回避するため、4倍体胚由来の胚をホストとして用いる方法が開発されてきた。4倍体胚由来の細胞は体を構成する細胞へ分化できないが、胎盤を形成できることが知られている。この特徴を利用してES細胞と4倍体胚のキメラ胚を作出すると、その産仔の胎盤は4倍体胚由来で、体は全てES細胞由来となる(以下「ES動物」という)。ES動物ではほぼ全ての細胞がES細胞由来であるため、その生殖細胞も全てES細胞由来となる(非特許文献2および3)。このようにES動物を作出することにより、ES細胞由来の産仔を効率的に得ることが可能であるが、この技術の問題点はES動物の作成効率自身が非常に低いことであった。なお、本明細書において「ES動物」とは、ES細胞および4倍体胚から作成されるキメラ胚に由来し、体が実質的に全てES細胞に由来する細胞からなる動物をいう。
【0004】
これまでにES動物の作成効率を改善するために行われてきた方法は、主にES細胞の樹立という観点からであった。例えば、F1由来のES細胞を樹立することにより、ES動物の作成効率の改善が可能であると報告されている(非特許文献4)。この方法により確かにES動物の作成効率は改善したが、この方法ではこれまで樹立されているES細胞には適用できない。すなわち、大部分のES細胞に対しては依然として有効な方法は存在していなかった。
【非特許文献1】Capecchi MR. The new mouse genetics: altering the genome by gene targeting. Trends Genet 1989;5:70-76
【非特許文献2】Nagy A, Gocza E, Diaz EM et al. Embryonic stem cells alone are able to support fetal development in the mouse. Development 1990;110:815-821
【非特許文献3】Nagy A, Rossant J, Nagy R et al. Derivation of completely cell culture-derived mice from early-passage embryonic stem cells. Proc Natl Acad Sci USA 1993;90:8424-8428
【非特許文献4】Eggan K, Akutsu H, Loring J et al. Hybrid vigor, fetal overgrowth, and viability of mice derived by nuclear cloning and tetraploid embryo complementation. Proc Natl Acad Sci USA 2001;98:6209-6214
【発明の開示】
【発明が解決しようとする課題】
【0005】
したがって、本発明の解決課題は、4倍体胚をホストとする非ヒトES動物の作成方法において、効率的に非ヒトES動物を得る方法の開発であり、かつこれまでに樹立されているあらゆるES細胞に対して適用可能にすることである。
【課題を解決するための手段】
【0006】
本発明者らは、上記課題を解決するために鋭意研究を行った。そして驚くべきことに、3個または4個の4倍体胚にES細胞を移入する方法を試みたところ、ホストとして使用する4倍体胚の機能が大幅に改善されることを見出した。さらに、本発明者らは、この方法がこれまで樹立されているES細胞に適用されうることを示した。
【0007】
すなわち、本発明は:
(1)非ヒトES動物を作成する方法であって、下記工程:
(a)3個または4個の4倍体胚にES細胞を移入してキメラ胚を作成すること;次いで
(b)該キメラ胚を偽妊娠非ヒト動物に移植すること
を含む方法、
(2)該4倍体胚が3個である、(1)に記載の方法、
(3)該4倍体胚の発生時期が2細胞期または4細胞期である、(1)または(2)に記載の方法、
(4)該ES細胞が近交系のES細胞である、(1)~(3)のいずれか1項に記載の方法、
(5)(1)~(4)のいずれか1項に記載の方法により得られうる、非ヒトES動物を提供するものである。
【発明の効果】
【0008】
本発明によれば、きわめて効率的に非ヒトES動物を得ることができ(従来法と比べて数倍の効率)、しかも従来は困難とされていた近交系ES細胞などからでもES動物を作成することができる。
【発明を実施するための最良の形態】
【0009】
本発明は、非ヒトES動物の作成方法に関するものである。本発明の方法に使用される動物はヒトを除く動物であってよいが、好ましいのは哺乳類である。適する動物の例として、マウス、ラット、モルモット、ウサギ、ウシ、ヒツジ、ヤギ、ウマ、ならびにアカゲザル、チンパンジーなどの霊長類が挙げられるが、これらだけに限定されない。現在、トランスジェニック動物やノックアウト動物に関して研究が進んでいる実験動物としてマウスが挙げられ、本発明の方法を適用するのに最も好ましい動物の一例である。
【0010】
本発明の方法を、手順を追って説明する。
本発明では、まず、ホスト胚として4倍体胚を作成する。ここで、「ホスト」とはES細胞を導入する動物と同種類の動物に由来することをいう。4倍体胚は、当業者間で周知の方法によって作成することができ、例えば、胚の融合によって得られてよい。胚の融合を、電気融合法、注入法などのごとき一般的な手法を用いて行ってもよい。本発明で用いられうる胚の融合の方法は、好ましくは電気融合法である。また、電気融合を、当該技術分野で一般的に用いられる電気融合装置を用いて行ってもよい。一般的な電気融合では、例えば、複数の2細胞期胚を直線状に並べ、両側を平行二重電極で挟み、電気融合を行ってよい。その後、電気融合に成功した胚を選別し、培養して4倍体胚を得ることができる。4倍体胚の培養は、例えば、CZB培地にて一晩行ってもよい。これらの条件は当業者が適宜定めうる。
【0011】
本発明では、例えば、4倍体胚を2種類の胚を融合して作成してもよい。一方の胚を、当該技術分野において一般的な手法である顕微授精(IVF)を用いて作成してもよい。例えば、BDF1マウスの精子および卵子から、IVFにより胚を得てもよい。ここで、「BDF1マウス」はC57BL/6の雌とDBA/2Nの雄の交配によって作出される当該技術分野で一般的な系統をいう。IVFは、例えば、卵子を過排卵処理を施した雌から採取し、TYH培地にて培養し、精子を約1×105細胞/mlのごとき一定濃度となるように添加して行うことができる。その後、授精した胚を培養して2細胞期の胚を得てもよい。この時の培養は、例えば、CZB培地にて24時間行ってもよい(Chatot CL, Ziomek CA, Bavister BD et al. An improved culture medium supports development of random-bred 1-cell mouse embryos in vitro. J Reprod Fertil 1989;86:679-688)。もう一方の胚を、例えば、動物を交配させることにより得てもよい。例えば、ICRマウスの雄および雌を一晩交配させ、翌朝交配を確認し、その24時間後に卵管を採取して2細胞期の胚を回収することにより胚を得てもよい。「ICRマウス」とは、一般的に使用される系統のマウスで、仔を多く産み、子育てを得意とする性質を有する。好ましくは、ICRマウスの雄がGFP遺伝子を有する。例えば、pCAG-EGFPベクターを有するICRバックグランドのGFPトランスジェニックマウス(ICRGFP)を含むが、これだけに限らない。これにより、ES動物を作成した際に、ホスト4倍体胚に由来する細胞をGFPタンパク質の発現で確認できることが好ましい。
【0012】
また、本発明に用いられる4倍体胚は、1種類のみの胚から作成されてもよい。1種類の胚は、例えば、前記のようにIVFまたは交配により作成される2細胞期の胚を含んでよいが、それらだけに限らない。所望により胚の融合は前記の方法と同様に行ってもよい。
【0013】
次いで、4倍体胚をディッシュなどの適当な容器に入れ、ES細胞を移入し、培養してキメラ胚を作成する。4倍体胚をホストとしてES動物を作成する際に、一般的には、1個または2個の4倍体胚が使用されている(非特許文献2および3)。本発明における4倍体胚の個数は、好ましくは3個または4個であり、最も好ましくは3個である。本発明において、3個の4倍体胚をホストとしてES動物を作成する場合には、1個または2個の4倍体胚を用いる場合に比較して、目的とするES動物の作成効率が2.5~8.5倍程度上昇する。一方、5個またはそれ以上の4倍体胚を用いる場合には、ES動物の作成効率は3個または4個の場合より低下する。また、本発明で用いられる4倍体胚の発生時期は、電気融合直後の1細胞期以上であってよく、好ましくは、2細胞期から柔実胚期までである。最も好ましくは、4倍体胚の発生時期が2細胞期または4細胞期である。本発明では、4倍体胚を適当な容器に入れる前に、アシッドタイロード溶液などの当業者間で一般的な溶液を用いて透明帯を除去してもよい。本発明において、ES細胞の移入工程でディッシュを用いる場合には、当該分野で一般的に用いられるものであってよく、例えば、プラスチックディッシュを用いてもよい。その場合において、好ましくは1ウェルごとに小さな窪みを作成し、ES細胞の移入の際に4倍体胚を支持できるようにすることにより作成効率を向上させることができる。従って、本発明は、このような4倍体胚を支持できる窪みを有する容器、ならびに該容器を必須構成成分として含むES動物作成用キットなども提供する。
【0014】
本明細書で用いられる「ES細胞」は、胚盤胞期の受精卵の内部細胞魂(ICM)に由来する、インビトロで未分化状態を保ったまま培養維持できる細胞を意味する。これまで、ホストとして4倍体胚を用いる効率的なES動物の作成においては、樹立されたF1由来のES細胞が使用されている(非特許文献4)。しかし、本発明では、ES細胞はあらゆる種類のものであってよく、当業者間で一般的に用いられるものを適用することができる。例えば、E14、R1またAB-1などの129系統に由来する細胞株、あるいはTT2などの市販されている細胞株を含むが、これだけに限らない。また、本発明で用いるES細胞は、近交系または交雑系に由来する上記のごとき一般的なES細胞株であってよく、好ましくは近交系に由来するものである。本明細書で用いられる「近交系」とは、20世代またはそれ以上にわたり兄妹交配が行われてきた系統のことをいう。また、129B6F1G1、BDmt2またはDFC3Hのごとき核移植胚由来のES細胞(ntES細胞)を本発明の方法に用いることができる。これらのntES細胞は発明者らの研究室にて樹立された。ここで、129B6F1G1はGFPを発現する129B6F1系統のセルトリ細胞であり、BDmt2はBDF1系統の繊維芽細胞を用いた核移植胚由来のntES細胞であり、ならびにDFC3HはC3Hの脳細胞から樹立した近交系に由来するntES細胞である。
【0015】
さらに、本発明に用いられうるES細胞は、ES細胞に類似した性質(本発明でドナーのES細胞として用いることができる性質)を有する細胞であってもよく、例えば、人工多能性幹(iPS)細胞などを含むが、それだけに限らない。
【0016】
また、本発明に用いられるES細胞において、特定の遺伝子が導入されているか、または特定の遺伝子がノックアウトされていてもよい。
【0017】
ES細胞を適当な条件で培養してから、4倍体胚に移入することができる。ES細胞の培養は、当該技術分野において一般的な方法を用いて行われてよい。例えば、基本培地として、KNOCKOUT(登録商標)DMEM(Invitrogen, CA, USA)を用いてよい。また、添加物として20%胎児ウシ血清(Sigma-Aldrich, MO, USA)、白血病抑制因子(LIF)(1000ユニット/ml,Invitrogen)、1%ペニシリン-ストレプトマイシン(invitrogen)、1%L-グルタミン(Specialty Media, NJ, USA)、1% 非必須アミノ酸(Specialty Media)、1% ヌクレオシド(Specialty Media)、または1% β-メルカプトエタノール(Specialty Media)を添加して培養したものであってよい。
【0018】
また、本発明では、ES細胞の培養は、ゼラチンでコートしたディッシュ(フィーダー細胞非存在)上で行われてもよい。
【0019】
本発明では、4倍体胚に移入されるES細胞の数は、1個以上であってよい。また、4倍体胚に移入されるES細胞の数は、好ましくは8~15個である。移入されるES細胞は細胞塊として移入されてよい。本発明における移入は、例えば、ES細胞を4倍体胚に接触させる、あるいは注入することによって行ってもよい。これらの操作は、顕微鏡またはマイクロマニピュレータを使って行われるのが一般的である。
【0020】
本明細書で用いられる「キメラ胚」は、由来の異なる2個以上の胚由来の細胞の結合により得られた胚を意味する。本発明に用いられうるキメラ胚は、好ましくは3個または4個の4倍体胚にES細胞を移入して培養した胚である。キメラ胚の培養は、例えば、CZB培地にて約24時間行われてよい。これらの条件は当業者が適宜定めうる。
【0021】
次に、3個または4個の4倍体胚にES細胞を移入し、適当な条件で培養することにより得られたキメラ胚を偽妊娠非ヒト動物に移植して、子供を産ませ、ES動物を得る。キメラ胚を、偽妊娠非ヒト動物の卵管内または子宮内に移植してもよい。偽妊娠非ヒト動物は、ES細胞が由来する動物と同種の動物であればよく、系統が異なっていてもまたは同一であってもよい。例えば、偽妊娠非ヒト動物は、偽妊娠2.5日目のICRマウスであってもよいが、これだけに限定しない。本発明で用いられるキメラ胚の移植は、当業者間で一般的に用いられる方法によって行われうる。次いで、当業者間で一般的な方法に従って仮親(キメラ胚を移植された偽妊娠非ヒト動物)を飼育し、出産させてES動物を得ることができる。
【0022】
前記のごとく、3個または4個、とりわけ3個の4倍体胚をホストとしてES動物を作成すると、従来の方法と比較して作成効率を高めることができる。また、この本発明の方法では、あらゆるES細胞が使用でき、例えば近交系由来のES細胞でも使用可能という利点を有する。さらに本発明の方法により得られたES動物は、奇形頻度が従来のESマウスと同程度であり、十分に実用に供することができる。
【0023】
次に、下記の実施例に用いた主な材料および方法を示すが、本発明を限定するものではない。
【0024】
使用したマウスおよびES細胞
BDF1およびICRマウスはSLC(浜松、日本)から購入し使用した。ICRバックグラウンドのGFPトランスジェニックマウスはpCAG-EGFPをベクターとして以前発明者らの研究室で樹立したものを使用した。使用したES細胞は4種類で、一般的なES細胞としてE14(Hooper M, Hardy K, Handyside A et al. HPRT-deficient (Lesch-Nyhan) mouse embryos derived from germline colonization by cultured cells. Nature 1987;326:292-295)、核移植胚由来のES細胞(ntES細胞)として、129B6F1G1、BDmt2およびDFC3Hを使用した。E14は、1985年にMartin Hooper博士(Edinburgh, Scotland)らにより樹立され、Peter Mombaerts博士(Rockefeller University)が培養・保存していたものを分与して頂いた。129B6F1G1は、GFPを発現する129B6F1系統のセルトリ細胞、BDmt2はBDF1系統の繊維芽細胞を用いた核移植胚由来のntES細胞で、以前発明者らの研究室で樹立されたものを使用した。また近交系由来のntES細胞として、C3Hの脳細胞から樹立したntES細胞(DFC3H)を10株ランダムにESマウスの作成に使用した。
【0025】
ES細胞の培養条件
ES細胞の培養は一般的な条件に従って行った。基本培地としてKNOCKOUT(登録商標)DMEM(Invitrogen, CA, USA)を用い、下記の添加物を加えたものを培養液として使用した:20%胎児ウシ血清(Sigma-Aldrich,MO,USA)、白血病抑制因子(LIF)(1000ユニット/ml,Invitrogen)、1%ペニシリン-ストレプトマイシン(invitrogen)、1% L-グルタミン(Specialty Media, NJ, USA)、1% 非必須アミノ酸(Specialty Media)、1% ヌクレオシド(Specialty Media)、1% β-メルカプトエタノール(Specialty Media)。ES細胞はゼラチンコートを施したディッシュ上においてフィーダー細胞非存在下で培養した。
【0026】
4倍体胚の作成とESマウスの作成
本実験で使用した4倍体胚は2種類の胚から作成した。1つはBDF1の精子と卵子を顕微授精(IVF)させた胚(BDF2)であり、もう1つはICRGFP雄とICR雌を交配させた胚を使用した。後者の胚(ICRGFP)ではGFPを発現するため、4倍体胚に由来する細胞の存在を緑色蛍光により確認できる。IVFは一般的に用いられている方法を用いて行った。具体的には、BDF1マウスの精巣上体尾部から精子を回収し、TYH培地(Toyoda Y, Yokoyama M, Hoshi T. Studies on fertilization of mouse eggs in vitro: I. In vitro fertilization of eggs by fresh epididymal sperm. Jpn J Anim Reprod 1971;16:147151)にて培養した。卵子は過排卵処理を施した雌BDF1から採取し、TYH培地を用いて培養した。ここに精子を1x105細胞/mlになるように添加することによりIVFを行った。IVF後、授精した胚を回収し、2細胞期胚を得るため、CZB培地(Chatot CL, Ziomek CA, Bavister BD et al. An improved culture medium supports development of random-bred 1-cell mouse embryos in vitro. J Reprod Fertil 1989;86:679-688)において24時間培養した。一方、ICRGFP胚を得るためには、過排卵処理を施したメスICRと、オスICRGFPマウスを一晩交配させた。翌朝、交配を確認し、その24時間後に卵管を採取し、2細胞期の胚を回収した。BDF2およびICRGFPの2細胞期の胚を、電気融合装置(Model LF101、ネッパジーン(株)、千葉)を用いて電気融合させ、4倍体胚を作成した。電気融合に成功した胚を選別し、CZB培地にて一晩培養した。
【0027】
2~4細胞期に発生した4倍体胚を選別し、アシッドタイロード溶液を用いて透明帯を除去した。次いで、プラスチックディッシュ上に作成した小さな窪みに、1個(1x)から3個(3x)の4倍体胚を入れた。さらにその上から8~15個のES細胞の細胞塊を入れることにより、ES細胞と4倍体胚のキメラ胚を作成した。CZB培地にて24時間培養後、得られたキメラ胚を偽妊娠マウス(偽妊娠2.5日目)の子宮内に戻した。これらのマウスを妊娠18日目で帝王切開し、産仔の有無を解析した。
【0028】
胚盤胞期胚の細胞数の解析
胚盤胞期の細胞数を数えるため、ヨウ化プロピジウム(PI)染色およびCdx2の免疫染色を行った。PIでは全ての細胞が染まるため、全細胞数の指標として使用し、抗Cdx2抗体では栄養外胚葉(TE)細胞のみが染まるため、TE細胞の細胞数の指標として使用した。細胞内部魂(ICM)の細胞数を、全細胞数(PI陽性細胞)からTE細胞数(Cdx2陽性細胞)を差し引いた細胞数として計算した。具体的には、1xから3xの4倍体胚を胚盤胞期まで発生させ、4%パラホルムアルデヒドを用いて固定し(室温で45分間)、PBSで3回洗浄後、抗Cdx2抗体(1:200; BioGenex, CA, USA)を室温で1時間反応させた。PBSで3回洗浄後、2次抗体(goat anti-mouse IgG conjugated with Alexa Fluor 488; Molecular Probes, Eugene, OR, USA)と室温で30分間反応させた。PBSで3回洗浄後、1μg/mlのPI溶液に移し、共焦点顕微鏡を用いてそれぞれの胚盤胞期胚を撮影した。細胞数のカウントには、焦点写真をMetaMorph software(Universal Imaging Co., Downingtown, PA, USA)を用いて立体構築し、PI陽性細胞数およびTE陽性細胞数をカウントした。この解析には、正常胚(1x2n)および1xないし3xの4倍体胚に由来する胚盤胞期の胚をそれぞれ10個ずつ使用してカウントした。
【0029】
新生仔ESマウスの解析
ESマウスを妊娠18日目で帝王切開し、体重、胎盤重量の測定、および奇形(腹部ヘルニア、開眼、巨大産仔)の有無を観察した。また、ホストとしてICRGFPの4倍体胚を用いた実験では、ESマウスにおいて4倍体胚由来の細胞の有無を調べるため、ESマウスの肝臓および脳組織を用いてフローサイトメトリーを行った。具体的には、新生仔ESマウスから肝臓および脳を摘出し、ハサミで細切後、0.25%トリプシン-EDTA溶液で培養した(37℃で15分間)。10%FBSを含むDMEMを等量添加後、遠心分離し(300gで7分間)、上清を捨てた後、PBSで同様に2回洗浄した。細胞を10%FBS含有のDMEMに懸濁し、フローサイトメトリーに使用した。フローサイトメトリーにはFACSaria(BD Biosciences, San Jose, CA, USA)を用い、それぞれの組織における主要細胞群を側方散乱(SSC)および前方散乱(FSC)を指標として同定した。主要細胞群中のGFP陽性細胞数を測定することにより、4倍体胚由来の細胞を同定した。
- 2019/05/14(火) 11:26:56|
- 非ヒトES動物
-
-
| コメント:0
(感想氏の英文)
Interview with Prof. Chong Li
Prof. Li (李) is an associate professor in TongJi University (同济大学), one of the top universities in China, located in Shanghai city. He was a member of Wakayama lab in RIKEN CDB in Japan. A storage box containing the tubes of ES cells has been found in a freezer in Obokata lab. This box had Prof. Li's name on it. There are still debates on what are these ES cells and why they were found in Obokata lab. Althogh Ms. Obokata said she just stored it after she found the box when Wakayama lab was moved to Yamanashi University, some people are casting doubts on what she said. I had an interview with Prof. Li via e-mail to clarify the facts about the box and the ES cells. He kindly answered to all of my questions. He wrote he is feeling a sense of responsibility to tell the truth to the public.
Here I present all of my questions and Prof. Li's answers without editing under his permission.
Q - Kanso
A - Prof. Li
Chong Li教授へのインタビュー
Li(李)教授は上海市にある中国最高の大学のひとつである同済大学の准教授である。 日本の理研CDBの若山研究室のメンバーであった。小保方研究室の冷凍庫にES細胞チューブを収容した収納ボックスが見つかっている。この箱には教授の名前があった。これらのES細胞が何であるか、なぜそれらが小保方研究室で見つかったのかについてまだ議論がある。 小保方さんは若山研究室が山梨大学に移転した時に箱を見つけ、その後保管していたと言っているが、一部の人々は彼女の言葉を疑っている。私はボックスとES細胞についての事実を明らかにするために電子メールでLi教授へのインタビューを行った。受けました。 彼は私の質問すべてに親切に答えてくれた。 彼は真実を一般に伝える責任感を感じていると書いている。
ここで私は彼の許可の下で編集せずに私の質問と李教授の答えのすべてを提示する。
Q - 感想
A - 李教授
(感想氏の英文)
Q. RIKEN found a box labeled "ntES Box Li" in the freezer in Obokata lab. Is this your box? The box contained total 78 tubes labeled with the names of ntES cells dated at 2011.7.8 or 2011.7.30. The box seemed to be found around 2014.5.14.
Q.理化学研究所の冷凍庫に「ntES Box Li」と表示された箱が見つかりました。 これはあなたの箱ですか? ボックスは、2011.7.8または2011.7.30の日付が記されたntES細胞の名前がラベルされた計78本のチューブを含んでいた。 その箱は2014.5.14辺りに発見されたようだ。
(李氏の日本文)
A. もちろん、あの箱の中の細胞が私が作成したntES細胞です。すべてのクレーオチューブに貼ったラベルが私の実験ノートにバックアップが御座います。
A. もちろん、あの箱の中の細胞は私が作成したntES細胞です。すべてのクライオチューブに貼ったラベルは私の実験ノートにバックアップが御座います。
(感想氏の英文)
Q. Were these ntES cells in the box required for your work? If so, why did you give up finding the box?
Q.ボックス内のこれらのntES細胞あなたの仕事に必要だったのですか? もしそうであったのなら、なぜあなたはその箱を見つけることをあきらめたのですか?
(李氏の日本文)
A. この78本の細胞がまだ未解析のサンプルです。これは当時の実験で、未だに発表してない研究内容でした。CDBから離れた時、若山先生に未完成の研究を将来共同研究の形にやると話しました。細胞が若山研に保存することになります。ですから、すでに若山研にあるべきの細胞を探すことが御座いません。当然、放棄することが御座いません。細胞がなくなったことより、研究を支障に出ることが間違いありません。
A. この78本の細胞はまだ未解析のサンプルです。これは当時の実験で、未だ発表してない研究内容でした。CDBから離れた時、若山先生に未完成の研究を将来共同研究の形でやると話していました。細胞を若山研に保存することになります。ですから、すでに若山研にあるべき細胞を私が探すことは御座いません。当然、放棄することも御座いません。細胞がなくなったことにより、研究に支障が出ることは間違いありません。
(感想氏の英文)
Q. There was an article in a magazine telling that you gave up doing research in Yamanashi University because of the disappearance of the box. Is this story true?
Q.雑誌には箱がなくなったために山梨大学での研究をやめたという記事がありました。 この話は本当ですか?
(李氏の日本文)
A. 週刊誌の記事がもちろん嘘です。日本の研究業界のことが多分XXX様がもっと詳しいです。簡単に大学教員の就職が極めて難しいです。私が元々山梨大に仕事することも考えましたが、ポジションがないから、中国に就職しました。私が2012年の8月に帰国したが、細胞があの人の冷凍庫に発見したのは2014年の6月でした。だから、細胞が見つからないから、山梨大に行かないことが冗談みたいです。
A. 週刊誌の記事はもちろん嘘です。日本の研究業界のことは多分XXX様の方がもっとお詳しいでしょう。簡単に大学教員の就職を求められるかというと、それは極めて難しいです。私は当初山梨大で仕事することも考えましたが、ポジションがなかったから、中国で就職しました。私は2012年の8月に帰国しましたが、細胞があの人の冷凍庫で発見されたのは2014年の6月でした。ですから、細胞が見つからないから、山梨大に行かないということは冗談みたいな話です。
(感想氏の英文)
Q. When did you last see your box? When did you know the box was missing? What did you do after that? Did the other members of Wakayama lab help you?
Q.あなたがあなたの箱を最後に見たのはいつですか? 箱がなくなったことに気づいたのはいつですか? その後あなたはどうしましたか? 若山研究室の他のメンバーはあなたを手伝ってくれましたか?
(李氏の日本文)
A. 最後に箱を見たのは、帰国前のチェックでした。若山研究室が山梨大に引っ越したとき、あの箱について、研究室同僚とのメールやり取りもございます。あれは2013年の2月のことで、恐らく、2013年2月まで、あの箱がまだ若山研の冷凍庫に保管されてました。当時、あの細胞箱が山梨大に移るという結論に出したが、その後行方不明になりました。
A. 最後に箱を見たのは、帰国前のチェックでした。若山研究室が山梨大に引っ越したとき、あの箱について、研究室同僚とのメールやり取りもございます。あれは2013年の2月のことで、恐らく、2013年2月まで、あの箱はまだ若山研の冷凍庫に保管されてました。当時、あの細胞箱は山梨大に移管するという結論になりましたが、その後行方不明になりました。
(感想氏の英文)
Q. What are the genotypes of these ntES cells? Are these nuclear transferred ES cells from GOF (Oct4-GFP) mice?
Q.これらのntES細胞の遺伝子型は何ですか? これらの核移植されたES細胞はGOF(Oct4-GFP)マウスですか?
(李氏の日本文)
A. 多分それは皆様に最も関心を持ってることです。その細胞が悪用されたかどうか。残念ですが、B6D2F1のntESです。あの人の実験に何にも役割がない細胞でした。あくまでも私の解析ですが、細胞のラベルに“RG”という表示が御座います。多分あの人がこの細胞がGFPとRFP両方入ってると勝手に思いながら、自分の冷凍庫に入れました。(それはある先生の提案で、あの人にRFPを持ってるキメラマウスも作製しなさいという指示で、若山研がなかなかRFPを持つES細胞がないことから、宝が見つかったではないでしょうか)
A. 多分それは皆様が最も関心を持ってることでしょう。その細胞が悪用されたかどうか。残念ですが、B6D2F1のntESです。あの人の実験には何にも役割がない細胞でした。あくまでも私の推測ですが、細胞のラベルに“RG”という表示が御座います。多分あの人がこの細胞がGFPとRFP両方入ってると勝手に思いながら、自分の冷凍庫に入れたのではないでしょうか。(それはある先生の提案で、あの人にRFPを持ってるキメラマウスも作製しなさいという指示で、若山研になかなかRFPを持つES細胞がないことから、宝を見つけたつもりではなかったでしょうか)
(感想氏の英文)
Q. Dr. Obokata says she found the box left in a freezer when Wakayama lab was being moved to Yamanashi University. She say she “stored” it. What do you think about what she says?
Q.小保方博士は若山研究室が山梨大学に移籍された時に冷蔵庫に残されていた箱を見つけたと言っている。彼女はそれを「保管」した言っています。 彼女の言うことをどう思われますか?
(李氏の日本文)
A. 彼女の話が信用できますか?私の細胞がトータル4箱が御座います。なぜ別の3箱が現在正常に山梨大若山研の冷凍庫に保存してますか?なぜこの一箱があの人の冷凍庫に見つかったか?答えが簡単です。あの人が4つの箱が全部チェックしただと思います。ただ、この箱の中身が将来の実験にも使えるものだ。簡単に言うと、警察さんがあの3箱に指紋チェックをしたら、すべてわかるだと思います。今回の刑事訴訟が警察側の怠慢もあります。私に数時間の事情聴取をしても、あの変な結論を出したのは、私が被害者として納得できません。
A. 彼女の話を信用できますか?私の細胞はトータル4箱御座います。なぜ別の3箱が現在正常に山梨大若山研の冷凍庫に保存されているのですか?なぜこの一箱だけがあの人の冷凍庫に見つかったのですか?答えは簡単です。あの人は4つの箱を全部チェックしたのだと思います。ただ、この箱の中身だけが将来の実験にも使えるものだと考えたのでしょう。簡単に言うと、日本の警察があの3箱の指紋チェックをしたら、すべてわかることだと思います。今回の刑事訴訟は警察側の怠慢もあります。私に数時間の事情聴取をしただけで、あの変な結論を出したのは、私は被害者として納得できません。
(感想氏の英文)
Published at 7/31/2016
Note by Kanso: In Prof. Li's answers, "あの人" "彼女" means Ms. Obokata. "XXX" is my real name.
2016年7月31日に公開
感想による注:李教授の回答の中の「あの人」「彼女」は小保方さんを意味している。 「XXX」は私の本名です。
- 2019/05/14(火) 11:11:20|
- 李感想問答
-
-
| コメント:2
(英文)
CURRICULUM VITAE
NAME: Chong LI GENDER: Male
DATE OF BIRTH: 22 November 1980
NATIONALITY: China
LABORATORY ADDRESS:
Tongji University School of Medicine
Tongji University Advanced Institute of Translational Medicine
China Stem Cell Bank, East China Branch
No. 1239, SiPing Road, Shanghai, 200092, China
Phone/Fax: +86-21-6598-4257
Email: lichong@tongji.edu.cn
履歴書
名前:Chong LI 性別:男性
生年月日:1980年11月22日
国籍:中国
病院住所:
同済大学医学部
同済大学先端探索医学研究所
中国幹細胞バンク、東中国支店
No. 1239、SiPing Road、Shanghai、200092、China
電話/ファックス:+ 86-21-6598-4257
電子メール:lichong@tongji.edu.cn
(英文)
EDUCATION:
Ph.D. in Reproductive Biology, Kwansei Gakuin University, March 2010
M.S. in Developmental Biology, Kwansei Gakuin University, March 2007
B.S. in Biology, Shanghai Normal University, July 2003
Junior college education in Fine chemical, Shanghai Institute of Technology, July 2001
Senior high school, Shanghai Wuai Senior High School, July 1998
教育:
博士号 再生細胞学、関西学院大学、2010年3月
修士号 発生生物学、関西学院大学、2007年3月
学士号 生物学、上海師範大学、2003年7月
ファインケミカルに関する短期大学教育 上海工科大学、2001年7月
高等学校 上海武夷高等学校、1998年7月
(英文)
RESEARCH/EMPLOYMENT HISTORY:
Research Associate Professor September 2012~
Tongji University School of Medicine, Department of Regenerative Medicine
Research Scientist April 2010 to August 2012
RIKEN, Center for Developmental Biology, Laboratory for Genomic Reprogramming
Junior Research Associate April 2008 to March 2010
RIKEN, Center for Developmental Biology, Laboratory for Genomic Reprogramming
研究/職歴:
研究准教授2012年9月~
同済大学医学部、再生医学科
研究科学者2010年4月~2012年8月
理化学研究所ゲノムリプログラミング研究センター
ジュニアリサーチアソシエイト2008年4月~2010年3月
理化学研究所ゲノムリプログラミング研究センター
- 2019/05/14(火) 10:46:36|
- 李履歴書
-
-
| コメント:0
(英文)
Contamination in FI-SC samples
Examination of allele frequencies in RNA-seq fragments enables detection of specific characteristics of the sampled cells. When a set of genes specifically expressed in a particular cell type shows a distinct genotype, the origins of the cells can be assumed. This analysis of SNPs in mRNA sequence reads from the Obokata et al. study showed that the origins of the STAP and FI-SC samples were likely different than originally reported. The simulation showed that the mode of the distribution depends both on the cellular composition of the sample and possible variance caused by PCR bias. When we ignore the PCR bias of binomial distribution, the ratio of contaminating cells can be roughly estimated by the peak position in the distribution.
FI幹細胞サンプルの汚染
RNA-seq断片の対立遺伝子頻度の検討はサンプリングされた細胞の特性検出を可能にする。特定の細胞型において特殊に発現される一組の遺伝子が異なる遺伝子型を示す場合、その細胞の起源を想定することができる。メッセンジャーRNA配列におけるSNPに関するこの分析は、提示された小保方らの研究から、STAPとFI幹細胞サンプルの起源が最初に報告されたよりも異なっているらしいことを読み取る。シミュレーションは分布の様態がサンプルの細胞組成とPCRバイアスに起因する分散の両方に依存することを示している。二項分布のPCRバイアスを無視すると、我々は混入細胞の割合を大雑把に分布のピーク位置によって推定することができる。
(英文)
The most likely cell type to be contaminating the FI-SCs in the original study, TSCs, had many more heterozygous (B6/non-B6) alleles than non-B6 homozygous alleles. SNPs of FI-SCs were counted in alleles where B6 and 129 have different nucleotides and duplicated experiments resulted in 6859 and 7243 heterozygous SNPs and 24 and 14 non-B6 homozygous alleles, respectively. The percentage of contaminating cells can be assumed with genotype observation because the peak allele frequencies were at approximately 95–96%, whereas the population peak is expected to be at approximately 10%. This result was concordant with the expression of TSC marker genes. All of the examined TSC marker genes examined were expressed in approximately 10% of TSCs (Fig. 4A).
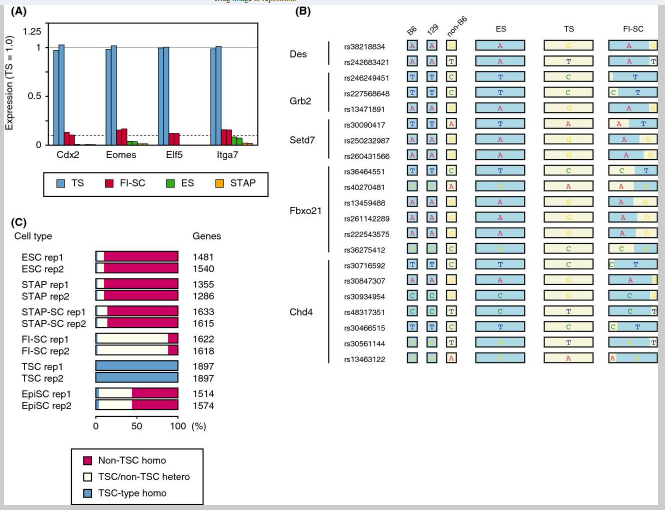
最初の研究でFI肝細胞を汚染する最も可能性の高い細胞型であるTS細胞は、非B6同型接合対立遺伝子よりももっと多くのヘテロ接合(B6/非B6)対立遺伝子を有していた。 FI幹細胞のSNPは、B6および129が異なるヌクレオチドを有し、かつそれぞれ、6859と7243のヘテロ接合SNPおよび24と14の非B6ホモ接合性対立遺伝子に結果された重複実験をされた対立遺伝子の中で数えられた。混入細胞の割合は遺伝子型観察によって推定することができる。なぜなら総数のピークが約10%であることが期待されているのに対して、ピークの対立遺伝子頻度が約95から96パーセントであったためである。この結果はTS細胞のマーカー遺伝子の発現と一致している。調べられたTS細胞のマーカー遺伝子の全てが約10%のTS細胞(図4A)の中で発現している。
(Fig. 4のリジェンド)
Figure 4. Examination of incorporated mRNAs in FI-SCs. (A) Expression of TSC marker genes in ESCs, TSCs, and FI-SCs. The solid line indicates the average TSC gene expression and dashed line indicates 10% of the average. (B) Heterozygous SNPs detected in Des, Grb2, Setd7, Fbxo21, and Chd4 where B6 and 129 share the same SNPs but TSCs did not. (C) Gene-wise analysis of distribution of heterozygous/homozygous SNPs having TSC-specific alleles.
[図4] FI幹細胞に組み込まれたメッセンジャーRNAの検討。 (A)ES細胞、TS細胞及びFI幹細胞の中のTS細胞マーカー遺伝子の発現。実線は平均のTS細胞遺伝子発現を示し、破線は平均の10%を示している。 (B)B6と129が同じSNPを共有し、TS細胞はそうでない場合にDes、Grb2、Setd7、Fbxo21、およびChd4で検出されたヘテロ接合SNP。(C)TS細胞独自の対立遺伝子を有するヘテロ接合/ホモ接合のSNP分布のジーンワイズ分析。
(本文続き)
FI-SC-specific SNPs were also examined where B6- and 129-derived sequences shared the same nucleotide but FI-SC-derived ones did not. Most of these SNPs matched with the CD1 background of the TSCs used in the experiment and were different from B6 or 129. Gene-by-gene representation (Fig. 4B) and whole-SNP analysis (Fig. 4C) showed that FI-SCs shared TSC-specific SNPs. As the proportion of contaminating TSCs in the FI-SCs was minor, most of the TSC-specific SNPs appeared heterozygous. These results support the view that the RNA-seq data contained transcripts from two major cell populations, with approximately 90% B6 cells having an ESC-like expression pattern and approximately 10% CD1-like cells having a TSC-like pattern. Therefore, the claim in the Obokata et al. paper that FI-SCs contribute to the placenta might have been based in error on contamination by TSCs, which are known to be organ stem cells (Tanaka et al. 1998).
B6と129由来の配列が同じヌクレオチドを持ち、かつFI幹細胞由来のものはそうでない場合のFI幹細胞独自のSNPが検討された。これらのSNPの大部分は実験で使われたTS細胞のCD1バックグラウンドと一致し、かつB6または129とは異なっていた、対応遺伝子ごとの表示(図4B)及び全SNP分析(図4C)はFI幹細胞がTS細胞独自のSNPを共有していることを示している。FI幹細胞の中に混入したTS細胞の割合が軽微だったので、TS細胞独自のSNPのほとんどがヘテロ接合として現れた。これらの結果は、RNA-seqデータが、ES細胞のような発現パターンを持つ約90%のB6細胞と、TS細胞のようなパターンを有する約10%のCD1のような細胞の、二つの主要な細胞集団からの転写物を含んでいるという見解を支持する。したがって、FI幹細胞が胎盤に貢献するという小保方ら論文の主張は臓器幹細胞であることが知られている(Tanaka et al. 1998)TS細胞の混入による間違いを根拠にしているかも知れない。
(英文)
Interpretation of the STAP phenomenon
Detection of aneuploidy using SNPs also suggested contamination in the original experiments. Both genotype and phenotype analyses suggested that the STAP cells used in the Obokata et al. experiments had trisomy of chromosome 8, and a transcription assay indicated atypical expression of genes on chromosome 13. Trisomy 8 is the most common chromosomal abnormality in mice, and chromosome 8 has been reported to fuse to the end of chromosome 13 (Kim et al. 2013). These observations might explain the results of virtual karyotyping in Fig. 3B. The RNA-seq data used in Fig. 3 were annotated as being derived from neonatal mouse spleen cells cultured in conditions under which the STAP cells did not grow. This description does not agree with the dominance of cells having trisomy because mice carrying pure trisomy 8 are embryonic lethal. This therefore leads to the conclusion that the cells were cultured cells that possessed expression characteristics that were very similar to those of ESCs.
STAP現象の解釈
SNPを使った異数性検出はまた元の実験に汚染のあることを示唆している。遺伝子型と表現型の両方の解析はObokataらの実験で使用されたSTAP細胞が8番染色体にトリソミーを有することを暗示し、転写因子検査は13番染色体上に遺伝子の異型の発現があることを示している。トリソミー8はマウスにおいて最も一般的な染色体異常であり、第8番染色体は13番染色体の末端に融合することが報告されている (Kim et al. 2013)。これらの観察は図3(b)の仮想染色体分析の結果を説明しているかもしれない。図3で使用されたRNA-seqデータは、STAP細胞が増殖しなかった条件下で培養された、新生児マウス脾臓細胞に由来するとして注釈されている。純粋なトリソミー8を有するマウスは胎生致死であるため、この記述は、トリソミーを有する細胞の優位性とは一致しない。したがって、これは、細胞がES細胞のものと非常に類似した発現特性を保有する培養細胞であったという結論に導く。
(英文)
Conclusion
The SNP allele frequency method described here is limited by the fact that if the contaminating and examined cells share a common genetic background, the allele frequencies will not be different enough to detect the contamination. However, this method is, in principle, applicable to any RNA-seq data that contain polymorphisms and will be useful for both prospective and retrospective quality control of RNA-seq experiments, especially for studies using cultured cells such as ESCs and iPS cells and their derivatives.
結論
ここで記述されたSNP対立遺伝子頻度法は汚染検査されている細胞が共通の遺伝的背景を共有している際には、対立遺伝子頻度が汚染を検出するのに十分なだけ異ならないかもしれないという事実によって制約されている。しかし、この方法は、原理的には、多型を含むどのRNA-seqのデータにも適用可能であり、また前向きであれ遡及的であれ両方の品質管理のために、特にES細胞やiPS細胞及びそれらの派生細胞などの培養細胞を用いた研究にとって、有用であろう。
(英文)
Experimental procedures
Dataset
Mouse variation data were obtained from the Sanger Mouse Genomes Project (*ttp://www.sanger.ac.uk/resources/mouse/genomes/), and the version 137 VCF-formatted dataset was retrieved from dbSNP (*ttp://www.ncbi.nlm.nih.gov/SNP/).
The locations of genes and included exons were obtained from iGenomes (*ttp://support.illumina.com/sequencing/sequencing_software/igenome.ilmn). SNPs outside exons were excluded from the original VCF file using this annotation, and thus, 1 016 227 SNPs were used for the entire dataset.
<実験手順>
データセット
マウスバリエーションデータはサンガーマウスゲノムプロジェクト (*ttp://www.sanger.ac.uk/resources/mouse/genomes/)から入手された。またバージョン137 VCF-フォーマットデータセットはdbSNP (*ttp://www.ncbi.nlm.nih.gov/SNP/)から検索された。
遺伝子および含まれているエキソンの位置はiGenomes(*ttp://support.illumina.com/sequencing/sequencing_software/igenome.ilmn)から入手された。 エキソンの外側にあるSNPはこのiGenomesの注釈を使用して元のVCFファイルから除外されている。従って1016227のSNPはデータセット全体のために使用されている。
(英文)
Raw sequencing data from the original RNA-seq experiments examined in this study were downloaded from the short read archive (SRA) at NCBI. The accession number of the project is SRP038104. Genome sequences of mouse (version 38, mm10) obtained from B6 mouse strain were downloaded from NCBI GenBank and encoded into a bowtie database using the bowtie-build (for colored space fastq files) or bowtie2-build (for fastq files) program. The accession numbers (i.e., SRA ID) of the RNA-seq experiments are listed in Table S1 (Supporting Information), and the checksums of archived sequences were confirmed by Dr Teruhiko Wakayama, Yamanashi University, one of the corresponding authors of the original paper.
この研究で調べられた元のRNA-seq実験からの生シーケンシングデータは、NCBIのショートリードアーカイブ(SRA)からダウンロードされた。 プロジェクトの受託番号はSRP038104である。 B6マウス株から得られたマウスのゲノム配列(バージョン38、mm10)をNCBI GenBankからダウンロードし、bowtie-build(着色スペースfastqファイル用)またはbowtie2-build(fastqファイル用)プログラムを使用してbowtieデータベースにコード化した。 RNA-seq実験の受託番号(すなわち、SRA ID)は表S1(支援情報)に列挙され、アーカイブされた配列のチェックサムは原著論文の責任著者の一人である山梨大学若山照彦博士によって確認されている。
(英文)
RNA-seq analysis
The SRA database sra-format files were converted into fastq format using sratoolkit.2.3.4-2. The Bowtie2 (version 2.1.0, bowtie-bio.sourceforge.net/bowtie2/index.shtml) and tophat2 (tophat.cbcb.umd.edu) programs were applied for sequence alignment. Because this study did not consider the structure of the mRNAs, all sequences were fragmented into 50-bp reads and aligned using tophat or tophat2 with the parameter “–no-coverage-search -G genes.gtf” allowing two mismatches. The tophat program was used only to analyze SOLiD colored space fastq files. Levels of gene expression were evaluated with fragments per kilobase of exon per million reads (FPKM) values calculated using cufflinks (version 2.1.1). A program written in C++ was developed to detect and enumerate SNP alleles in BAM files. The program is open-source software available in a public repository (github.com/takaho/snpexp/).
RNA-seq分析
SRAデータベースsra-形式ファイルはsratoolkit.2.3.4-2を使ってfastq形式に変換されている。 配列アライメントのためにBowtie2(バージョン2.1.0、bowtie-bio.sourceforge.net/bowtie2/index.shtml)とtophat2(tophat.cbcb.umd.edu)プログラムが適用されている。この研究はメッセンジャーRNAの構造を考慮していなかったので、すべての配列は、50 bpの読み取り断片に断片化され、2つのミスマッチを許容する “–no-coverage-search -G genes.gtf”パラメータを指定してトップハットまたはtophat2を使用して整列させた。トップハットプログラムは SOLiD colored space fastq filesを分析するためだけに使用された。遺伝子発現のレベルは、(バージョン2.1.1)のcufflinksを用いて算出されたfragments per kilobase of exon per million reads (FPKM) 値で評価されている。 C ++で書かれたプログラムはBAMファイルの中のSNP対立遺伝子を検出、列挙するために開発されています。プログラムは、公開リポジトリ(github.com/takaho/snpexp/)で入手可能なオープンソースソフトウェアです。
(英文)
SNP identification and heterozygosity tests
Sequence fragments aligned on the mouse genome were analyzed using the SNP detection and enumeration program mentioned above, and only SNPs with cover ratios ≥20 were retained. If ≥95% of the SNP sequence was the same on the two strands, the allele was designated as homozygous.
Genomewide heterozygosity between B6 and 129 was analyzed using a subset of the SNPs described above with different alleles between B6 and 129. SNPs were classified by known expression characteristics (TSC-specific, ESC-specific or other) and genotypes (B6-type homozygous, 129-type homozygous or other). SNP distributions were tested with P-values obtained using Fisher's exact test with Monte-Carlo Markov chain approximation.
SNPの識別およびヘテロ接合性のテスト
マウスゲノム上に整列した配列断片は、SNP検出および上記の計数プログラムを使って分析されている。かつ20以上のカバー率のSNPだけが残されている。 95%以上のSNPシーケンスが二本鎖上で同じだった場合、対立遺伝子はホモ接合と定義される。
B6と129との間の全ゲノムのヘテロ接合は、B6と129 との異なる対立遺伝子による上述したSNPサブセットを使用して分析されている。SNPは既知の発現特性(TS細胞特異的、ES細胞特異的、またはその他)と遺伝子型(B6型ホモ接合、129型ホモ接合またはその他)によって分類されている。SNP分布は、モンテカルロマルコフ連鎖近似に関するフィッシャーの確率検証を用いて得られたP値によって調べられている。
- 2019/05/14(火) 10:20:38|
- 遠藤論文
-
-
| コメント:0



