『AC129を巡る問題』(1) AC129とは何であったのかA.経緯AC129-1,2とFLS-1,2は129B6F1ES-1によって捏造されたと桂報告及びBCA報告は主張している。
BCA報告のその部分は以下です。(参考 *ttps://www.researchgate.net/publication/283439572_STAP_cells_are_derived_from_ES_cells)
>>
Control genomic DNA sequences for STAP cell chromatin immunoprecipitation sequencing (ChIP-seq) experiments (Fig. 4 in ref. 2) had been deposited in the NCBI database(2). To gain sufficient sequencing coverage, we re-sequenced the genomic DNA prepared from the STAP cell lysate used for ChIP-seq (Extended Data Fig. 1a). We confirmed that this STAP cell sample shared all the genomic characteristics described above for 129B6F1 ES1 (Extended Data Fig. 2c),indicating that the STAP cell sample used for ChIP-seq was derived from 129B6F1 ES1 cells.ここで(Fig. 4 in ref. 2)と指定されているのは、Letter Figure 4-bのことです。
その図のSTAPとESのデータが入れ替わっているというのが若山さんによるリトラクション理由書にあるのはよく知られている。序ですから引用しておきましょう。
>>
(4) In Fig. 4b of the Letter, STAP cell and ES cell are wrongly labelled in a reverse manner.Letter Figure 4-bは以下です。よくこの程度の粗雑な簡略図でSTAPとESの取り違えに気づけたものです。誰かラボ内に手伝った人でもいて、元データを持っていた人がいたことが推察されます。
BCA報告の(Extended Data Fig. 2c)の図は以下です。
上表のSTAP ChIP lysateに関してBCA報告本文に説明がありません。書かれているのはNCBIのデータベースと、自分たちの分析図です。
>>
Control genomic DNA sequences for STAP cell chromatin immunoprecipitation sequencing (ChIP-seq) experiments (Fig. 4 in ref. 2) had been deposited in the NCBI database(2). To gain sufficient sequencing coverage, we re-sequenced the genomic DNA prepared from the STAP cell lysate used for ChIP-seq (Extended Data Fig. 1a). NCBIのデータベースに関しては、すでに我々が以前分類しているもので以下の分です。
>>
ChIP-Seq
⑩SRR1171582 小保方 Low pH treated CD45 positive Cells:ChIPSeq.H3K27me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑪SRR1171583 小保方 Low pH treated CD45 positive Cells:ChIPSeq.H3K4me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑫SRR1171584 小保方 Low pH treated CD45 positive Cells:ChIPSeq.input Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロこれがLetter Figure 4-bのSTAPの欄で上の緑がH3K4me3で下の赤がH3K27me3になっています。若山さんの取り消し理由書に書かれているのはESの分とSTAPの分が逆に提示されているという主張です。このデータはTs.Marker氏が再分析していますが、素人では見方が分からなくて入れ替わっているのかどうか確認できません。そのことに関する彼の見解もありません。
他方、⑫に関してデータだけでなく、混合液であるSTAP ChIP lysateが残っていたのは別の話ですから由来を説明しないと分かりませんが、BCA報告の(Extended Data Fig. 2c)の図は全く同じものが桂報告書のスライド版にも使われていて、本文16Pに以下のようにあるが、どういう形で保管されていたのかは説明されていない。
>>
3)STAP 細胞由来 ChIP-seq (input)サンプルは 129B6 F1ES1 から取得された
(調査結果) 小保方氏が CDB ゲノム資源解析ユニット(以下「GRAS」という)に持参し残されてい た STAP 細胞由来 ChIP-seq (input)サンプルを再度 NGS 解析した結果、STAP 細胞由来と される ChIP-seq input データは CAG-GFP の挿入を持つ 129xB6 へテロ系統由来の細胞か ら取得されたものと判明した。さらに SNPs の解析、特異的な欠失変異の解析により(2 -3-1-1(d)参照のこと)CAG-GFP が挿入された 129B6 F1ES1 とほぼ同一細胞由来の データであることが明らかとなった。2年前の生データがSTAP細胞だけ冷凍されて残っているというのも変で、残っているならこの時の試料の全てのはずですが、記者会見では伊藤氏が口頭で記者の質問に答えて、この時の分析の試料がGRASにたまたま残っていたのだと証言しただけで、他の試料の有無に関しての更なる追及も無かった。又その保管に関して文書で追加説明があったということもない。伊藤さんはBCA報告の著者の一人です。
>>
Daijiro Konno, Takeya Kasukawa, Kosuke Hashimoto, Takehiko Itoh, Taeko Suetsugu, Ikuo Miura, Shigeharu Wakana, Piero Carninci & Fumio Matsuzaki桂会見での古田女史の質問に対する答えは以下でした。言い終わった後の彼のしぐさと表情は精神分析学の研究対象に好例かもしれない。(*ttps://www.youtube.com/watch?v=M9oJGioHvIQ 1:15:00から)
>>
ただ、唯一やったのが、あ、GRASの方に残っていたChIP seq のinput 、しかもSTAP細胞のインプットはDNAとして残っていたので、それが10ページバワーポイントの、えーと、ところに書いてある、3番のアイテマイズして書いてあるところですけれども、これに関しては、えーと、30倍になるだけ、他のゲノムと同量となるまで読んでいます。そして、読んだ結果、この、同一であるということを、ま、認定したということで、それ以外に、それ以外に関しては、ま、残っていませんのでやってない、やってません。なぜ、たくさんある中の、唯一、STAP ChIP lysateだけが残されていたのか、どういう形で唯一残されていたのか、誰かの恣意で残されていたのか、それともGRASには残し方に関する規則は無いのか。裁判だったらまず最初に検討されるもので、ここにちゃんとした答えが無いと証拠採用はされない。
B.報告書の非論理と重大な疑念a.非論理まず、先にBCA報告のこの件に関するあからさまな嘘を指摘しておきましょう。
>>
We confirmed that this STAP cell sample shared all the genomic characteristics described above for 129B6F1 ES1 (Extended Data Fig. 2c),indicating that the STAP cell sample used for ChIP-seq was derived from 129B6F1 ES1 cells.上に貼り付けた(Extended Data Fig. 2c)には
129B6F1 ES1は存在していません。あるのはES6です。ES6を図示していながら、これもどこから持ってきたかも正式に記述しないSTAP ChIP lysateが、図に無いES1と同じSNPsパターンであることを確認したと書いているわけです。
東の空を指さしながら美しい夕焼けですねと言ってるような論理です。言ってる事実が少なくとも指さしている方向には無いので、言ってる事自体も本当か嘘か分からない。科学者が書いたとはとても思えない
非論理な文章です。
b.最大の疑念以下に示すのは木星リストですが、まず129B6F1 ES1はそもそも小保方フリーザーには存在していないものです。右端の書き込みは調査のために管理部署から持ち出された記録です。129 B6 ES-4と5には持ち出された記録がないということにも注意が必要でしょうね。PCRに掛けられたデータがありますから持ち出したことは確かのはずですが、記録をつけないまま杜撰に持ち出して調べている実態がうかがわれる


では129B6F1 ES1はどこにあったのか。以下の若山さんの事後MTA契約書添付の持ち出しリストの中にある。下から二段目です。

よりによって若山さんは129B6F1GFP-1を持ち出していました。AC129-1,2もFLS-T 1,2も若山さんが持ち出していたControl ES cell の 129B6F1GFP-1 で捏造されているとBCA報告は証明しているわけです。ところが桂報告書もBCA報告書もAC129とFLS-Tは129B6F1ES1によって捏造されたと声高に主張しながら、その試料がどこにあったかを一切書いてない。小保方さんのフリーザーにあったものではないどころか
若山さんが山梨大に持ち出していたのです。桂報告書もBCA報告も、AC129の実験は小保方さんが若山さんに129B6F1GFP-1を渡して捏造させたものと示唆しているのですが、使われた細胞は理研には無くて、山梨大にあったのです。そして、報告書はこんな重要な事実を報告していないのです。これが分かったのは木星さんを中心にした大勢の人々の公開資料の取り寄せ努力によるもので、理研自らが発表したことではありません。むしろ
彼らはそれを隠していたことになるのです。これが疑念の最初で最大のものです。
c.その他の疑念次に、桂報告とBCA報告の遺伝子解析によるAC129と129B6F1ES-1との一致証明に関する疑義です。これは以下の表で、桂報告書とBCA報告のどちらにも掲載されています。BCAのは遺伝子異常のPCR結果もついていますからそれを貼り付けます。
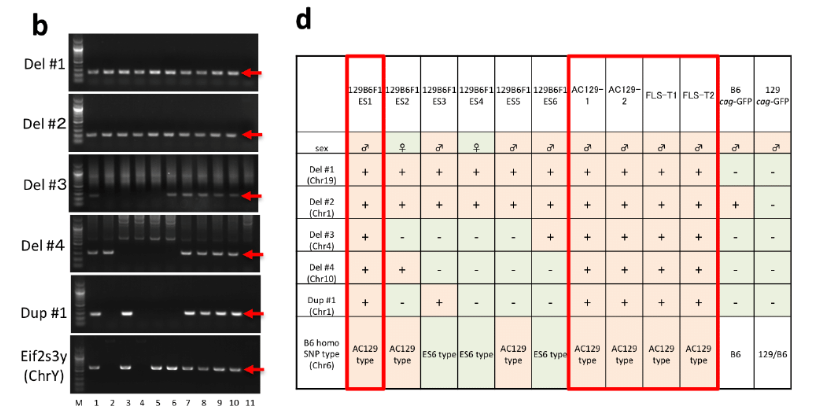
この表にある129B6F1 ES4と5には持ち出された記録がないということと、ES1が山梨の若山さんのフリーザーにあったということが書かれていないということは既に指摘した通りです。通常の事件ですと、こういう情報が揃うとまず最初に疑われなければならないのは小保方さんではなくて若山さんの方なんですけどね。先にこの所謂「僕のマウス」ESのサンプル保管の状況から検討しましょう。
(サンプルの状況)これに関しては既に見てきた通り二通りのラベル表記がある。若山さんの持ち出しリストに嘘か誠か6ライン樹立したと書かれています。嘘か誠かというのは、TSに関しては2ラインになってるが実際には129 B6 TS-3,4,5,6が理研側に残されていますからこれも6ラインのようです。嘘か勘違いかは問わず、今はESに関してのみ考えましょう。想定されるもともとあったラインは以下です。
①129 B6 ES-1,2,3,4,5,6
②129B6F1 GFP ES-1,2,3,4,5,6
ここで重要なのは今桂報告として知られている調査結果が世間に知られる以前の聞き取り調査段階で、小保方さんが①は若山さんが作ったと証言しているのに、離れた場所に入っていた②の6に関して「由来不明」と証言していることです。
つまり、小保方さんが知っているラベルの書き方は①であったということです。そして、②の1は若山さんが持っていて、②の6が誰かの置忘れか、意図的にそこに置かれたものだということになる。現状は以下です。
>>
ES細胞 129B6F1 GFP-1(若山持ち出しリスト)
129 B6 ES-2(小保方さん「若山TL樹立」証言)
129 B6 ES-3(小保方さん「若山TL樹立」証言)
129 B6 ES-4(小保方さん「若山TL樹立」証言)
129 B6 ES-5(小保方さん「若山TL樹立」証言)
129B6F1 GFP ES-6(小保方さん「由来不明」証言)
2013年の3月の引っ越し時に若山さんによるサンプルの整理が行われている。そして山梨大から放医研にサンプル調査に出したのは1年後の2014年の3月です。そして4月になってMTAの持ち出しリストに自分は現在1しか持ってないように書いた。
彼は記者会見で放医研に出した129B6F1 ESを5株と言っています。なぜ6株では無いのか。
そして、この5株は放医研側のPCR結果のデータには1,2,3,4,6と書かれている。5がなぜ提出されなかったのかの理由説明もない。
更にMTAの持ち出しリストには6株樹立されて、今自分が持っているのは1だけのように記載されている。放医研には株分けして出したのであれば少なくとも1,2,3,4,6が持ち出しリスト上に無ければならない。仮に株分けせず手持ちのものをそのまま出したのなら、放医研に出したものは記載しないのなら何もないことになる。1だけがあるという理由は1だけは株分け分があったということになるしかない。
ところがこのESは理研側でも調査されていているからにはそのときには全ラインあったということになる。理研側の調査したサンプルの由来はどうなっているのか。
小保方さんのフリーザーに残されていたものとその松崎氏の持ち出し状況は以下です。
>>
129 B6 ES-2 (2014/6/20 松崎氏持ち出し記録あり)
129 B6 ES-3 (2014/6/20 松崎氏持ち出し記録あり)
129 B6 ES-4
129 B6 ES-5
129B6F1GFP ES-6(2014/6/9 松崎氏持ち出し記録あり)
若山さんの記者会見スライドにあった放医研の記載の仕方は以下でした。ラベルは全部新たに書き換えられて提出されていることが分かります。
>>
129B6F1-1 ES
129B6F1-2 ES
129B6F1-3 ES
129B6F1-4 ES
129B6F1-6 ES
放医研のサンプルを回して貰ったのなら5がありませんが、どこから持ってきたのでしょうかね。129 B6 ES-5 は少なくとも持ち出し管理記録上は持ち出されていないことになっている。調査が如何に杜撰で恣意的かということです。
ただし、1~6は欠失重複等が全部違っていますから理研側が6種類揃えたことは間違いありません。何かの中身が重複しているということはありません。調査細胞の入手経緯が明らかになっていないだけです。
我々の推測では[129B6F1 GFP ES-6(小保方さん「由来不明」証言)]を小保方さんのボックスに置いたのは若山さんだと推測しています。若山さんは1を置いておいても6を置いておいても大して違わないと思っていた。というのも遺伝子異常があって全ライン識別されるとは思ってもいなかったでしょうし、雌雄の違いも考えている暇がなかったんでしょうね。1も6もたまたま雄でしたが、違ってたら言い訳のつかないことになってたでしょう。核型解析に出せばすぐ分かりますから事前に雌雄確認をしていたかもしれません。ただ、遺伝子異常まではさしもの若山さんも気づかなかったでしょう。ES-1は無論、直前にFLS-T1,T2を作っていた時に使った株分け分が手元に残ったのではないか。これが致命傷になった。これ見よがしに置かれていたES-6は使われていなくて、若山さんが山梨に持ち出していたES-1が使われていたという藪蛇になってしまった。
問題は理研側に残されていた分で129 B6 ES-1と129 B6 ES-6がなぜ無いのかということです。このコントロールESは小保方さんは論文に使っていますから、実際に使用している。別名でどこかにあるかもしれません。後に詳しく調べてみましょう。
(SNPs解析)SNPsでAC129タイプと書かれているのはES1タイプということです。ES1タイプのマウスとES6タイプの2タイプの卵が使われているということです。なぜこういうことになるのかを考えてみる。
SNPは一塩基性多型と呼ばれている突然変異で人間でもそれぞれの人の顔が違うというような表現型の多様性等を作っているものですが、無論人間であることは共通ですから大半の遺伝子は共通です。
一方近交系マウスは20世代以上の兄妹交配を重ねて相同染色体の同一遺伝子座の遺伝子をホモにそろえてありますからSNPsもホモです。近交系マウスはマウス業者(ラボ)が作って系統立てて管理しているわけですが、例えば129/SvJ独特のSNPs分布というのは最初にジャクソン研究所で作ったマウスのSNPsを引き継いでいるものです。B6のラインを作るときは又別のマウスを使いますからこれは亜種が違うので当然全然違う分布になる。しかし、129でもB6でも最初に使われたマウスの個性が引き継がれるので仮に別の人が一から作ると別のSNPs分布になる。それでは研究目的に使えないので、同じ名前にはなりません。ですから例えば129/SvJと言ったら最初にジャクソン研究所の作ったマウスと決まっていてそれが国際的にも引き継がれていて、SNPs分布も決まっている。

継代繁殖で新たにできるSNPsというのはわずかなので、長く蓄積すると更に別の表記を加えますが、直接的には関係ないんです。
で、SNPsパターンでES1タイプとES6タイプの二つあるというのは、雑種交配させたんですから、129とB6の混ざり方の違う2個の受精卵が使われているということになる。完全な近交系マウスでは減数分裂時にできてくる卵も精子もそれぞれに遺伝子配列は全く同じですからこんなことになることはない。従って一方が完全な近交系マウスになってないということです。その原因は所謂「僕のマウス」作成の経緯にある。
若山さんはロックフェラー大学にいるときにB6のCAGホモマウスを作った。日本に戻ってからそれを129と掛け合わせてその後戻し交配を続けてGFPだけを129に移し替えた。129もB6も無論近交系で、マウス生産業者が20世代以上の兄妹交配を続けて対立遺伝子をホモにそろえて販売しているものです。契約があって原則自家繁殖させてはいけない。特に人にあげてはいけないです。マウス業者がその純度を保証できないから予期せぬところでの責任を負いきれないというのが理由です。
最初にB6の卵にウイルスベクターを使ってCAG-GFPを挿入する。どこに入ったかは分からないが、入ったら生まれたマウスは蛍光する。この蛍光している一匹のマウスに又B6を掛け合わせてたくさん子供を作る。この時の子マウスのCAG-GFPが光っている雄雌だけをメイティングする。GFPヘテロ同士の掛け合わせになります。ヘテロ同士を掛け合わせると第3世代で1/4の確率でホモができる。ランダムに交配させて十分な子マウスを作って一匹もGFP無しの子マウスが生まれてこなかったペアはホモ同士という確認で継代を重ねることを省略できる。もともとが近交系マウスですからGFP以外は全部ホモです。そういうことをしないで念を入れて最初から20世代継代してもホモになりますね。(放医研の検査で18番にホモに入っていました。)
それに対して129に移し替えるというのはよりテクニカルです。ただ単に129のCAGマウスを作るだけならB6と同じやり方でできる。でもこれだと又GFPがどこに入るか分からない。GFPが染色体の同じ場所にホモで入っているB6と129が作りたいということです。
それでまず一匹のB6-CAGホモマウスと一匹の普通の129とを交配させた。これは最初129B6F1です。B6がGFPホモですから子供には全部GFPがヘテロに来る。ただし、雑種にしましたから他の遺伝子もヘテロに変わってしまっています。
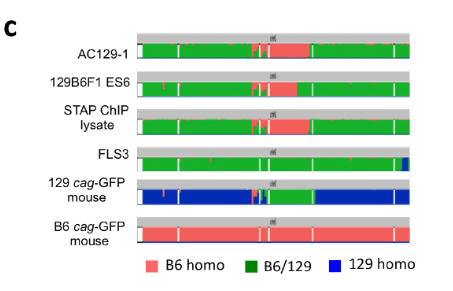
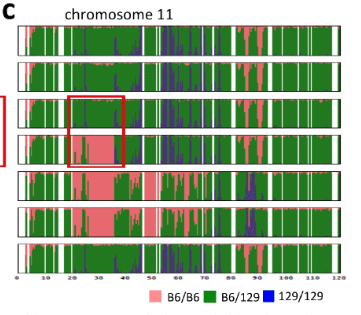
そこで129への戻し交配を行って、GFP遺伝子以外を親の129の近交系に戻す。そのやり方はGFP蛍光のある子供に対してのみ戻し交配を行うんです。これを繰り返すとGFPのみヘテロの129近交系マウスに戻ります。このヘテロ同士を掛け合わせて129GFPホモを作るんですが、この戻し交配が完全に行われていないとGFP以外の他の遺伝子にもB6の遺伝子が残ってしまう。それが上に貼り付けているBCA報告のExtended Data Figure 2-cのSNPs分析です。再掲しておきましょう。

上図のリジェンドです。
>>
c, Distribution of B6-type and 129-type SNPs along chromosome 6. The B6-homozygous SNP cluster(magenta) in the middle, which probably arose from the inheritance of the parental 129, is heterogeneous in length among six control ES cell lines. The four cag-GFP STAP stem-cell lines share the same length of the B6 SNP cluster with control ES 129B6F1 ES1. Note that the 129/B6-heterozygous SNP region in the 129 cag-GFP mouse is longer than that of AC129-1.下二つが今説明した親マウスの6番染色体のSNPs分布図です。B6は全部ピンク(magenta) です。これはロックフェラー大学で作ったGFPマウスです。GFPを挿入しただけですから対立遺伝子は全く同じで全部ホモにそろっていますから問題ない。対して129の方は全部ブルーになってないといけませんが、ピンクの部分とグリーンの部分が残ってます。ピンクの部分は相同染色体の両方がB6で、グリーンは一方がB6で他方が129ということです。戻し交配の回数が不足している状態でGFPヘテロ同士を掛け合わせてホモにしてしまった結果かもしれません。因みにGFPホモであるかどうかは更にもう一代子供を作って、全部蛍光しているかどうかで確認できます。
問題は129です。近交系マウスに戻っていない。厳密にいうとGFPだけでなく、129/Svだということすらできない状態です。もう少し長くバッククロスを続けてから兄妹交配させないといけなかったのかもしれない。でも、調べない限り本人は気づいていませんからやり直すことはできない。できたものの兄妹交配を続けるだけです。しかし、これはこれなりに20世代交配させていたら近交系マウスになります。
マウスは20日で生まれて更に妊娠可能になるまで50日かかります。70日の20継代は1400日です。3.8年です。若山さんは日本に帰ってきてから129へのGFP移し替えを行いましたから、戻し交配でも3.8年かかっている。しかし、その後の20継代維持を入れると計7.6年ですが、理研の10年目だったわけですから、ちゃんと継代できていたら、全部ホモにそろっていたはずです。にもかかわらずヘテロ領域がこんなにあるというのはもっと近い時期にB6とのコンタミがあったということになるのではないでしょうか。又そもそもの129には歴史的にジャクソン研究所で一度B6が混入したという事件がありました。従ってピンク部分はもともと存在していたものかもしれません。
そもそも論としてこういう図形がどうしてできるかということですが、ひとつづつの対立遺伝子のあり方はホモかヘテロしかありません。大多数のDNAはマウスである限り共通ですから、129かB6のどちらということはできません。言えるのはSNPのある個所だけです。
このケースの場合で、6番染色体のDNAの並びをある一定区間で区切って1単位とします。この長さは適当に人が決める。この区間にSNPsが何もなければ白抜きで表示する。遺伝子のあるところは大事なところなんでSNPsは少ない。そういう場所は白抜きになる。存在しているSNPsがB6特異的な変異だったらピンクで表示する。129特異的な変異だったらブルーで表示する。半々だったらグリーンです。F1ですから父と母とから来た両方があるのが普通です。だから大半グリーンになる。
では混じっているが半々でないときはどうするか。半々の部分とB6が勝ってる部分、129が勝っている部分と1単位の中で棒グラフとして表示している。
1単位の区間を細かくすると例えば以下のBCA報告のExtended Data Figure 1-c図のようになるし、区間を広くとると上のExtended Data Fig. 2cのようなものになるわけです。
ともあれ、母親の129の染色体の一部にヘテロ部分があったわけですから卵原細胞が減数分裂するとき別々のSNPs分布を持つ二種類の卵ができる。マウスは多産ですのでたくさんの受精卵ができて、胚盤胞にまで成長させてから中のインナーセルマスを取り出してES細胞を培養する。8株できたわけです。それがES1型とES6型に分かれたのはそういう理由です。雌雄は以下です。
>>
ES1タイプ→ES1(オス)、ES2(メス)、ES5(オス)
ES6タイプ→ES3(オス)、ES4(メス)、ES6(オス)
この解析結果の不可思議はES6以外のSNPs解析データが図示されていないことです。このことは後に述べます。
(関連細胞)Letter Figure 4-bで使われている各種細胞は以下です。
>>
①CD45
②STAP
③ES
④STAP-SC
⑤FI-SC
⑥TS
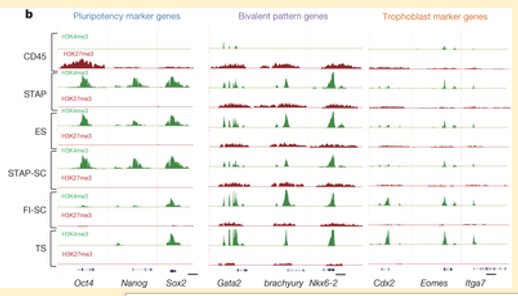
Letter Figure 4-bそのものはこれです。再掲します。

この図に対応しているのが公共データベースに登録されている以下のデータです。
>>
ChIP-Seq
①SRR1171553 小保方 CD45 positive Cells:ChIPSeq.H3K27me3 derived from spleen Oct3/4::gfp C57BL/6 GOF
②SRR1171554 小保方 CD45 positive Cells:ChIPSeq.H3K4me3 derived from spleen Oct3/4::gfp C57BL/6 GOF
③SRR1171555 小保方 CD45 positive Cells:ChIPSeq.input derived from spleen Oct3/4::gfp C57BL/6 GOF
④SRR1171562 若山 Embryonic Stem Cells:ChIPSeq.H3K27me3 C57BL/6x129/Sv
⑤SRR1171563 若山 Embryonic Stem Cells:ChIPSeq.H3K4me3 C57BL/6x129/Sv
⑥SRR1171564 若山 Embryonic Stem Cells:ChIPSeq.input C57BL/6x129/Sv
⑦SRR1171567 若山 FI-SC(Fgf Induced Stem Cells):ChIPSeq.H3K27me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-Acr-CAG
⑧SRR1171568 若山 FI-SC(Fgf Induced Stem Cells):ChIPSeq.H3K4me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-Acr-CAG
⑨SRR1171569 若山 FI-SC(Fgf Induced Stem Cells):ChIPSeq.input Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-Acr-CAG
⑩SRR1171582 小保方 Low pH treated CD45 positive Cells:ChIPSeq.H3K27me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAG
⑪SRR1171583 小保方 Low pH treated CD45 positive Cells:ChIPSeq.H3K4me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAG
⑫SRR1171584 小保方 Low pH treated CD45 positive Cells:ChIPSeq.input Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAG
⑬SRR1171587 若山 STAP-SC(STAP derived Stem Cells):ChIPSeq.H3K27me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAG
⑭SRR1171588 若山 STAP-SC(STAP derived Stem Cells):ChIPSeq.H3K4me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAG
⑮SRR1171589 若山 STAP-SC(STAP derived Stem Cells):ChIPSeq.input Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAG
⑯SRR1171592 丹羽研 Trophoblast Stem Cells:ChIPSeq.H3K27me3 CD1
⑰SRR1171593 丹羽研 Trophoblast Stem Cells:ChIPSeq.H3K4me3 CD1
⑱SRR1171594 丹羽研 Trophoblast Stem Cells:ChIPSeq.input CD1最初に注意しておかなければなりませんが、これらの試料がGRASに提出されたのは2012年の8月です。従って細胞の製作者は基本小保方さんと若山さんです。8月の解析は笹井さん参加前の実験結果で、しかも幹細胞化実験ですから、3誌論文の中には取り入れられてないものです。手記によると8月になってから若山さんが幹細胞化実験の成果を自分に51%割合で特許申請手続きを開始したとされている。そこから米国側と揉めて11月には小保方さんが米国に引き上げようとした経緯につながっている。
笹井さんが引き継いでからNGS解析をつけようということになってこの8月のデータが検討された。ChIP seqのデータもこのときにLetter Figure 4-bに纏められた。
若山さんは多忙で手伝えないということですから、新たに幹細胞やキメラを作ることはできない。公開データは笹井さん参加後にレター論文に使われたものです。若山さんは手伝っていませんから、若山さんが作ったものは2012年8月以前のものです。ES細胞とFI幹細胞は若山さんしか作れない。STAP幹細胞は小保方さんがSTAP細胞を作って若山さんがそれを使って作成した。CD45細胞とSTAP細胞は小保方さんはマウスさえあれば何時でも作れる。TS細胞は若山さんしか作れませんが、若山さんはF1で作ったと言ってますから、CD1マウス背景のTSは丹羽研のものと分かる。このことは若山さんの作ったTSが分化しているようだったので丹羽さんが提供したと桂報告書にも書かれています。桂報告書26P。
>>
TSについては細胞培養時に分化し たことが考えられたことから、丹羽研のメンバーが培養・サンプル調製を行ったものを 追加して作図に用いた。⑯⑰⑱は丹羽研のものと分かっているわけです。論文の論旨としては比較のためのコントロールですから誰のTSでもいい。2012年8月に提出されていたもともとの細胞はF1背景のTS細胞だった。
①②③は白血球ですが、GOFだと書かれている。これも論文ではGOFとF1が使われているわけですが、どちらでもいい。ただし、2012年8月のGRAS提出目的はレター論文用ではありません。笹井さんはまだ参加してない時期です。特許申請手続きを始めていますから、若山さん主導の幹細胞論文や特許申請書に添付するデータとして使う目的です。この時にGOFであったかどうかは分かりません。もしかしたら他と同じF1の分であったかもしれない。
その他は2012年8月のときのままです。酸浴細胞は小保方さんは何時でも作れますが、若山研のF1マウスは笹井研時代には使えませんから当時のままだと分かる。そしてすべての試料はF1背景です。ただし、理研が2012/2/13にNCBIに提出したときには今見られるとおりのC57BL/6x129/Svになっていた。しかし、小保方さんが理研に提出した2012/11/3の時点でどう書かれていたかは分かっていません。ここには若山さんの作ったES細胞があってそのマウス背景は129/SvxC57BL/6だということは分かっていますから、この登録の少なくともこの分は間違っている。ただ、それを小保方さんが間違えたのかは分かりません。
小保方さんはNCBIに直接登録はしていない。行ったのは理研の別人です。ここではF1であるという事にとどめておきます。
④⑤⑥がそのES細胞です。
⑦⑧⑨はFI幹細胞ですが、後の調査でアクロシンが出た。桂報告では太田ESだということになっていますが、若山さんは細胞の大きさで気づかないということはあり得ないと証明済ですので、これは岡部マウスとのF1由来の小保方細胞核使用ntESをTS培地誘導したものと分かる。
⑩⑪⑫がSTAP細胞ですが、これはアクロシンが出ていない。しかもCAG-GFPです。129でもB6でもCAG-GFPの入っているマウスは「僕のマウス」の親しか知られていない。小保方さんは見かけでは親の雌雄とGFPがホモかヘテロかはわかりません。
⑬⑭⑮が上の⑩⑪⑫をSTAP幹細胞誘導したものということになるが、これも後の調査でCAG-GFPと判明している。我々の判断ではこれもntES化しているということになるが、胎盤が光ると言われたキメラは小保方さんはB6/129としていますから、その時にSTAP幹細胞が作られていると知らされているのかもしれない。いずれにせよ。FLSは「僕のマウス」を渡したが中身は太田ESだったとされているが、
ここに公共データ登録では背景がB6x129と記載されている細胞で、事実中身が「僕のマウス」から作られているSTAP幹細胞と称する細胞があるということは驚きで、このことに誰も気づいていません。もしこれが「僕のマウス」ESの1~6のどれかなのなら、今でもデータはあるのですから調べたらわかります。しかし、SNPs解析も遺伝子異常解析もされていません。ただ、マウス背景が129B6F1のCAGホモだと桂報告が言ってるだけです。
Letter Figure 4-bは、ES細胞に代表される多能性遺伝子(Pluripotency marker genes)であるOct4,Nanog,Sox2、二価クロマチン(Bivatent pattern genes)であるGata2,brachyury,Nkx6-2、そしてTS細胞特異的遺伝子(Trophoblast maker genes)であるCdx2,Enomes Itgα7に関して、それぞれの、その巻き取られているヒストン領域がH3K4me3(H3ヒストンのK4サイトのリジンの3つあるアミノ基が全部メチル基に置き換わっているトリメチル化と呼ばれる現象を起こしている)とH3K27me3の状態にあるDNAをクロマチン免疫沈降(Chip=chromatin immunoprecipitation)で抽出した後に定量PCRに掛けた結果である。
(H3K27me3)は常時転写抑制として働き、(H3K4me3)は活性化を許可する働きを持つ。二価クロマチンは間に立ってその両方をコントロールする。従って例えばOct4遺伝子のCD45を見ると緑の(H3K4me3)状態にあるものはなく、赤の(H3K27me3)状態にあるDNA断片がたくさんあることを示している。その下のSTAP細胞を見ると赤はなく緑がたくさんある。つまりOct4遺伝子のプロモーター領域が活性化されているので結果的にはOct4遺伝子を発現させてOct4蛋白質をたくさん産出しているということになる。この状態でOct4-GFPが組み込まれていると細胞は蛍光することになる。
ChIP seqに限らず、この遺伝子解析結果を重視する人々、Ts.Markerさん、DORAさん、学さん、和モガさん、そしてOoboeさんとそのパートナーさんが"STAP有る派"となっていることは衆知の事です。そして彼らは同時にキメラはナイフ切り分けであれ、スタンダードなやり方で論文通りに出来ていると考えている。若山さん本人が太田ESのコンタミだと言って明らかな嘘をついて論文を否定した理由は何らかの陰謀であると考えておられるらしいが、それがどういうものかに関して何ら推論も論証も無い。
対して小保方細胞核使用ntES論は説明した通りである。
1.キメラはntESでできている。
2.幹細胞もntESでできている。
3.STAP酸浴細胞は小保方さんが作った本物である。
丹羽論文はティシュー論文の一部を再現したが、理研での酸浴Oct4-GFP蛍光細胞が本物のOct4遺伝子をほんのわずかしか含まず、GFP蛍な光の漏れ出し現象を指摘した。それに対して、このLetter Figure 4-bの実験結果はたくさんの内在性Oct4遺伝子の発現を定量的に立証している。つまり小保方さんの再現実験があまりに過酷で十分な再現条件でなかった可能性を示している。
以下は彼女の再現実験の予備実験時の写真です。旧DORAブログにあるものですが、近々閉鎖になりますから、あちこちに証拠保存しておくという意味でも貼り付けて置きましょう。参考までに、後にOoboe さんのパートナーさんの取り寄せた、赤色フィルター切り替えで自家蛍光の無いことの確認されている、全てが、緑色スフィア塊の写真も西岡さんのガンバレブログに楠本さんが貼り付けています。
しかし、だからと言って、キメラができたことにはならない。
さて、一方で、レター論文にNBCI登録されていると書かれているのは以下の分ですが、上述したChIP seq分、或はここにも書かれているChIP-seq files はここに挙げられているリストにはない。
>>
RNA-seq and ChIP-seq files have been submitted to the NCBI BioSample databases under accessions SAMN02393426, SAMN02393427, SAMN02393428, SAMN02393429, SAMN02393430, SAMN02393431, SAMN02393432, SAMN02393433, SAMN02393434 and SAMN02393435.このリストに対応している登録サンプルは以下です。
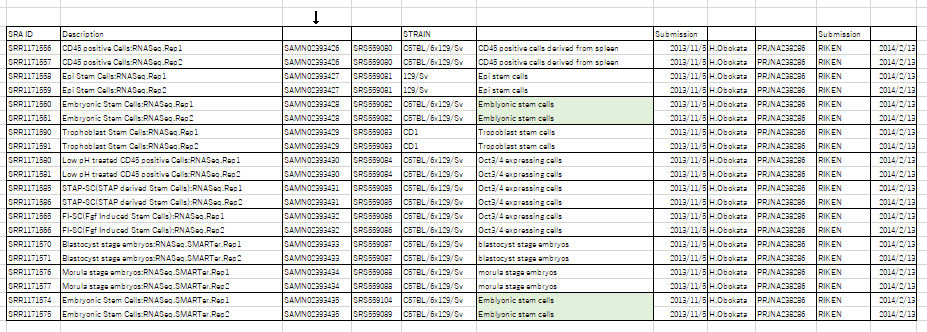
こちらのRNA分析にはES細胞が二つ登録されています。恐らく若山さんの作った分と、レター論文で使われたBFPのES細胞ではないかと思われます。これは丹羽さんが作っているのではないか。何にも確認されていません。ほんとに杜撰な調査です。すべて小保方さんや丹羽さんに聞いたら分かる事ばかりです。
報告書は二年前の2012年8月にGRASに提出されたLetter Figure 4-bのSTAPのグラフのもととなった上記⑫の生資料が保存されていたと説明しました。桂報告書16Pです。
>>
3)STAP 細胞由来 ChIP-seq (input)サンプルは 129B6 F1ES1 から取得された
(調査結果) 小保方氏が CDB ゲノム資源解析ユニット(以下「GRAS」という)に持参し残されてい た STAP 細胞由来 ChIP-seq (input)サンプルを再度 NGS 解析した結果、STAP 細胞由来と される ChIP-seq input データは CAG-GFP の挿入を持つ 129xB6 へテロ系統由来の細胞か ら取得されたものと判明した。さらに SNPs の解析、特異的な欠失変異の解析により(2 -3-1-1(d)参照のこと)CAG-GFP が挿入された 129B6 F1ES1 とほぼ同一細胞由来の データであることが明らかとなった。 ⑫があるにも関わらず、調査チームは保存されていたinputデータの混合液をもう一度解析したというのです。公共データ登録されているデータとどう違うかの説明はありません。inputデータは提出された細胞のDNA、RNAの全てを含んだ混合液のシーケンスデータです。その混合液自体が残されていて、これを他のゲノムと同量となる30倍になるまで読んだという伊藤さんの説明ですが、ど素人なのでその意味は分からない。
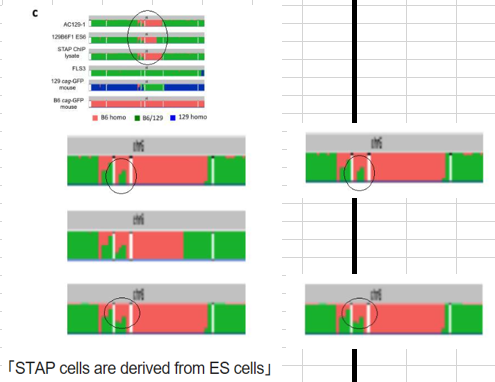
このSNPsの解析表がBCA報告の(Extended Data Fig. 2c)ですが、129B6F1 ES1が無いということは既に指摘しています。129B6F1 ES6とSTAP ChIP lysateはど素人が見ても全くパターンが違います。一方、AC129-1は129B6F1 ES1と同じだと言われているんですが、そのAC129-1とSTAP ChIP lysateとの比較はどうかというと、これはとても初期段階でTs.Marker さんが問題提起しました。
Ts.Marker さんは後にグラフィックスの精度の問題の範囲内かもしれないとおっしゃってますが、本日(2019/8/14)学ブログに以下の書き込みがありました。以前から指摘されていることですが、1Mbの範囲内で半分ほど違ってると改めて指摘されると、確かにこれは大きいですね。今のところSTAP ChIP lysate≠AC129-1(≒?129B6F1 ES1)ということではないでしょうか。
>>
拡張データ2CのSNPパターンの図ですが、縦の1枠(白抜きになっている部分)が1Mb(塩基配列100万個)です。縦枠150個並べて第6染色体の150Mbの全塩基を表しています。ChIP-seqとAC129は67Mb~68Mbの1Mbの範囲に違いがあり、AC129は全てB6ホモだけど、ChIP-seqは約500kbがB6ホモ、残りが129/B6ヘテロになっています。FLSの欠失は8番染色体で17kb、3番染色体の欠失は5kbで同じ欠失があることでFES1他と同一認定されましたが、違いが500Kbもあるというのは大きな違いです。細胞株が違うことを証明するのは、違いを1つ示せばいいので、これで細胞株が違うことは証明できています。
2019/8/14(水) 午前 9:56[ カツラ報告書 ]返信する黒線のところが1Mb幅で半分のSNPs数の違っているところです。
最初に指摘して置いたように、若山さんはややこしいことに取り下げ理由書にLetter Figure 4-bのSTAP細胞とES細胞のデータが取り違えられていると書いています。再掲します。
>>
(4) In Fig. 4b of the Letter, STAP cell and ES cell are wrongly labelled in a reverse manner.この取り違えは作表時なのか、サンプル提出時なのかわかりません。STAP ChIP lysateが提出時のES細胞の取り違えだったのなら、ここから129B6F1ESらしきものが出ても変ではありません。
ではその際は間違えられた方のESがSTAPだということになるが、そのマウス背景は何なのか。データ登録されたマウス背景はCD45がGOFで、TSがCD1、そしてその他は全部B6 X 129です。そしてそれを確かめるためのinputデータは揃っているが、検証はされていない。凍結lysateはSTAPだけだったのかどうかもわかってない。つまり、何事も系統立てては調べられていない。調査はとても恣意的で場当たり的だと言わざるを得ない。
- 2019/08/27(火) 09:34:47|
- AC129
-
-
| コメント:0
















