まずはデータですね。
河本論文です。(*ttps://onlinelibrary.wiley.com/doi/full/10.1002/eji.200323461 Extensive proliferation of T cell lineage‐restricted progenitors in the thymus: an essential process for clonal expression of diverse T cell receptor β chains)
Figure3
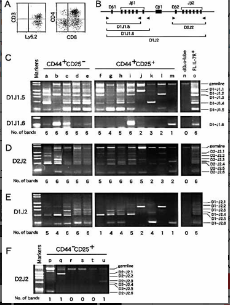
Figure4
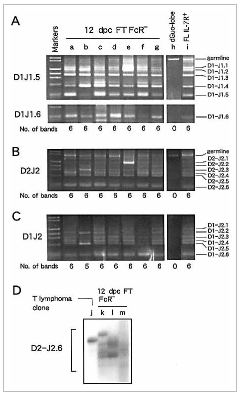
Figure3-B(拡大参考)
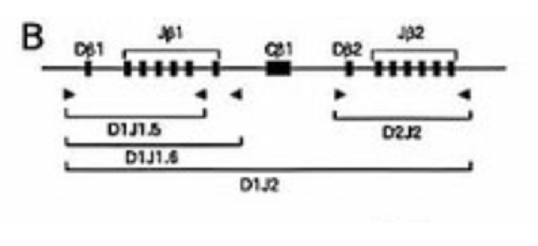
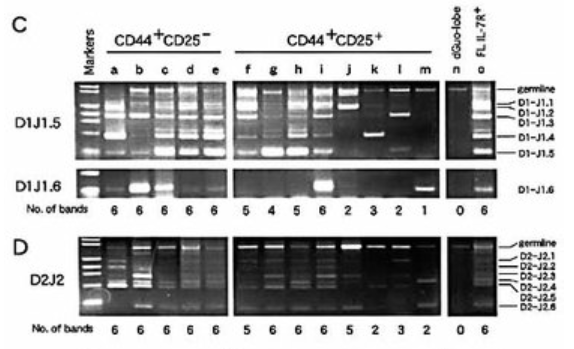
Figure3-C,D(拡大参考)
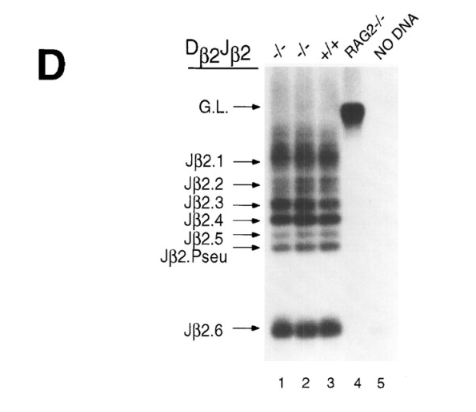
Charles E Whitehurst論文Figure3-D(*ttps://www.cell.com/immunity/fulltext/S1074-7613(00)80031-X# Control of V(D)J Recombinational Accessibility of the Dβ1 Gene Segment at the TCRβ Locus by a Germline Promoter)
- 2019/07/18(木) 08:50:23|
- STAP事件
-
-
| コメント:33
<<
胎盤に関する考察38 |
ホーム |
胎盤に関する考察36>>
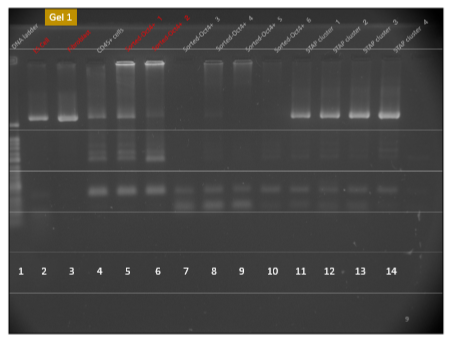
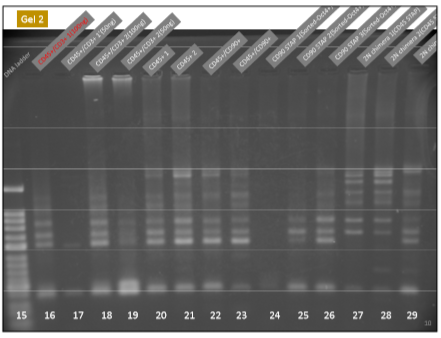
はたしてGel1,2は河本論文にあるバンドの出方から大雑把にでもGLも含めた8本のリアレンジメントバンドのどれがどれと特定できるかという問題から確認していきましょうかね。
- 2019/07/18(木) 16:38:54 |
- URL |
- #-
- [ 編集 ]
河本論文というのは以下の論文で、ここに書かれているプライマーがSTAP論文のプライマーと同じであることからアルイミオウジ変態糞ジジイが、小保方さんたちが参考にした論文だと主張しているものだね。
>>
Extensive proliferation of T cell lineage‐restricted progenitors in the thymus: an essential process for clonal expression of diverse T cell receptor β chains(*ttps://onlinelibrary.wiley.com/doi/full/10.1002/eji.200323461)
Hiroshi Kawamoto
Koichiro Ohmura
Shinji Fujimoto
Min Lu
Tomokatsu Ikawa
Yoshimoto Katsura
- 2019/07/18(木) 16:40:16 |
- URL |
- #-
- [ 編集 ]
この中のFigure3と4で使われているプライマーがマテメソに以下のように書かれている。
>>
"4.5 PCR analysis of D–J rearrangement in TCRβ chain gene
Cells (4×103 cells) were suspended in 20 μl of 1× PCR buffer (10 mM Tris‐HCl, pH 9.0, 50 mM KCl and 1.5 mM MgCl2) including 0.45% NP40, 0.45% Tween 20 and 1.2 μg proteinase K (Sigma, St. Louis, MO), and incubated at 55°C for 1 h, then 95°C for 10 min. Samples of these disrupted cells were used as templates for PCR amplification. Primers were: Dβ1, 5′‐TTATCTGGTGGTTTCTTCCAGC‐3′; Dβ2, 5′‐GCACCTGTGGGGAAGAAACT‐3′; Jβ1.5, 5′‐CAGAGTTCCATTTCAGAACCTAGC‐3′; Jβ1.6, 5′‐GGTAGAAAGGTAGAGGGTTCCAGA‐3′; Jβ2.6, 5′‐TGAGAGCTGTCTCCTACTATCGATT‐3′. The reaction volume was 20 μl containing 5 μl of the cell extract (equivalent to 1,000 cells), 1.5 μl of 10× PCR buffer, 0.16 μl of 25 mM dNTPs, 0.4 μl of each primer (10 mM), and 0.6 U Taq polymerase. Thermocycling conditions were as follows: 5 min at 94°C followed by 35 cycles of 1 min at 94°C, 1 min at 60°C and 2 min at 72°C and 10 min at 72°C. Amplified DNA products were loaded on a 1.2% agarose gel, electrophoresed, and stained with ethidium bromide. In the experiments in Fig. 4D, 32P‐labeled dCTP was added to the PCR, and PCR products were loaded on a 6% denaturing polyacrylamide gel. "
- 2019/07/18(木) 16:41:27 |
- URL |
- #-
- [ 編集 ]
Figure3-Bにプライマーの位置が図解されているね。全部で5か所だ。
>>
①Dβ1 5′‐TTATCTGGTGGTTTCTTCCAGC‐3′ (22bp)
②Dβ2 5′‐GCACCTGTGGGGAAGAAACT‐3′(20bp)
③Jβ1.5 5′‐CAGAGTTCCATTTCAGAACCTAGC‐3′(24bp)
④Jβ1.6 5′‐GGTAGAAAGGTAGAGGGTTCCAGA‐3′(24bp)
⑤Jβ2.6 5′‐TGAGAGCTGTCTCCTACTATCGATT‐3′(25bp)
- 2019/07/18(木) 16:42:14 |
- URL |
- #-
- [ 編集 ]
これらのプライマーで挟まれる区域は以下ね。
>>
(1)Dβ1-Jβ1.5
(2)Dβ1-Jβ1.6
(3)Dβ1-Jβ2.6
(4)Dβ2-Jβ2.6
- 2019/07/18(木) 16:42:59 |
- URL |
- #-
- [ 編集 ]
(1)はどうして必要なのかな。(2)があれば十分じゃないか。
- 2019/07/18(木) 16:44:19 |
- URL |
- #-
- [ 編集 ]
小保方さんのように検体にTCR再構成があるかないかを調べるだけなら(1)と(2)を分ける必要はないけど、彼らが調べようとしているのはそんな単純なことではないからな。
- 2019/07/18(木) 16:45:12 |
- URL |
- #-
- [ 編集 ]
まずこの論文は小保方さんの調べたような所謂成熟T細胞のTCR再構成の有無を調べているんじゃない。もっと専門分野の話なので、胎児胸腺の中でT 細胞が作られていく過程で存在している謎を解明しようとしている。分析対象が違っていてかつ、PCRの目的も違ってる。
- 2019/07/18(木) 16:46:06 |
- URL |
- #-
- [ 編集 ]
でも、TCRのジャームラインはそれこそ生まれた時から受け継いでいるままなんだから、T細胞のDJリアレンジメントバンドの出方は共通しているのよね。
- 2019/07/18(木) 16:46:46 |
- URL |
- #-
- [ 編集 ]
バンドの出方というより、あり得る切断断片の大きさはどんなT細胞でも同じだということね。だからバンドの出方に関しては参考になるということだ。実際に出る出ないは別問題で、河本論文はそれを調べている。でも我々が知りたいのはゲル1,2のどのバンドがリアレンジバンドかということだけだからね。
- 2019/07/18(木) 16:47:55 |
- URL |
- #-
- [ 編集 ]
リアレンジのための切断箇所はRSSなのね。この場所は決まっているはずなんだけど私たちは見つけられないからアルイミオウジ変態糞ジジイが探しだしてくるのを持っているわけね。
- 2019/07/18(木) 16:48:50 |
- URL |
- #-
- [ 編集 ]
RSSはイントロンの中にある。ここはTCR再構成だけでなくて、mRNA前駆体をスプライシングするための認識コードも存在しているんで、どちらがどちらか分からないし、そもそもマウスではどういう仕組みなのか、我々は全く分かってない。今いろいろと調べてはいるがこちらは大変なんで時間がかかる。
- 2019/07/18(木) 16:49:46 |
- URL |
- #-
- [ 編集 ]
この論文で私たちにとって重要なのは最後のレーンのポジコンね。Figure3のoレーンとFigure4のiレーンね。
>>
lane o, a clone from IL‐7R+ population of fetal liver (FL) cells
- 2019/07/18(木) 16:50:38 |
- URL |
- #-
- [ 編集 ]
IL‐7R(The interleukin-7 receptor )の説明は以下だ。
>>
インターロイキン7(Interleukin-7、IL-7)は1988年に,骨髄ストローマ細胞株が産生するプレB細胞の増殖を促すサイトカインとして同定された.その後,IL-7は初期T細胞の増殖・生存にも働くことがわかるとともに,末梢でのT細胞の絶対数を一定に保つ恒常性増殖反応において重要な働きをしていることが明らかとなった.さらに,記憶T細胞がIL-7受容体(IL-7R)を高発現しており,その維持にIL-7が重要な働きをしていることがわかっている.また最近では,自然リンパ球がIL-7Rを高発現していることから,IL-7の関与が注目されている.
- 2019/07/18(木) 16:51:21 |
- URL |
- #-
- [ 編集 ]
河本さんは胎児胸腺でT細胞が作られていく過程を研究しているのね。その研究のTCR再構成のポジコンとして胎児肝臓にまで来ている成熟T 細胞のリアレンジメントバンドを提示しているのね。これって小保方さんの脾臓採取のT細胞と原理的には同じ成熟細胞だということね。バンドの出方としてはこうならないといけないのよね。
- 2019/07/18(木) 16:52:28 |
- URL |
- #-
- [ 編集 ]
我々はマウスのJ1は7つあり、J2も偽遺伝子を入れると7つあるということの調べをつけている。ただ、RSSがどうなっているのかが分からないので、リアレンジメントの起きない箇所があるのか否かは分からない。ただ、この河本論文ではJ1に関してJ1.7は調べられていないのと、J2はGLを入れて7本になっていて、偽遺伝子がどれなのかが分からないね。
- 2019/07/18(木) 16:53:18 |
- URL |
- #-
- [ 編集 ]
アルイミ変態糞ジジイの紹介しているもうひとつの論文には偽遺伝子も入れてGL込みで8本のバントのあるやつがあるな。Control of V(D)J Recombinational Accessibility of the Dβ1 Gene Segment at the TCRβ Locus by a Germline Promoter(*ttps://www.cell.com/immunity/fulltext/S1074-7613(00)80031-X)
- 2019/07/18(木) 16:53:59 |
- URL |
- #-
- [ 編集 ]
あれのプライマーは以下で、小保方さんのとは違うわね。
>>
Dβ2 *** #5 (5′-CAGCCCCTCTCAGTCAGACAAACC-3′)
Jβ2.6 *** #6 (5′-CTACTCCAGGGACCCAGGAATTTG-3′)
Jβ2.6の更に後ろ *** #7 (5′-CCCGGAGATTCCCTAACCCTGGTC-3′)
- 2019/07/18(木) 16:54:41 |
- URL |
- #-
- [ 編集 ]
Charles E Whitehurst論文Figure3-Dは偽遺伝子も入れてはっきり8本のバンドが出ているんだよな。小保方さんは河本論文のプライマーを真似して使ってるんだよな。そこは間違いなさそうだ。そして河本論文には7本しか見えない。
>>
アーティクル論文にある小保方プライマー
Dβ2: 5′-GCACCTGTGGGGAAGAAACT-3′
Jβ2.6: 5′-TGAGAGCTGTCTCCTACTATCGATT-3′
>>
河本論文にあるFigure3-Dで使われたプライマー
Dβ2, 5′‐GCACCTGTGGGGAAGAAACT‐3′
Jβ2.6, 5′‐TGAGAGCTGTCTCCTACTATCGATT‐3′
- 2019/07/18(木) 16:55:31 |
- URL |
- #-
- [ 編集 ]
とりあえず小保方さんのアーティクル論文にある概念図はJ2は偽遺伝子も入れて7個でいいんだけど、小保方さんは河本論文と全く同じプライマーを使っているので、河本論文は偽遺伝子を数えずにJ2.6と書いている以上、概念図は1.2.3.4.5.p.6と並んでいるのでなければならない。
- 2019/07/18(木) 16:56:14 |
- URL |
- #-
- [ 編集 ]
小保方さんは慌てて間違えたか、河本論文をよく読まずに実験をしているのね。その場合7つある6番目で切ってると思っていたかもしれないわね。
- 2019/07/18(木) 16:57:08 |
- URL |
- #-
- [ 編集 ]
それは本人に聞かないと分からないが、図と使っている河本プライマーとの間で矛盾があるというのは別に本人に聞かなくても分かる単なる事実だ。なにしろプライマーの並びが一緒だからね。
- 2019/07/18(木) 16:57:47 |
- URL |
- #-
- [ 編集 ]
でもそういう細かいことは実は別にGel2の2NキメラにTCR 再構成があるという事実には関係してこないんだよな。
- 2019/07/18(木) 16:58:28 |
- URL |
- #-
- [ 編集 ]
学ブログ以来なぜ我々が出るべきバンドに拘ってきたかやっと理解してもらえる地点にやってきたかな。小保方さんがリアレンジバンドのポジコンとしてこだわった例の明確に出ている3本のバンドを見てごらん。2Nキメラ1,2には3本の一番下のバンドが無い。
- 2019/07/18(木) 16:59:13 |
- URL |
- #-
- [ 編集 ]
小保方さんはD2-J2.6で挟もうとしているから、D1-J1の間での再構成は全部GLバンドになるね。D1-J2の間での再構成はD2そのものが切り取られるんで検出できなくて0本になるね。D2-J2の間で再構成が起きるとGLも含めて8本のバンドがあり得るということだね。
>>
①Ps-D2-J2の12345p6-Pe(GLバンド)
②Ps-D2-J2の12345p6-Pe(GLと同じだが違いはD2-J2.1の中間部が切り取られて短くなっているもの)
③Ps-D2-J2の2345p6-Pe
④Ps-D2-J2の345p6-Pe
⑤Ps-D2-J2の45p6-Pe
⑥Ps-D2-J2の5p6-Pe
⑦Ps-D2-J2のp6-Pe
⑧Ps-D2-J2の6-Pe
- 2019/07/18(木) 17:00:13 |
- URL |
- #-
- [ 編集 ]
それがまさにCharles E Whitehurst論文Figure3-Dに出ているのね。でも小保方さんのプライマーは河本論文のプライマーだからFigure3-Dのlane o, a clone from IL‐7R+ population of fetal liver (FL) cellsになるわけね。そして二つがどうしてバンド数が1本違ってしまうのかの原因が謎なのね。
- 2019/07/18(木) 17:01:08 |
- URL |
- #-
- [ 編集 ]
そうだね。多分GLラインをどの幅で取っているかによって、長さの近いバンドがくっついてしまうんじゃないかと推測されるよな。
- 2019/07/18(木) 17:01:52 |
- URL |
- #-
- [ 編集 ]
それも少しはあると思うけど河本論文はでてるpバンドを文字表示してないだけだと思うけどな。
- 2019/07/18(木) 17:02:34 |
- URL |
- #-
- [ 編集 ]
私たちの調べではマウスのJ1も7セグメントあるのね。この件に関しては両論文とも6個しか調べてない。河本論文は5個だわ。どういうこと?
- 2019/07/18(木) 17:03:18 |
- URL |
- #-
- [ 編集 ]
姉御、俺が初めてハワイに遊びに行ったとき、オアフ島を一周しようと思ってレンタカーを借りたと思いなせえよ。ダイアモンドヘッドをぐるっと回って、突端のノースショーに行こうと思ったんだけど、どこかで道を間違えてるらしくて何度もワイキキの海岸に戻ってしまうんだ。二回ダイヤモンドヘッドを回った次に別のルートに入ったらしくて、広い道をどんどん上っていくとずっと向こうに大きな建物の門が見えて行き止まりになってるみたいだったんで、一度車を降りてボンネットの上に地図を広げながらもう一度門の方をよく見たら銃を構えた兵士が二人こっちを怪訝そうな顔をしてみてたんで、這う這うの体で逃げ帰った。道に迷って軍司令部に入り込みそうになってたんだな。
- 2019/07/18(木) 17:04:28 |
- URL |
- #-
- [ 編集 ]
鉄、お前の隠し持ってる改造銃程度では自動小銃にはかなわんぞ。ひひひ。どうもど素人が迷子になると知らずに専門分野の最先端に出てたりするようだな。
- 2019/07/18(木) 17:05:23 |
- URL |
- #-
- [ 編集 ]
そういうところが分からないと間違いを犯しやすいけど、とりあえずD2-J2に関してはほぼ見えたんで、そちらを先にやろう。それを終えて仮結論を得てからよく調べて後修正しよう。そうでないとどこを歩いているのか自分でもわからなくなっちまいそうだよ。
- 2019/07/18(木) 17:06:36 |
- URL |
- #-
- [ 編集 ]
🌀 🔓 🔥 🔰 🎴 ✨ 💦 ❄ 💨 🎐 💉 🍷 ❗ 💡 🚧 🎏 🕖 🕚 ⛔
- 2019/07/18(木) 17:10:13 |
- URL |
- #-
- [ 編集 ]









