あらかたの問題は解けた。
幹細胞化の実験は若山さんの研究ですからね。
僕が小保方さんだったらこう言うでしょうね。
私はES細胞でSTAP細胞を捏造してなんか居ないからES細胞に近い性質を持つと書いたと。
ぽこちゃんぺこちゃんブログからOoboeさんは撤退したようだ。私もお気に入りから消した。時間の無駄だ。そもそもあそこは何も新しい情報の無いところだった。照井は血管の専門家だつたかな。道理で詳しいはずだ。素直な心も少しは残っていてチラと出るんだな。はは。TCRのヒントはもらったからな。損はしてない。でももはやああいうことしかできないところまで堕落してるということで、国の研究者は真の発見ができないと教育者になるよりないんだけど、すると人生の目的が違ってしまう。昔デモシカ教師と言われた人々の新版だな。教育は国の根幹で、これはこれでプロの仕事だからな。ここも崩れて久しい。ああいう奴らは愛国心も無いからシナの工作も受けやすくなるということで、今政治でもIR関係で浸透されているのが表立ってきた。米国の圧力だというのが情けないところだが、これからどうなっていくかDORAブログの方が面白いかもね。
同じ、スピン屋でもまだアルイミオウジトゥイッターはフォローしている。あそこにはまだヒントがある。どういう具合にスピンを掛けようとしているかを考えると隠したいことが見える。ぽこちゃんぺこちゃんブログには口汚さしかない。心の中に何か憎しみがあるんだな。妙な奴らだ。国籍が違うのかな。朝鮮総連には成りすましている奴らが多くて、こいつらが実際には日本人誘拐犯だからね。こういうブログではどうせ分からないと思って心の中がさらけ出される。日本人憎しなんだろうな。
Ooboe さんは書き込むところが無くて困まってるだろうね。でもあそこには一般の人はいないよ。一般の人にこの不正を知らせたいなら、まず根本さんブログ、西岡さんガンバレフェイスブックだけどここはスマートフォンからは書き込みにくそうだ。するとやっぱり、パートナー氏のブログ開設が一番ということになる。ガンバレにアップされている動画はもう15万人に増えてる。Ooboe さんたちはこういう人たちに訴えかけたいんでしょ。和モガさんは今ブログを停止しているから相談して、復活させてもらったらどうかな。この間の資料発表会で楠本さんとともに面識を得たんでしたよね。
和モガさんは最初からこの問題を追ってる人だからね。ご自分の説をもう一度纏めて新知見も盛り込んでリヴァイズしてもらうといいですよね。そのシリーズのコメント欄でOoboe さんがサンプルの入れ替えの件を論じさせてもらうといいかもしれない。
Ts.Markerさんは聞いてみないと迷惑かもしれない。ただ、私が延々書きこんでもとりわけ消したりはしないようだ。人は人という感じかな。あの人も言葉不足は有名だからね。でも人は喋れば喋るほど人間が出る。僕は門外だから平気だけど、Ts.Markerさんは一応門内にいる人みたいなんで、あまりしゃべらないように注意しているらしい。このことは以前一研究者ブログで僕に注意してくれたことがあるんで、ああ意識的な行動なんだなと認識している。
それとTs.Markerさんはアルイミオウジ氏を評価している。連絡を取り合ってるようで、流石アルイミさんと言ってる。アルイミ氏はテラトーマにわずかながらOct4-GFPが検出されたのを指摘した人で、最初は無論私は小保方擁護だと思っていた。それとこの発見自体が結構重い事実で、小保方さんがテラトーマを作った時の由来マウスがGOFマウスだという証拠になっている。これはF1マウスは若山さんしか交配許可されていないので他の誰も手に入れられないから当然だと思われているが、それは論理的推論で、アルイミオウジ氏が見つけた事実は物証なんだよな。元のマウスがF1だったということは絶対にないということです。
そのうえでアクロシンを持ち込んだのは誰だ、GFPの無い体細胞は如何にできたのかの問題に解が与えられないといけない。
①無論、私は若山さんがソート前の幹細胞を上から注射したから、リシピエントのES細胞が分化したものだと結論している。
②対して、桂報告やBCA報告は小保方さんが太田ESを使って捏造したと言ってる。そしてGFPの無い組織は免疫不全マウスの体細胞を切り出したと言ってる。間抜けな在米ポスドクに至っては貼り付けて工作したとまでいってる。GLSの捏造には学生にもらったGOF ESが時期的に一致しているのでそれを使ったと言いながら、同じく既に持っていたはずのGOF ESをテラトーマでは使わずに背景の違う太田ESを使ったという。そして小保方さんが捏造したと言っていながら三誌論文までこれを使わなかった理由も答えることができないでいる。小保方さんが出来過ぎを疑ったからに決まっている。
もうこれだけで、桂報告書のインチキは証明されているくらいだ。ただ若山さんが小保方細胞をntES化させたという可能性に気づいていない場合は真相は分からないので、この場合は報告書に分からないと書かないといけない。この段階で既にインチキ報告書になっている。
その上、彼らはntES化の可能性には気づいていたはずである。専門家はキメラが出来るのは既知の知識内ではESかIPSかntESしかないことを知っている。ES捏造を考えてストーリーを作るときに矛盾がたくさんある。その時に彼らはIPSかntESの可能性を考える。特にマウスntESは若山さんが世界の先駆者なんですから考えない筈はない。
ぽこちゃんぺこちゃんブログは桂報告書の構造をそのまま反映しているもので、ESによる捏造説と小保方さん無実説の間に人々を集めようとしている。この中にある限り、ESによる捏造であり、しかし、犯人は分からないというごまかしの中に人々を閉じ込めることが出来ると思っている。そこではntES説は触れられてはいけない説なのである。だからシカト方針になっている。彼らはその可能性を検討しようとはしない。分かり切った話ですね。あの動画を見ようとしている人たちは変だなと考えて居る人たちなんで、ぽこちゃんぺこちゃんブログではだれも納得しないということになる。存在自体が無意味なブログです。
(c,d問題)最後の問題ですね。
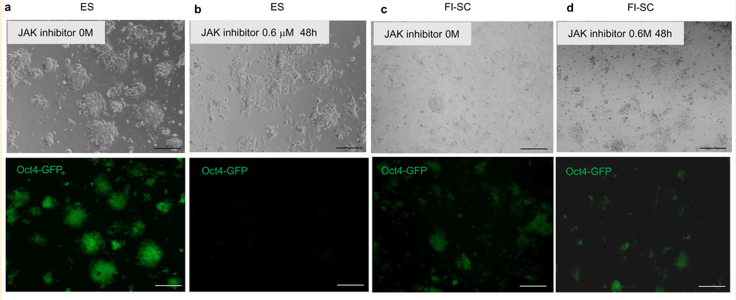
GOF由来FI-SCが存在しなければこの実験はできない。捏造写真は簡単に作れる。さあ、どうですかという問題。ぽこちゃんぺこちゃんブログではシカトされなければならない問題ですね。ぽこちゃんとぺこちゃんの立ち位置の中間のどこにもない問題です。
まずDORAさんの考察から始めましょうかね。
>>
2020年01月14日 12:56
2015年1月1日
じつは遠藤高帆論文もウソだった。
今回の調査報告で、もうひとつはっきりしたことがあります。それは、遠藤高帆論文がでっち上げだったということです。遠藤高帆論文の要点は次の二つでした。
(1)129B6由来の細胞にトリソミーがある。(2)FI幹細胞はESとTSの混ぜ物である。ところが驚いたことに、この二つとも、今回の調査報告では検証されていないのです。まず、トリソミーについてですが、調査報告では、トリソミーはみつかったのですが、それは129B6ではなく、B6の方でした。これでは辻褄が合いません。それから、もっと変なのは、FI幹細胞の方です。遠藤高帆が解析したのはOct4GFPの入ったB6由来のFI幹細胞です。ところが、今回の調査委の報告では、アクロシンの入った129B6由来のFI幹細胞は見つかったものの、
遠藤高帆が解析したB6由来のFI幹細胞は見つからなかった、というのです。?!?!‥‥んなアホな。
遠藤高帆論文を錦の御旗にして「STAPは捏造」と騒いだマスコミ。「STAP細胞・見えてきた実態」とかいう見出しをつけた日経サイエンスの古田彩。このオトシマエどうつけるんじゃ、おんどれ。「遠藤高帆論文はでっち上げでもいいです。忘れてください。今度は調査委員会が別の証拠をでっち上げてくれたので、こんどはこっちを錦の御旗にします」ってか?
これがあんたらのやり口か?結局、遠藤高帆論文の解析結果は、今回の調査報告では全く検証されなかったことになります。遠藤高帆論文は、でっち上げだった。‥‥しかし、問題はそれだけに止まらない。遠藤高帆の解析したFI幹細胞に限って見つからないなんて、ヘンだと思いませんか?他は全部見つかっているのに、これだけ見つからないなんて。これは偶然なんでしょうか? 本当は、見つかったけれども、都合の悪い結果が出たので、隠しているのでないですか? そうとしか考えられないですよ。ますます怪しい調査報告。みなさん、こんなものを鵜呑みにするのですか?
「遠藤高帆が解析したB6由来のFI幹細胞」とDORA氏が言っているのは公共データバンク登録されている以下のデータです。
>>
⑦SRR1171565 若山 FI-SC(Fgf Induced Stem Cells):RNASeq.Rep1 Oct3/4 expressing cells C57BL/6x129/Sv GOF+CD1(丹羽研のBFP-ESの可能性あり)
⑧SRR1171566 若山 FI-SC(Fgf Induced Stem Cells):RNASeq.Rep2 Oct3/4 expressing cells C57BL/6x129/Sv GOF+CD1(丹羽研のBFP-ESの可能性あり)
遠藤論文のTable S1 RNA-seq raw data used in this studyの2P目は以下でした。
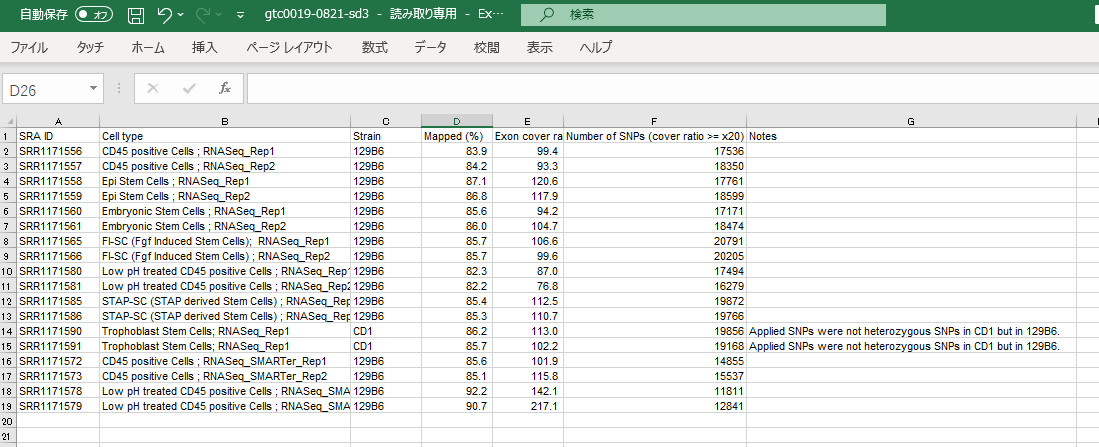
上から8,9行目です。確認できますね。
Ts.Marker さんのSRR1171565データは以下でしたね。


このFI-SCは大量にESマーカーを発現し、同時にわずかながらTSマーカーを発現している。2段目のES細胞との違いは明確ですね。これはTru-seq分です。
さて、DORAさんが「じつは遠藤高帆論文もウソだった。」と怒っている遠藤論文の検討です。
>>
Re-analysis of STAP paper: Genotype analysis of fibroblast growth factor-induced stem cells (FI-SCs)
This study examined how SNP allele frequencies in RNA-seq data can be used to show properties of the dataset. Obokata et al. recently reported the phenomenon of STAP, the induced cellular reprogramming of committed somatic cells into pluripotent stem cells that can produce embryonic and placental tissues when injected into blastocysts (Obokata et al. 2014a,b). The allele frequency approach described above was used to examine the NGS dataset provided by the researchers. Allele frequencies between reference allele (equivalent to B6 genotype for dbSNP) and alternative allele (corresponding to 129 genotype in this study) were examined in RNA-seq data from seven replicate experiments obtained using the TruSeq reagent (Figs2A and S1 in Supporting Information). The allele distributions of six of the seven experiments showed the equal representation of parental chromosomes expected in Fig.1A. For the experiments that involved ESCs, STAP cells, and STAP stem cells (STAP-SCs), there were no 0% peaks (Fig. S1 in Supporting Information), possibly because the cells were obtained from mice backcrossed in the laboratory that may have a different genotype than those in the public database (J. Sharif and K. Isono, personal communication).グーグル訳からちょっと手直ししたものです。
>>
STAP論文の再解析: 線維芽細胞増殖因子によって誘導される幹細胞(FI-SC)の遺伝子型解析
この研究はRNA-seqデータの中のSNP対立遺伝子頻度がいかにしてデータセットのプロパティを表示しうるに至るのかを検討している。 小保方らは最近STAP現象を報告した。それは胚盤胞に注入された場合に胚および胎盤組織を作り出すことができる多能性幹細胞へと変化した体細胞の誘導細胞再プログラミングを意味する(Obokata et al. 2014a,b)。上述の対立遺伝子頻度のアプローチは研究者らによって提供されているNGSデータセットを調べてきたものである。参照対立遺伝子(dbSNPのB6遺伝子型に相当)と、代替の対立遺伝子(この研究では129の遺伝子型に対応する)間の対立遺伝子頻度は、TruSeq試薬を使用して得られた7回の反復実験から得られたRNA-seqデータの中で検討されている(サポート情報の図2AおよびS1) 。7回の実験中6回の対立遺伝子分布は図 1Aで期待されている親の染色体と同じ表現を示した。ES細胞、STAP細胞、およびSTAP幹細胞(STAP-SCS)を含む実験では全くの0%のピークは無かったが(サポート情報の図S1)、おそらく細胞が実験室で戻し交配されたため、公開データベース (J. Sharif and K. Isono, personal communication)のものとは異なる遺伝子型のマウスから得られたからであろう。Figure 2-aは以下です。
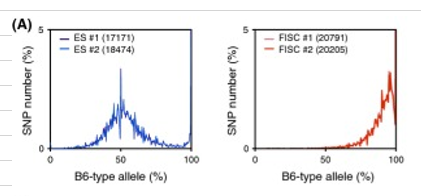 SNPs detected in FI-SC mRNAs indicating contamination. (A) Allele distributions obtained from ESC and FI-SC RNA-seq experiments used in the STAP paper. Both ESCs (blue) and FI-SCs (red) are annotated as having a 129B6F1 genetic background. The number of applied SNPs for each experiment is shown in parentheses in the boxes.
SNPs detected in FI-SC mRNAs indicating contamination. (A) Allele distributions obtained from ESC and FI-SC RNA-seq experiments used in the STAP paper. Both ESCs (blue) and FI-SCs (red) are annotated as having a 129B6F1 genetic background. The number of applied SNPs for each experiment is shown in parentheses in the boxes. Figure S1は以下です。
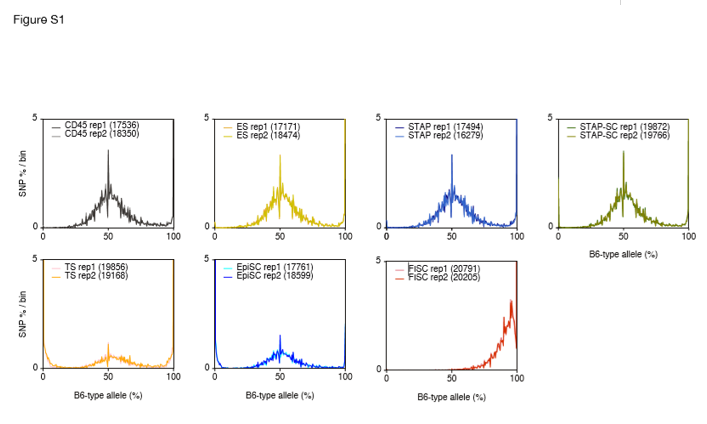
ここで分析されているデータセットは以下です。因みに背景より後ろは後の調査結果の参考メモです。作成者も私のメモです。
>>
Tru-Seq
①SRR1171556 小保方 CD45 positive Cells:RNASeq.Rep1 derived from spleen C57BL/6x129/Sv 129xB6-CAGヘテロ
②SRR1171557 小保方 CD45 positive Cells:RNASeq.Rep2 derived from spleen C57BL/6x129/Sv 129xB6-CAGヘテロ
③SRR1171558 丹羽研 Epi Stem Cells:RNASeq.Rep1 129/Sv
④SRR1171559 丹羽研 Epi Stem Cells:RNASeq.Rep2 129/Sv
⑤SRR1171560 若山 Embryonic Stem Cells:RNASeq.Rep1 C57BL/6x129/Sv
⑥SRR1171561 若山 Embryonic Stem Cells:RNASeq.Rep2 C57BL/6x129/Sv
⑦SRR1171565 若山 FI-SC(Fgf Induced Stem Cells):RNASeq.Rep1 Oct3/4 expressing cells C57BL/6x129/Sv GOF+CD1(丹羽研のBFP-ESの可能性あり)
⑧SRR1171566 若山 FI-SC(Fgf Induced Stem Cells):RNASeq.Rep2 Oct3/4 expressing cells C57BL/6x129/Sv GOF+CD1(丹羽研のBFP-ESの可能性あり)
⑨SRR1171580 小保方 Low pH treated CD45 positive Cells:RNASeq.Rep1 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑩SRR1171581 小保方 Low pH treated CD45 positive Cells:RNASeq.Rep2 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑪SRR1171585 若山 STAP-SC(STAP derived Stem Cells):RNASeq.Rep1 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-Acr-CAG
⑫SRR1171586 若山 STAP-SC(STAP derived Stem Cells):RNASeq.Rep2 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-Acr-CAG
⑬SRR1171590 丹羽研 Trophoblast Stem Cells:RNASeq.Rep1 CD1
⑭SRR1171591 丹羽研 Trophoblast Stem Cells:RNASeq.Rep2 CD1
Figure S1を見て直ぐに気づくのは、TSは丹羽さんがCD1のTSを提供したものですからB6タイプアレルなんてあるわけない。若山さんのTSが分化しているようだったから丹羽さんが提供したというのは桂報告が出て後に分かったものですが、データの末尾にCD1と書いてあるのは最初からあるSTRAINの記載です。Epi Stem Cellsも同じで、129/Svと書かれている。で、この原因をbecause the cells were obtained from mice backcrossed in the laboratory that may have a different genotype than those in the public database (J. Sharif and K. Isono, personal communication).と書いていることになる。推測ですね、どんなB6特異的アレルを使ったのかも心もとない。
因みにEpi Stem Cellsは取り敢えず丹羽さん提供と考えているが、使われているのがアーティクル側なので若山さんの作製かもしれない。今AC129に関してここで考えている問題とからんでいる。キメラは作らなかったがEpi Stem Cellsは作ったのかという問題。
ともあれ、Figure 2-aはESは129とB6のF1だが、F1-SCはB6背景だということを示している。ここをDORAさんが「今回の調査委の報告では、アクロシンの入った129B6由来のFI幹細胞は見つかったものの、遠藤高帆が解析したB6由来のFI幹細胞は見つからなかった、というのです。?!?!‥‥んなアホな。」と批判した。
桂報告書が言ってるのは現存しているサンプルとして「B6由来のFI幹細胞は見つからなかった」と言ってるので、このデータベース登録された分は再解析されていて、B6+10%のCD1とされているわけですから、そこは遠藤調査と一致している。というよりもっと細かく調べていることになる。
>>
⑦SRR1171565 若山 FI-SC(Fgf Induced Stem Cells):RNASeq.Rep1 Oct3/4 expressing cells C57BL/6x129/Sv GOF+CD1(丹羽研のBFP-ESの可能性あり)
⑧SRR1171566 若山 FI-SC(Fgf Induced Stem Cells):RNASeq.Rep2 Oct3/4 expressing cells C57BL/6x129/Sv GOF+CD1(丹羽研のBFP-ESの可能性あり)
さて、もう少し遠藤論文の本文の続きを読み進みましょう。
>>
Surprisingly, FI-SCs that were annotated as coming from the F1 129Sv (129) and B6 cell populations did not show the allele distribution pattern of unbiased nonimprinting genes (Fig. S1 in Supporting Information). The distribution was more similar to that of cells with unequal chromosomes. These FI-SCs were reported to be induced from STAP cells with Fgf4 and to have characteristics similar to trophoblast stem cells (TSCs), such as their gene expression profiles and potential to contribute to the placenta (Obokata et al. 2014a).
The obvious difference in the FI-SC curve from the 129B6F1 genotype, combined with the fact that the majority of SNPs were similar to B6, suggested that the FI-SCs originated from neonatal mice of a nearly pure B6 background. Further analysis of gene expression patterns suggested that the heterogeneity of SNPs between B6-type allele and non-B6 could be caused by the expression characteristics of genes. As shown in Fig.2B, SNPs expected to be heterogeneous between 129 (i.e., non-B6) and B6 were examined in several ESC marker genes. ESCs carried alleles from both the 129 and B6 backgrounds at these loci, but the FI-SCs, although described as having the same genetic background as the ESCs (Obokata et al. 2014a), carried only SNPs from B6. This dominance of the B6 genotype was not observed in TSC marker genes (Fig.2C). The FI-SC specificity was not limited to the genes shown in Fig.2B and C. When all heterogeneous SNPs were classified into three groups, SNPs in ESC-specific genes, SNPs in TSC-specific genes, and SNPs in other genes, only FI-SCs had widely heterozygous SNPs for these groups (Fig.2D). If all included cells in a sample share the same cellular features, one would not expect to see this phenomenon of particular gene sets having different genotypes.同様にグーグル訳です。
>>
驚くべきことにF1 129SV(129)とB6の細胞集団由来と注釈されているFI幹細胞はバイアスのないノンインプリンティング遺伝子の対立遺伝子分布パターンを示さなかった(サポート情報の図S1)。分布は不均等な染色体を有する細胞のものにより類似している。これらのFI肝細胞はFGF4<線維芽細胞増殖因子-4 >によったSTAP細胞から誘導され、かつそれらの遺伝子発現の特徴と胎盤に貢献する能力のように、栄養膜細胞(TS細胞)に似た特性を有することが報告されている(Obokata et al. 2014a)。
大多数のSNPがB6と似ているという事実と組み合わせると、129B6F1遺伝子型とFI幹細胞曲線の明らかな差異はFI幹細胞がほぼ純粋なB6バックグラウンドの新生仔マウスに由来することを示唆している。遺伝子発現パターンの更なる分析は、B6型対立遺伝子と非B6間のSNPの不均一性が遺伝子発現特性に起因することが示唆されている。図2Bに示めされているように、129(すなわち、非B6)およびB6間で異質であることが期待されているSNPは、いくつかのES細胞マーカー遺伝子の中で調べられている。ES細胞はこの遺伝子座において129とB6の両方のバックグラウンドからの対立遺伝子を持ち込んでいるが、FI幹細胞は、ES細胞と同じ遺伝子背景を持つと書かれているにもかかわらず(Obokata et al. 2014a)、B6からの対立遺伝子しか持ち込んでいない。 B6遺伝子のこの優勢はTS細胞のマーカー遺伝子には観察されなかった(図2C)。FI幹細胞の特異性は図2B及びCに示す遺伝子に限定されなかった。すべての異質SNPが、ES細胞に特異的遺伝子のSNP、TS細胞に特異的遺伝子のSNP、および他の遺伝子のSNPの3つのグループに分類されているとき、FI幹細胞のみがこれらのグループに広くヘテロ接合のSNPを有していた(図2D)。試料中に含まれるセルのすべが同じ細胞の特徴を共有している場合、異なる遺伝子型を有する特定の遺伝子セットのこの現象は見られ得ないであろう。Figure 2-bです。
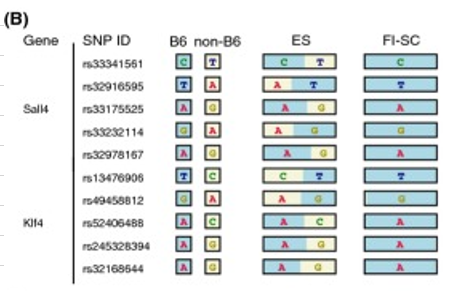 (B) SNPs detected in Sall4 and Klf4, which are highly expressed in ESCs. B6-type alleles are shown in blue and 129-type alleles (i.e., non-B6) are in yellow.
(B) SNPs detected in Sall4 and Klf4, which are highly expressed in ESCs. B6-type alleles are shown in blue and 129-type alleles (i.e., non-B6) are in yellow. 以下のように述べている。
>>
As shown in Fig.2B, SNPs expected to be heterogeneous between 129 (i.e., non-B6) and B6 were examined in several ESC marker genes. ESCs carried alleles from both the 129 and B6 backgrounds at these loci, but the FI-SCs, although described as having the same genetic background as the ESCs (Obokata et al. 2014a), carried only SNPs from B6.
ここは、公共データベース記載の背景にはC57BL/6x129/Svと書かれているが、実際の分析結果は基本B6だと言ってる。小保方さんが登録したんだろと思い込んでいるわけですが、小保方さんは2013/11/5に理研に提出していて、実際にNCBIに登録したのは理研の別の人間で2014/2/13です。
この事件化直後頃に、小保方は公共データベース登録しなければならないことを知らずにいて慌てて2月になって登録したという悪質な噂を流した人間が居て、ここに何か不正がありそうだと誰でも気づいているところですね。
遠藤氏は何かといえば小保方論文に書かれていると書きつけるが、警察でもないのに、何の証拠もなく小保方さんの所為であるかの如くに示唆してはいけない。確かに論文に書かれているが、責任著者には若山さんも居るんだよ。調べてから言わないと。科学者は事実だけを言えばいいんでね。
注に、「(B) SNPs detected in Sall4 and Klf4, which are highly expressed in ESCs. B6-type alleles are shown in blue and 129-type alleles (i.e., non-B6) are in yellow. 」と書かれているんで、ESはF1だがFI-SCはB6だと言ってる。最初に2万箇所前後を統計処理したはずだったが、ここでは全部で10個しか調べてないね。しかもなんだか恣意的な場所だね。小保方さんはESマーカーとしてSall4 なんて選んでないんだから、せめて論文にある9個の中から選べなかったのかな。それだと不都合な真実でもあったのかな。
でも、まあここは理研の後の調査でもFI-SCはB6ベースだったんだからよしとしよう。
10か所選ばれたローカスのどこからどこまでがSall4でどこからどこまでがKlf4なのか分からない。そこで和モガ氏の以下ブログを見ると、一番上のrs33341561はSall4となっているから、それぞれ5か所ずつと推測される。
>>
2017.11.20 Mon l STAP細胞事件 l
「STAP細胞事件」-やはり、「Chip-seq(input) FI幹細胞に欠失はない」でいいんじゃないか
和モガ氏はrs33341561をFLSに導いていって、ほぼ4対6を示すが、遠藤論文はあくまでもESとFI-SCの比較です。それとローカス番号が順に並んでないのはなぜなのか、ど素人には分からない、
和モガ氏の図も拝借しておこう。

ともあれ遠藤論文Figure 2-bはFI-SCのESマーカーのごく一部のSNPsに関して、これはB6背景マウスだと言ってる。ここでは2万箇所の中の10か所だけが示されているということになる。
次はFigure 2-cだ。
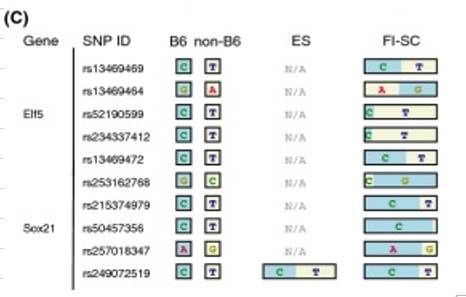 (C) SNPs detected in the TSC-specific genes Elf5 and Sox21.
(C) SNPs detected in the TSC-specific genes Elf5 and Sox21.bがESマーカーのごく一部を調べたのに対して、対照的に今度はTSマーカーを調べた。
Elf5とSox21を選んでいる。あれほど論文がCdx1を強調しているのに選んでないことはとても不思議です。また、Sox21は後にTSに発現するので研究対象になっていはいるが、TSマーカーとして一般的認識は無かったのではないか。その証拠にESでも出ている。
ただ、ここで意図されているのはFI-SCではTSマーカーとして出ているものの中にわずかなB6以外が出ているということを示している。ただし何度も言うがESマーカーもTSマーカーも10か所ずつしか選ばれていない。全体では2万箇所の統計と自分で書いている。
ESとTSを混ぜたのだという主張の根拠として作られているデータですね。小保方さんが挙げているESマーカーは7種、TSマーカーは3種です。全部示さないといけない。これでは何も言えてない。桂調査はこの混ざったとされる細胞をCD1だと示唆しているが、何を根拠にしているのかを示していないし、混ぜられているという根拠も遠藤調査とは独立して何か示しているかというと何もない。
丹羽さんがESとTSは混ざらないと言ってるのは良く知られている。ここに私が(c,d問題)と名付けた理由がある。あの写真がトリックだったら小保方さんの捏造です。あの実験は理研若山研では行われていません。JAKiの検証をするように指示したのは査読者です。若山さんは既に山梨にいます。
GOFのFI-SCがあったのか、それとも小保方さんがTSとESを混ぜてRNAデータを捏造したのかという問題です。写真はそんなことしなくても捏造なら都合のいい写真を選んで組み合わせるだけですから簡単です。遠藤氏が主張しているのはGOFのFI-SCなんか作られていない。GOFのESと誰かのTSの混ぜ物だと言ってるわけです。
Figure 2-dに移りましょう。
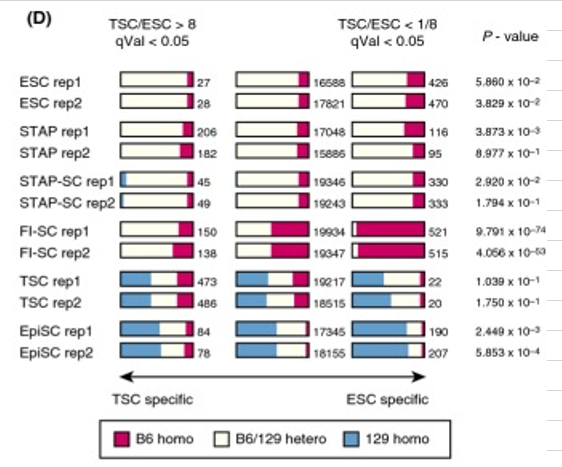 (D) The number of homozygous/heterozygous SNPs observed in the stem cells used in the original paper. Only the composition observed in FI-SCs would be predicted to affect gene expression. P-values were calculated using Fisher's exact test of genotype distribution between TSC-specific genes and ESC-specific genes. Rep1 and rep2 denote two replicated experiments.
(D) The number of homozygous/heterozygous SNPs observed in the stem cells used in the original paper. Only the composition observed in FI-SCs would be predicted to affect gene expression. P-values were calculated using Fisher's exact test of genotype distribution between TSC-specific genes and ESC-specific genes. Rep1 and rep2 denote two replicated experiments.CD1というのはICRのことで、これは近交系マウスではなくてクローズドコロニーマウスです。これの129特異的SNPだとかB6特異的SNPを調べるという言葉の意味が良く分からない。桂報告書には以下のようにあって、遠藤論文と同じことを書いているように受け止められる。
>>
2)FI 幹細胞の RNA-seq データは二種類の細胞種を含んだサンプルに由来する
(調査結果) FI 幹細胞の RNA-seq データと NGS 解析によるマウスゲノムデータとの比較解析により 得られた SNPs データを詳細に解析すると、大多数のアレルは B6 ゲノム配列と一致する のに対して、5-10%程度のアレル頻度を持つ SNPs 箇所が多く認められる。これは大部分 の RNA-seq データが B6 ホモ系統マウス由来の細胞から得られており、別系統由来を持っ た細胞から取得された RNA-seq サンプルが少量混じっている可能性を示す(少量混じっ ていると考えられる RNA-seq データが示す SNPs 分布は TS 細胞の RNA-seq データ(CD1 系統)と酷似している)。 Figure 2-dのTSCはCD1ですし、EpiSCは129/Svです。図の意図すること自体が理解しにくい。桂報告はGOFに混じっているのはCD1だとまで示唆している。そもそもどうやってそんなことが分かるのか。説明がない。ICR特異的SNPsと言うのはないからね。
以下、和モガさんブログにも書かれている。理屈でもそうなる。
>>
2018.01.29 Mon l STAP細胞事件 l
「STAP細胞事件」-ミトコンドリアの特異点はクローンマウスの痕跡である(3)
一方、クローズドコロニーは 5 年以上外部から種マウスを導入することなく、一定の集団内でランダム交配により維持されている系統で、このため、各個体の遺伝的性質はばらつきがある。従って、マウス系統としてのSNPsを登録できないことになる。問題は遠藤論文ではICRマウスのSNPs解析が実際に行われているということです。裏返すとTS細胞は若山さんの作ったF1のTSだと思っているのではないかという疑義です。遠藤氏はFI-SCに関して公共データベースにF1だと書いてあるにも関わらず、分析結果はGOFだぞ。だから捏造ではないかと言ってる。ということは普通に考えると公共データベースのTSはCD1だと書かれていることを知ってるはずですね。このTSがCD1だと知った上で、それでも129とB6の特異的SNPsで解析することの意味はどういうことになるのか。
まず、勘違いなのか、或はそれはそれで意味があるのかという問題です。彼は以下のように書いていた。
>>
For the experiments that involved ESCs, STAP cells, and STAP stem cells (STAP-SCs), there were no 0% peaks (Fig. S1 in Supporting Information), possibly because the cells were obtained from mice backcrossed in the laboratory that may have a different genotype than those in the public database (J. Sharif and K. Isono, personal communication).
Figure S1ではB6の特異的SNPsを調べているので、少なくとも129/SvとCD1ではゼロピークがあり得るのに、実際には無いからには、ここでは何かにバッククロスされたマウス細胞が使われているのではないかと推測している。
彼は理研の調査チームと違って129/SvのSNPsは調べていない。B6だけ調べていて、B6かB6でないか、B6とB6でないもののF1であるかを区別している。この場合、野生型が入ってしまう。
本文に以下のようにある。
>>
The FI-SC specificity was not limited to the genes shown in Fig.2B and C. When all heterogeneous SNPs were classified into three groups, SNPs in ESC-specific genes, SNPs in TSC-specific genes, and SNPs in other genes, only FI-SCs had widely heterozygous SNPs for these groups (Fig.2D). If all included cells in a sample share the same cellular features, one would not expect to see this phenomenon of particular gene sets having different genotypes.
何をしようとしているのかが示されていないのでチンプンカンプンであるが、まず遺伝子を先に3分しているようだ。
①ES特異的遺伝子
②TS特異的遺伝子
③その他の遺伝子
分析対象細胞は6種。
①ES細胞
②STAP細胞
③STAP幹細胞
④FI幹細胞
⑤TS細胞
⑥Epiblast幹細胞
肝心のマウス特異的SNPsが良く分からない。はっきりしているのはB6特異的SNPsは調べている。しかも、このdの表では2種調べたかのような表示になっている。一応以下だ。
①B6特異的SNPs
②129特異的SNPs
しかし、②は論文本文中では非B6となっている。つまり、B6特異的SNPsで無いものという表現である。129 (i.e., non-B6)という表現がある。B6で無ければ129だという認識だが、
B6特異的SNPのあるローカスにある塩基がB6特異的塩基でなかったとき、それは基本野生型の塩基であって他のマウス特異的SNPではない。129 (i.e., non-B6)の認識は本人が誤解しているのか、スピンを掛けているつもりなのか。
更にホモ、ヘテロの割合を調べている。3種。
①B6ホモ
②B6/129ヘテロ
③129ホモ
正しいのは①だけで、桂チームが行ったように②と③は129特異的SNPsを調べないと分からない。上記疑義したように、彼はB6の特異的SNPがあるとされているローカスの上で、違う塩基があったらそれは129だと誤解しているか、スピンをかけている。これだと③は野生型のホモという定義になる。近交系マウスは無論B6や129の仲間だけではなく、いろんな種類がある。にもかかわらず彼はB6で無ければ129という分類をしている。それは公開データベースに書かれている記載にB6/129と書かれているからなのである。ローカスの間違いと二つに限定するという雑さがミックスされている。
因みに、公開データはこの論文に書かれているように、若山さんがチェックしている。若山氏は背景記述にも責任があるということである。謝辞は慶応の吉村氏で、査読文をばらまいた人である。あちこちにいわば共犯者の名前が散りばめられている。税金に群がる仲良しクラブの構造だということである。
ともあれ、更にすべての遺伝子座が調べられているのではなく、恐るべきことに2百万か所を超えようかというマウスSNPsの中から数個の遺伝子だけを選んでいる。以下である。
①ES特異的遺伝子
↓
②TS特異的遺伝子
選ばれた遺伝子は何なのかの記載もない。例えばEpiSC Rep1のES特異的遺伝子数は190個、逆にTS特異的遺伝子数は87個である。どちらでもない真ん中の遺伝子数は17345個で無論圧倒的に多い。ところがこの圧倒的に多い数の半分しか129ホモではないのである。このEpiSCのマウス背景は129/Svである。
①マウス遺伝子数 約25億塩基対
②近交系マウス特異的SNPs数 約2百万塩基対
③ここでB6特異的SNPsとして選ばれているSNPs数 2万塩基対前後
④幹細胞特異的遺伝子の中でB6特異的SNPsとして選ばれているSNPs数 数10塩基対
捏造論文を疑われても仕方がないレヴェル。もしくは未熟者の書いた論文。
qVALはpVALの誤植ではないか。一番右の欄のp Value は10のマイナス74乗だとか53乗なんて、絶対正しいという確度だが、本当かね。
ESC/TSC>8と1/8の間と言うのは二つの比だけでしょ。どういう恣意的な比較なのかね。ほとんど何を言いたいのか分からない。ただはっきりしている主張はFI-SCはB6背景だということだけみたいだな。雑然とした整理の悪い頭の中だと見える。嘘つきの頭の中も大体こういう構造になっている。
それを確認するために先にFigure 4-bを検討してみよう。

リジェンドは以下である。
(B) Heterozygous SNPs detected in Des, Grb2, Setd7, Fbxo21, and Chd4 where B6 and 129 share the same SNPs but TSCs did not.ここではちゃんと129特異的SNPsが調べられている。ただし、B6と129のそれぞれの特異的SNPの一致しているローカスだけである。つまり
"分かっている"のだ。そして、TSには共通のものが無いと言ってる。当たりまえだろ。それは丹羽研のICRマウスだ。
そして、このTSがFI-SCに1割ほど混ぜられたのだと主張したがっていることになる。ところがまず、TSは全21か所のローカスの中で発現してない遺伝子が9か所あって、分かっているのが12か所そして、FI-SCの中でTSの混ぜ物と示唆されている1割程度の中で一致しているのが12か所である。塩基は4種類しかないので、今、12/21になっているものが12/48であれば単なる偶然ということになる。従って、この一致によってそれがTSの混ぜ物であるということを証明したかったらすべての同様の個所を示さないと有意であると証明できない。ただ単に同じ箇所だけを拾い集めただけじゃないのかと思われて当然だ。
嘘つきなんだな。人格が低劣ということだ。まあ、さもなければ、博士なのに馬鹿ということだ。どっちか選べ。コンチクショウ。
もう一度貼り付けましょう。
2015年1月1日
じつは遠藤高帆論文もウソだった。
今回の調査報告で、もうひとつはっきりしたことがあります。それは、遠藤高帆論文がでっち上げだったということです。遠藤高帆論文の要点は次の二つでした。
(1)129B6由来の細胞にトリソミーがある。(2)FI幹細胞はESとTSの混ぜ物である。
ところが驚いたことに、この二つとも、今回の調査報告では検証されていないのです。まず、トリソミーについてですが、調査報告では、トリソミーはみつかったのですが、それは129B6ではなく、B6の方でした。これでは辻褄が合いません。それから、もっと変なのは、FI幹細胞の方です。遠藤高帆が解析したのはOct4GFPの入ったB6由来のFI幹細胞です。ところが、今回の調査委の報告では、アクロシンの入った129B6由来のFI幹細胞は見つかったものの、遠藤高帆が解析したB6由来のFI幹細胞は見つからなかった、というのです。?!?!‥‥んなアホな。
遠藤高帆論文を錦の御旗にして「STAPは捏造」と騒いだマスコミ。「STAP細胞・見えてきた実態」とかいう見出しをつけた日経サイエンスの古田彩。このオトシマエどうつけるんじゃ、おんどれ。「遠藤高帆論文はでっち上げでもいいです。忘れてください。今度は調査委員会が別の証拠をでっち上げてくれたので、こんどはこっちを錦の御旗にします」ってか? これがあんたらのやり口か?
結局、遠藤高帆論文の解析結果は、今回の調査報告では全く検証されなかったことになります。遠藤高帆論文は、でっち上げだった。‥‥しかし、問題はそれだけに止まらない。遠藤高帆の解析したFI幹細胞に限って見つからないなんて、ヘンだと思いませんか?他は全部見つかっているのに、これだけ見つからないなんて。これは偶然なんでしょうか? 本当は、見つかったけれども、都合の悪い結果が出たので、隠しているのでないですか? そうとしか考えられないですよ。ますます怪しい調査報告。みなさん、こんなものを鵜呑みにするのですか?
2015年1月1日
謎が解けた。
記者会見で桂勲氏が「ES細胞を作製するとき(遺伝子に)かなり変異がはいる」と言ってましたが、だったら細胞の培養中にも変異がはいるのではないでしょうか。「培養変異」として知られています。STAP幹細胞やFI幹細胞の培地は特殊です。そんなところにES細胞を混入したら、まともじゃすまないに決まってます。とくにFI幹細胞の培地では「ES細胞は全滅する」と丹羽さんが証言しているのです。全滅しないまでも、少なくとも遺伝子はダメージを受けて変異が生じるに決まってます。だとすれば、元のES細胞と、培地に混入した後のES細胞で、遺伝子が何から何まで同じってのは、むしろ変じゃないですか。皆さん、調査委の報告が絶対正しいと思い込んでいるもんだから、その結果がどんなに変でも真に受けてしまう。「FI幹細胞の培地ではES細胞は全滅する」っていうのに、「FI幹細胞=ES細胞」だなんて、デタラメですよ。
はっきり言いましょう。調査委の報告書は、何から何までデタラメです。完全なでっち上げなんですよ。それじゃあ、どうやってでっち上げたのかというと、そのカラクリは簡単です。若山研の誰かがSTAP幹細胞とES細胞のサンプルをすり替えて渡したんすよ。最初から、どちらもES細胞ですから、どちらも同じ遺伝子の特徴が現れるのは当たり前です。理研内部にも共謀者がいて、小保方さんの研究室のサンプルは、理研の共謀者がすり替えたんですよ。だとすれば、何もかも説明がつきます。
今回の調査で、遠藤高帆氏の解析したB6由来のFI幹細胞だけ見つからなかったのは何故か。それは、遠藤高帆氏が事前に厳密なSNP解析をしてしまったからですよ。遠藤氏の解析結果は非常に複雑なものでした。TS細胞の混入の可能性もあったし、FI幹細胞それ自体にもわずかにTS細胞の遺伝子の発現があった。非常にややこしかった。
STAP幹細胞は、ES細胞とそっくりの性格を持ちますから、ES細胞とすり替えてもばれる危険性はありません。しかし、B6由来のFI幹細胞は、すでに遺伝子を詳しく解析され、(わずかではあるが)TS細胞と同じ遺伝子の発現を持つという結果を出されてしまった。である以上、犯人は、それと同じような遺伝子の特徴を示すサンプルを用意できなかったのです。ESとTSを適当に混入しても、遠藤氏の出した複雑な解析結果と同じにはならない。話が面倒になるので、「B6のFI幹細胞は見つからなかった」ことにした。これが真相です。
2015年1月1日
謎が解けた(2)
わかりやすく補足します。FI幹細胞は、ネイチャー論文では胎盤を形成する細胞だとされていました。そこで、遠藤高帆氏は、B6由来のFI幹細胞の遺伝子データを解析して、「FI幹細胞はES細胞とTS細胞が9対1で混ざったもの」という結論を出しました。とすれば、今回調査された129B6由来のFI幹細胞の方でも同じような結果が出てもよさそうなものですが、そうではなかった。しかし、なぜ、それに疑問が生じないかいうと、こちらの方は事前に遺伝子解析がなされていませんから、どんな結果でも好きなように言えるわけです。「調査委が出した結果がこうなんだから、こうなんだ」と言われてしまえば、それまでです。しかし、B6由来のFI幹細胞の方は事情が違います。B6由来のFI幹細胞は、遠藤氏によってすでに遺伝子が解析されていますから、それと一致するサンプルとすり替えなければなりません。そんなことをするのは面倒だし、リスクも大きいので、「B6由来のFI幹細胞は見つからなかった」ことにしたのです。そういうことです。
2015年1月2日
調査委の報告が遠藤氏論文と食い違う件。
「遠藤氏の解析は正しいのか、捏造か、どっちだい?」という質問を受けました。正直、私も一瞬混乱しました。厳密に言えば、生データを知っているのは遠藤氏の方ですから、究極的には、私が遠藤氏論文の真偽を判定することはできない。しかし、少なくとも言えるのは、調査報告が遠藤氏論文と一致しないのは、おかしいということです。双方が一致しないということは、少なくとも、どちらか一方がウソを言っていることになります。もちろん、双方がウソという可能性もあります。
いずれにしても、遠藤氏は、「FI幹細胞はES細胞とTS細胞の9対1の混ざり物」という結論を出しました。それなら、調査報告でも、それと同じ結論が出てもいいはずです。ところが、調査報告では、そういう結論を示していません。これは変だと気づくべきです。
ですけれども、調査委の側にも言い逃れをする余地があります。‥‥「遠藤氏が解析したデータはB6由来のFI幹細胞であり、当委員会が解析したのは129B6由来のFI幹細胞である。当委員会が解析したFI幹細胞にはTS細胞の混入は見られなかったのである」‥‥と主張できるわけです。
しかし、仮に、遠藤氏の解析したB6由来のFI幹細胞を犯人がでっち上げるとすれば、どうすればいいでしょうか? 若山研にあるB6のES細胞を持ってきて、丹羽氏のTS細胞と「9対1」の割合で混ぜる必要があります。しかし、そうやって、サンプルをでっち上げても、その遺伝子の解析結果が遠藤氏論文と一致するという保証はありません。たぶん、一致しないでしょう。となれば、非常に話がややこしくなります。ですから、そういう事態を避けるために、「B6由来のFI幹細胞は見つからなかった」ことにしたのです。これが真相だと思います。
2015年1月2日
調査報告は完全なでっち上げである。
今回の調査報告は完全なでっち上げである。以下、その根拠をまとめておきます。
(1)FES1は、かつて若山研にいた人が作製したものである。その人は、小保方さんが若山研にやってくる以前に若山研を去っており、そのさいFES1をすべて持ち去っている。それが若山研に残されていたという証拠も全くない。したがって、小保方さんがFES1を盗むことは出来ない。
(2)したがって、小保方さんの冷蔵庫にあった129GFP・ESは、FES1ではない。
(3)にもかかわらず、「129GFP・ES=FES1」であるという調査委の遺伝子解析はおかしい。⇒これは致命的な矛盾です。
(4)129GFP・ESが若山研で作製されたものならば、若山氏がそれを「知らない」というのはおかしい。誰がそれを冷蔵庫に置いたのかを知っていて「知らない」と言っているのであれば、若山氏はその人物をかばっており、したがって、冷蔵庫にそれを置いたのは小保方さんではない。
(5)若山研に所属するES細胞のケースについて、若山氏が本当に「知らない」のであれば、なおさら小保方さんがそれを「知っている」はずはない。いずれにしても、129GFP・ESを冷蔵庫に置いたのは小保方さんではない。
(6)「FI幹細胞の培地でES細胞は全滅する」と丹羽氏が証言しているにもかかわらず、「FI幹細胞はES細胞が混入して増殖したものである」という調査委の報告はおかしい。
(7)少なくとも、FI幹細胞の培地に混入されたES細胞は遺伝子にダメージを受けているはずであり、それが混入以前のES細胞の遺伝子とピタリ一致するというのは、むしろ不自然である。
(8)遠藤高帆氏が「FI幹細胞はES細胞とTS細胞が9対1で混入したものである」と結論しているのに対して、調査委が単に「FI=ES」と断定したのはおかしい。少なくとも、どちらかがウソを言っている(両方ウソもありうる)。
(9)調査委が遠藤高帆の解析したB6由来のFI幹細胞を「見つからなかった」というのは、きわめて怪しい。おそらく、「遠藤氏の解析結果と矛盾しないサンプルをでっち上げることができなかった」というのが、その真相だと思われる。
以上です。
理研には以下のFI-SCがあった。かつ若山さんはGOF由来FI-SCを作っていないと言っている。
①CTS-1 2012/5/25 Acr-CAG-GFP
②CTA-11~13 2012/7/9 Acr-CAG-GFP
対して、小保方さんはGOF由来FI-SCを作るときに当然GOFマウスを渡されたと思っているし、又、現実にGOF由来FI-SCを持っていたから、後の査読者要請に答えてJAKi検証ができたのである。
この矛盾を解決するのに以下の公開データバンク登録されたデータ解析が行われた。
>>
Tru-Seq
⑦SRR1171565 若山 FI-SC(Fgf Induced Stem Cells):RNASeq.Rep1 Oct3/4 expressing cells C57BL/6x129/Sv GOF+CD1(丹羽研のBFP-ESの可能性あり)
⑧SRR1171566 若山 FI-SC(Fgf Induced Stem Cells):RNASeq.Rep2 Oct3/4 expressing cells C57BL/6x129/Sv GOF+CD1(丹羽研のBFP-ESの可能性あり)
遠藤論文と理研チームの解析では、この細胞のマウス背景はGOFとCD1とされた。
遠藤解析ではTS細胞がCD1であるにも関わらずは129ホモが半分近くあって、データ記載のCD1を失念して、F1であるかのように捏造間違いをしているかもしれない。
理研チームの解析結果は公表されていない。
まず丹羽さんの提供したOct4-GFPとOct4-BFP共挿入ES細胞のマウス背景を確認しないといけない。もしそれがCD1だったらあの実験はGOFのFI-SCと10%のCD1の混合で矛盾がなくなる。
もっとも、いずれにせよ、GOFのFI-SCはあるかないかのどちらかでなければならない。
DORA氏の疑義はまだ解決していない。
- 2020/01/14(火) 22:35:36|
- AC129
-
-
| コメント:0














