(英文)
Re-analysis of a retracted paper: Detection of chromosomal aberrations
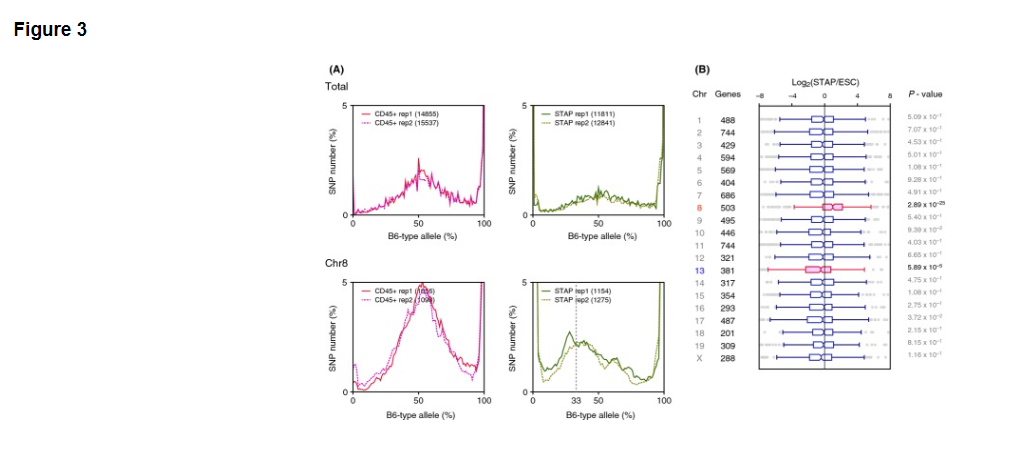
SNP distribution analysis can also be applied to detect aneuploidy. In examining allele frequencies for each chromosome, abnormal chromosomes are assumed to have skewed distributions when paired chromosomes do not have the equal number of duplicates. As shown in Fig. 3A, chromosomal analysis showed abnormality of chromosome 8 in the STAP cells used in the original study. We can expect the peak allele frequency to occur at approximately 50% if a cell has two chromosomes from parents of different strains. The peak allele frequency of the STAP cells, however, was at approximately 33%, meaning that one of the two chromosomes appears to be duplicated leading to three copies of chromosome 8.
撤回された論文の再分析:染色体異常の検出
SNPの分布分析は異数性を検出するためにも適用することができる。各染色体についての対立遺伝子頻度を調べる際には、対の染色体が同数の複写をもたないときに、異常な染色体が歪んだ分布を持つのだと推定される。図3Aに示すように、染色体分析は最初の研究で使われたSTAP細胞における8番染色体の異常を示している。細胞が異なる株の親からの2つの染色体を持っている場合には、我々はピーク対立遺伝子頻度が約50%に起こることを期待することができ.る。 STAP細胞のピークの対立遺伝子頻度は、しかし、約33%であった。それは2つの染色体の一つが染色体8の3つのコピーをもたらすように複製されたように見える。
(英文)
The STAP cells used in the experiments were derived from 129 and B6 cells, and the duplicated chromosome is presumed to be from the 129 parent because it contained only non-B6 SNP alleles. It is notable that trisomy 8 is the most common chromosomal aberration in mouse ESCs, with 31 of 97 examined cell lines reported to carry this abnormality (Mayshar et al. 2010). ESCs having trisomy 8 have a growth advantage, but chimeras will not transmit the mutation to the germ-line (Ben-David et al. 2013), and trisomy 8 in mice results in prenatal death by day 12 or 13 (Kim et al. 2013).
実験で使用されたSTAP細胞は129とB6細胞由来である。重複染色体は129の親からのものであると推定される。なぜならそれが唯一の非B6のSNP対立遺伝子を含んでいたためである。トリソミー8がマウスES細胞の中での最も一般的な染色体異常であることは注目に値する。それは97回の細胞株を調べたうちの31回この異常をもつと報告されていることである(Maysharら、2010)。トリソミー8を持つES細胞は生育が優性であるが、キメラは生殖系列への変異を引き起こさず(Ben-David et al. 2013)、マウス内のトリソミー8は12日目または13日目での出生前死亡をもたらします (Kim et al. 2013)。
(Fig. 3のリジェンド)
Figure 3. Trisomy detected by SNP analysis of RNA-seq data. (A) Allele frequency distributions of whole chromosomes and chromosome 8. Only chromosome 8 of STAP cells had a peak that was not centered approximately 50%, indicating that chromosome 8 originating from the 129 strain was duplicated to produce trisomy of the chromosome. Unlike the RNA-seq data analyzed in Figs 1 and 2, the RNA-seq data analyzed in this figure were generated using the SMARTer reagent kit. (B) Expression analysis by chromosome. Only genes on chromosome 8 were significantly more expressed, and genes on chromosome 13 were significantly less expressed. P-values were calculated using two-sided Student t-tests.
[図3] RNA-seqデータのSNP解析によって検出されたトリソミー。 (A)全染色体と8番染色体の対立遺伝子頻度分布。STAP細胞の8番染色体のみが約50%を中心としないピークを持っていた。それは129株起源の8番染色体が染色体のトリソミー生成するように複製されたことを示す。図1及び図2で分析されたRNA-seqデータとは異なり、この図において分析されているRNA-seqデータはSMARTer試薬キットを使用して作られている。(B)染色体ごとの発現解析。 8番染色体上の遺伝子のみが有意に多く発現しし、13番染色体上の遺伝子が有意に低い発現している。 P値は二群スチューデントt検定を用いて計算されている。
(本文続き)
Because aneuploidy detection using transcriptome analysis has been reported (Gropp 1982; Liu et al. 1997), the expression of genes on each chromosome was analyzed in this study. Genes on chromosome 8 and chromosome 13 had significantly different expression patterns between STAP cells and ESCs. Chromosome 8 gene expression was 1.3 times higher in STAP cells than in ESCs (P-value = 2.89 × 10-25; Fig. 3B). This result is concordant with trisomy of chromosome 8 as detected by SNP analysis. The SNP allele frequency method used here can, therefore, be used to detect aneuploidy if we know the SNP genotype of the cells and if the control cells have a normal karyotype.
メッセンジャーRNA解析を用いた異数性検出が報告されているので(Gropp 1982; Liu et al. 1997)、各染色体上の遺伝子発現を本研究で分析した。 8番染色体と13番染色体上の遺伝子はSTAP細胞とES細胞の間で有意に異なった発現パターンを持っていた。第8染色体遺伝子発現はES細胞よりSTAP細胞において1.3倍高かった(P-value = 2.89 × 10-25; 図3B)。この結果はSNP解析によって検出された8番染色体のトリソミーと一致している。ここで使用されているSNP対立遺伝子頻度法は、したがって、我々が細胞のSNPの遺伝子型を知っていて、かつ、対照細胞が正常な核型を持っている場合には異数性を検出するために使える。
(英文)
Availability of SNP allele frequencies
The SNP allele frequency method detected contaminating cells in samples used to generate RNA-seq data, but the sensitivity of this method is dependent on the depth of sequence reads. Based on the limited number of RNA-seq datasets here, the graphs become too noisy to detect differences in the distributions if the number of available SNPs is fewer than approximately one thousand. For example, MEF cells having only 5948 SNPs generates a more noticeable amount of spikes than data for ESCs with 23 838 SNPs. SNPs assigned to each chromosome also suggested that the distributions could be very noisy when the number of SNPs become <1000 (Fig. S2 in Supporting Information). Cover ratios are also important for the sensitivity because the variance of binomial distribution depends on the number of trials. Therefore, when the average cover ratio is required to be 20× for all genes, the required read count will be approximately 1.3 × 109 nucleotides, roughly corresponding to a read count of approximately 1.1 × 109 bases, which was used to derive the distribution for MEFs (Fig. 1B).
SNP対立遺伝子頻度の有効性
SNP対立遺伝子頻度手法はRNA-seqデータを作成しようとするときに通常使用されるサンプル中の汚染細胞を検出するが、この手法の感度は配列リードの長さに依存する。ここで限られた数のRNA-seqデータセットを仮定すると、利用可能なSNPの数がおよそ千よりも少ない場合は、グラフは分配の違いを検出するにはあまりに雑音が多くなる。例えば、5948個のSNPを持つMEF<マウス胎児線維芽細胞>は23838個のSNPを持つES細胞のデータよりもスパイクのより顕著な量を生成する。各染色体に割り当てられたSNPはまた、SNPの数が1000より小さいときの分布が非常にノイズが多くなりがちなことを示唆している(サポート情報の図S2)。二項分布の分散は試行回数に依存しているため、感度にとってはカバー率も重要である。それ故、平均カバー率がすべての遺伝子の20倍であることが要求されている場合、必要な読み出し回数は、MEF<マウス胎児線維芽細胞>の分布を導出するために使用された約1.1×109塩基のリード回数にほぼ対応するところの、約1.3×109ヌクレオチドであろう(図1B)。
(英文)
Figure 1B also indicates some interesting aspects of genome stability of induced pluripotent stem (iPS) cells. The iPS cells generated from 129B6F1 had more homozygous SNPs of B6-type alleles. Although, as noted earlier, this may be the result of cellular contamination or this could be the result of differences in the properties of the cells used in the two experiments, there is also the intriguing possibility that the experimental process induced a transition of genotypes. As iPS cell engineering has been reported to induce genomic and/or epigenomic instability (Hussein et al. 2011; Chang et al. 2014), it will be important to examine allele frequencies of iPS cells in future studies.
図1Bはまた人工多能性幹細胞(iPS)のゲノム安定性のいくつかの興味深い側面を示している。 129B6F1から生成されたiPS細胞はB6型対立遺伝子のよりホモ接合したSNPを有していた。先に述べたように、これは細胞の汚染の結果であるかもしれないし、この二つの実験で使用された細胞の性質の違いの結果である可能性もあるが、実験プロセスが遺伝子型の転移を誘導したという魅力的な可能性もある。 iPS細胞工学がゲノム的な、および/または、ゲノム外環境的な不安定性を誘導すると報告されているように (Hussein et al. 2011; Chang et al. 2014)、今後の研究においてiPS細胞の対立遺伝子頻度を調べることが重要となるでしょう。
(英文)
SNP frequency differences detected in RNA-seq or other NGS experiments are probably not more accurate than those detected by direct observation of karyotypes; however, the method described in this study can be used to detect chromosomal abnormalities even if the test cells are no longer available, and from both genotype (SNPs) and phenotype (gene expression), which is expected to provide more reliable evidence than genotype or phenotype alone.
RNA-seqや他のNGS実験で検出されたSNP頻度の違いは、おそらく核型を直接観察することによって検出されるものと比べると、正確ではありません。しかし、この研究で記載された方法は、試験細胞がもはや利用不可能である場合でも、遺伝子型か表現型のみによるよりもより信頼できる証拠を提供することが期待されている遺伝子型(SNP)と表現型(遺伝子発現)の両者から染色体異常を検出するために使用することができます。
(英文)
One advantage of the SNP allele frequency method of this study is its potential ability to detect and determine the parental origin of chromosomes duplications. The other advantage over virtual karyotyping (Ben-David et al. 2013) is that it does not require control cells without aneuploidy because simulated models expect the allele frequencies of diploid cells would have peaks approximately 50% (Fig. 1A).
この研究のSNP対立遺伝子頻度法の1つの利点は染色体複製の親の起源を検出しかつ決定する、その潜在能力にある。仮想染色体分析(Ben-David et al. 2013)を介したもうひとつの利点は、異数性のない対照細胞を必要としないことである。なぜなら、シミュレートされたモデルはそもそも二倍体細胞の対立遺伝子頻度がピークを約50%に持っているだろうと期待しているからである(図1A)。
- 2019/05/14(火) 10:07:42|
- 遠藤論文
-
-
| コメント:0



