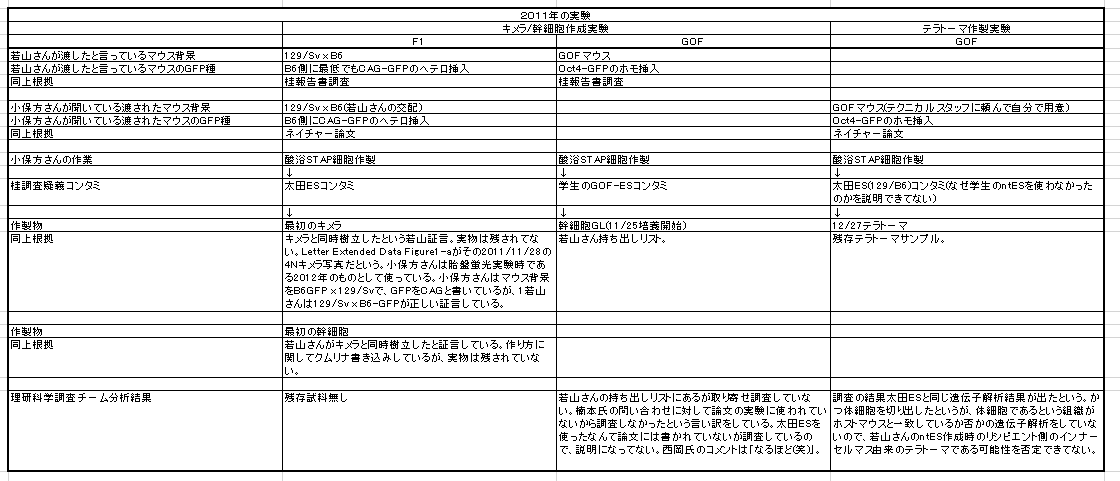
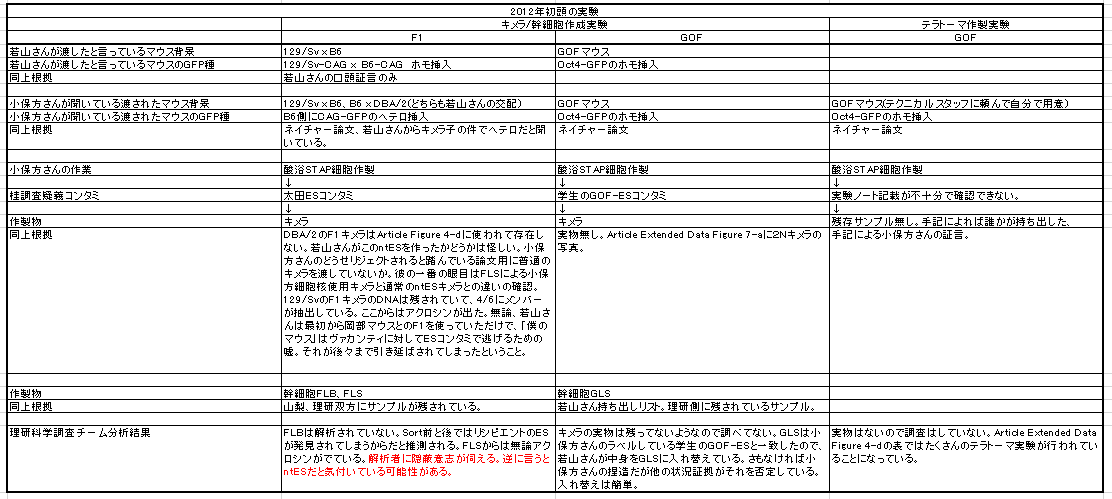
Ooboeさんから連絡が来たので、ちょっと中断してお返事しましょう。なにしろOoboeさんとは以前村松のフルートの話で盛り上がって以来の親友なので、こんな中断は意にも介しません。僕の分身たちも大喜びです。
小野小町
Ooboe さん、お久しぶりね。お元気ぃ。金髪さんもジムさんのところには見えるのにここには見えないから寂しいわね。いつもハワイアンコナを持ってきてくれてたのに、キラウェアが噴火してからきっと入手困難なのね。うふふ。
在原業平
今度又奈良に行きます。前回見れなかったところと、足を延ばして伊勢神宮にも行くつもりです。このブログで又写真アップします。
小野小町
取り敢えずあなたのコメントは以下に貼りつけるわ。
>>
居士さんのお陰でパートナーは質問の資料の整理ができました、ありがとうございました。
さて、パートナーと理研広報や情報公開室を介した調査委員会事務局とのやり取り(CD録音)により、複数の理研組織において、サンプル出所についての認識を、それぞれ組織が同一の報告を共有していたことが明確になってますが、これらの報告資料ではサンプル出所はFES1とntESG1は 山梨、若山氏から、FES2とntESG2は 別の処から、に対しBCA論文ではFES1と2、ntESG1とG2は太田浩から と矛盾。
この矛盾資料の確認質問書を京大、太田氏と桂調査委員に送付しました。併せて、居士さんに教えていただいたオス、メスの矛盾指摘のため、該当ページ論文と、検察への申告書も同封しました。
2019/11/12(火) 08:46:07
それから、検察には、以前の手続きでは提出できなかった、オス、メス矛盾資料をお陰さまで提出します。また今回の委員や太田氏あての質問書送付についても報告するとのことです。
2019/11/12(火) 09:18:11
居士さんのこと、すでに閲覧されているかも知れませんが、Lさんが、このAC129考察についてコメントされてます。私達は、居士さんのスピン評価のようには、彼のことを受けとめていません。
彼は、居士さんと同じく科学的にニュートラルなスタンスの方と思います。
ただ時系列的把握や小保方資料を文献的に認識する作業が海外ての研究制約のため時間的に不十分のようです。断片的なので誤解されやすいかもです。
トータルな把握がなされ(ここ切れてますので後で追加修正してくださいね。)
2019/11/13(水) 09:54:34
それから、お願いがあります、居士さんなら素人の代表として、研究者のLさんとの、純粋なスタンスでの科学議論が成立すると存知ます。結論ありきの雑談コーナでお願いします。
2019/11/13(水) 10:27:55
私は、雑談コーナに一度コメントしましたが、以後入れない判断をされたようです。
2019/11/13(水) 10:32:53鉄
相変わらず名前を入れないんだな。
ふふふ
鉄、Ooboeさんはなあ、スマートフォンだから入力が大変なんだよ。いちいちチイセエことに目くじら立ててんじゃねえや。
鉄
親分、俺たちは以前[「結論ありき」の非専門家ブログが社会を混乱させている:世界三大不正STAP事件の正しい理解を社会に広める会]という人間の屑たちの集まっているところに一度投稿しませんでしたかねえ。
ふふふ
そうだなあ、俺たちは一度[「結論ありき」の非専門家ブログが社会を混乱させている:世界三大不正STAP事件の正しい理解を社会に広める会]という人間の屑たちの集まっているところに一度投稿したよなあ。アク禁くらったっけなあ?
閲覧者
いやいや、我々はかつて[「結論ありき」の非専門家ブログが社会を混乱させている:世界三大不正STAP事件の正しい理解を社会に広める会]という人間の屑たちの集まっているところに一度投稿したけど、アク禁食らうほど長くはいなかったよ。あそこは臭くて長くは居れないところだよ。
小野小町
Ooboe さんったら、ちゃんと相手の家の門の表札見てる?[STAP細胞(実際には小保方氏の虚構)]と書かれているのを見てないの?
>>
「世界三大不正」の1つとも呼ばれる、STAP細胞ねつ造事件の正しい理解を社会に広めることが急務だと思い作成致しました。 現在この件について、科学的見地から正しい説明をしようとする研究者および良心的な方々に対し、STAP細胞(実際には小保方氏の虚構)があるものと思い込んだ人々からの脅迫行為なども行われています。その結果、良心的な研究者がほとんどSTAP細胞について発言できなくなりました。この異常事態を改善し、STAP細胞についての正しい知識を社会に広めるため、このブログを立ち上げました。鉄
ひひひひひ。
STAP細胞は若山氏の虚構だよな。
一言居士
Ooboe さん、あそこは感想さんの立ち上げているブログでしょ。あれは若山研に居たテクニシャンじゃなかったですか。李氏とも知り合いで、ノフラー氏ともハァーイかなんか言っちゃって無かったですか。大阪の万博館かどこかにいらっしたのかな。
鉄
あんなところに書き込んでるやつらなんて全部スピン屋だよ。いつも同じメンバーじゃないか。金もらってやってる。仕事なんだから邪魔しちゃいけないぜ。人は生きて行かないといけない。
ふふふ
まあ、そういうことで御期待には添えませんが、我々は常時チェックはしてるんです。何れことと次第によっては摘発されるかもしれない連中ですからな。我々のしたらば掲示板をああやって破壊した奴も連中の仲間です。我々は復讐を誓ったらとことんやる人間なんですよ。
鉄
親分、モンテクリスト伯みたい。ひひひ。釣りしながら復讐心を煮えたぎらせてるんだな。
ふふふ
あ、雨だと言ってたのに天気になったな。ちと、釣りに行ってくる。
小野小町
あら、ふふふさん、お帰りなさい。マブの入れ食いだったんですって? あはははは。
鉄
親分。今回の釣り紀行の写真はいけねえや。
ふふふ
なんで?
開高健
あの場所、分かるぜ。
アイザック・ウォルトン
君、STUDY TO BE QUIET.って僕が教えて置いたはずなんだけどなあ。
鉄
親分。復讐心でフツフツと心が煮えたぎってたんですね。あらぬこと考えてっから。ひひひひひ。
ふふふ
そうでもないよ。あんまり釣れないから早々と切り上げて周辺の草刈りしてた。
山村聰
釣れない時って帰るに帰れなくて要らん事するよなあ。
閲覧者
Ooboe さんのパートナー氏は着々と計画を前に進めておられるな。
一言居士
僕はCDを聞いてないから何とも言えないんだけど、BCA報告がFES1、2、ntESG1、G2の4つのサンプルを太田浩氏からと言ってるのは、ただ、これら4つの細胞は太田さんが作ってすべて京都大学に持ち出したと言っている以上、経由地点はともかくとしてすべて太田氏だと論理的に推測しているだけではないかな。出典はExtended Data Table 1だね。
>>
Obtained from H. Ohta. Full names: FES1: 129B6GFP1 FES male; FES2: 129B6GFP2 FES male; ntESG1: 129B6F1G1; ntESG2: 129B6F1G2.
閲覧者
桂報告書は少しニュアンスが違うね。
>>
STAP 幹細胞 FLS が、Acrosin プロモーター下に GFP を発現する Acr-GFP と、CAG プロモーター下に GFP を発現する CAG-GFP の共挿入を含むことが判明した後に、 過去に CDB ゲノム・リプログラミング研究チーム(以下「CDB 若山研」という)で作製さ れた Acr-GFP/CAG-GFP 共挿入 ES 細胞を取り寄せて解析したのが、下段の 4 種類の細胞である。小野小町
誰が何をして、どういう経路で、最終的に理研に来たのかが書かれてない。
一言居士
我々は戦略的にパートナー氏は情報を全開示していないと思ってるけどね。お任せしておきましょう。
小野小町
それで何? Lさんの話がどうなのよ。一応貼り付けましょうかね。
>>
5557. L 2019年11月12日 10:29
一言さんが、長い記事をあげてます。どなたか反論ありますか?
BCAで記載されているFES1とFLSのギャップ(獲得変異の不一致率30%)が、感想さんの解析では見られない(9割以上の獲得変異がFES1とFLSの間で一致)事に絡めて、一言さんの主張について考えてみたい気もするのですが、長い記事を細かく読む暇がありません。
ちなみに、一言さんが「残された疑問」としている3点のうち、1番と3番は解決済と思いますよ。CAG-GFP内にプライマーを設定したinverse PCRで挿入部位を同定し、捕まったアクロシンプロモータを内在性と誤解(実際にはコンストラクト内にある)して15番としたのでしょう。18番はもっとシンプルで、実験4でPCRがかかって、実験3でかからなければ、このlocusにはCAG-GFPなしでいいでしょう。
3番目の問題は、キメラ子の親で配偶子ができる過程で、減数分裂組み換えが起きて、Acr/CAG-GFPと欠失が分離したという事でしょう。
どのような意図でローザと説明したのかは不明なので、この問題は残りますね。 在原業平
Ooboe さんは賑やかしの好きな人だからねえ。でもこれってLという人の感想でしょ。人の感想には興味ないねえ。僕は忙しいんだ。
鉄
「長い記事を細かく読む暇が」無いんだろ。仕事せえや。若いうちからさぼり癖をつけるな。死ぬほど働いたこともねえんだろ。クソが。
ふふふ
鉄、お前は口汚くていけないよ。留学して何をしているのかは知らないが、誰の金でイギリスに行ってるのかな。親が泣いてるのか、国が泣いているのかは分からないが、若いうちの時間は濃密だからな。無為に流れたか身になったかでは後のレバレッジが違うよな。
小野小町
ふふふさん月末には奈良に行くんでしょ。早くホテルの予約しないと。コース決めなさいな。
ふふふ
法事の写真も送んないといけないんだったな。何しろ年取ると一日が短いのにやりたいことが多すぎて、そしてすぐ疲れる。どうしたらいいんだ。
鉄
親分。死ぬほど楽は無かりけり。もう少しの辛抱ですぜ。
ふふふ
鉄、お前間違ってるから。寝るほど楽は無かりけりだ。そうだ。家に帰って寝よう。
小野小町
もう寝ちゃうの。まだ宵の口だというのに。
アイザック・ウォルトン
釣れなかった日の心は重い。
井伏鱒二
さぞかし瞼も重たかろう。
- 2019/11/14(木) 09:41:48|
- AC129
-
-
| コメント:1
(本題に戻ります)問題はSNPs解析に戻ります。再掲しましょう。
一番下のたまたま分析を受けた129 cag-GFP mouseの15番染色体のSNPsパターンは最上段3つの細胞とは違うというところを確認してください。これは若山さんの129マウスにマウスコンタミがあるからでしたね。解析を受けた129と上3つを作った時の129が少し違うのです。でも上三つは同じマウスからできている。そしてAC129はどうせ若山さんが「僕のマウス」ES-6を詰め替えたものです。しかし、STAP Lysateは違いますよね。これは小保方さんがChIP検査のためにかき集めた細胞だと証言していると伊藤さんが記者会見で語っています。人から取り戻している細胞ですね。このときに誰かが129B6F1 ES6を渡しましたね。AC129の実験時ではありません。小保方さんはF1のSTAP細胞として提出しているのです。
このことは何を意味するかというと、既に2012年の8月には小保方さんは嵌められているということです。そして原因は恐らく特許での対立です。若山さんはこの対立での経緯でこのまま放置できなくなった。万が一の時には小保方さんの捏造で逃げる準備が行われている。と考えると何となく辻褄はあう。でも、これはSNPs解析結果ですね。思い出してください。Nanogの発現解析はSTAP細胞とES細胞は違っていましたよね。伊藤さん。本当にSTAP Lysateが残っていましたか?
>>
ただ、唯一やったのが、あ、GRASの方に残っていたChIP seq のinput 、しかもSTAP細胞のインプットはDNAとして残っていたので、それが10ページバワーポイントの、えーと、ところに書いてある、3番のアイテマイズして書いてあるところですけれども、これに関しては、えーと、30倍になるだけ、他のゲノムと同量となるまで読んでいます。そして、読んだ結果、この、同一であるということを、ま、認定したということで、それ以外に、それ以外に関しては、ま、残っていませんのでやってない、やってません。(*ttps://www.youtube.com/watch?v=M9oJGioHvIQ 1:15:00から)
因みにここで伊藤さんはこのたくさん集めた細胞は別の実験だと言ってますね。つまり彼らはこれがAC129の実験時のSTAP細胞ではないことを聞いているんです。にもかかわらず、「公開データ再解析の結果に よれば、論文に記載された実験の中では Letter Fig.4 に使われた可能性が高く」と書いているのです。嘘ですね。彼らはここで小保方さんが若山さんの「僕のマウス」ESを使ったと主張しているのです。そのくせNanogの件は無視している。それならこの時のESは何なのか。
GRASに残されている記録を全部発表してもらいたい。若山さんのこの頃の実験ノートを出してもらいたい。どうして小保方さんばかりに訳の分からない因縁をつけているんでしょう。小保方さんは調査チームを信用して実験ノートを提出しているのに、彼らは全コピーをNHKに違法に流出させて犯人の検挙もしてないだろうが。告発すらしていない。犯罪を知ったら警察に届けるという公務員義務違反をしていて恬として恥じない。日本の知的レヴェルは明らかに下がってますね。こんなのが博士だと思うと情けないね。ははは。
さて、いよいよこの時の129/Svは何なのかという問題に入れるわけです。FLS3以下5つの細胞株の6番染色体が一番下の129 cag-GFP mouseとは違うことを確認してください。これも又別のコンタミマウスなんですね。でも他のB6のSNPsパターンは全部同じだということも確認してください。これって市販の129/Svマウスではありませんよね。アルイミオウジ氏も疑義を呈している。でももし129 cag-GFP mouseだったら18番染色体にヘテロでGFPが入っているはずです。
やっと最初の問いに戻りましたね。どうしてFES3のGFPは岡部マウス側のAcr-CAGのみが調べられて、母親側の18番にCAGが無いかどうかを確認されてないのか。もしそれがあったらFES1,2には無いはずのものですよね。仮にterで作ったという記憶が間違いでX1であったとして、しかもそれは市販の129/Svではなく、更に若山さんの129/Sv-CAGホモだったとして、更に、どうして7年も前のコンタミマウスのSNPsパターンの同じ親にぶち当たるのだ。
FES1,2ともに中身の細胞は若山さんによってFLS3に入れ替えられているということです。
さあ、大問題です。若山記者会見です。
*ttps://www.youtube.com/watch?v=hD3Wd9cpo78
FLS1-8は15番にヘテロで4コピーのGFPがあり18番には無い。「僕のマウス」シリーズには15番にはGFPは無く、18番にホモで1コピー入っている。誰でも知ってることですね。
ところで15番にGFPが入っているという第三者機関の発表は間違いとされた。遠藤さんが発見したことになっているが、それはともかくとしてではFLSの18番にはGFPが無いという結論はどうなっているのか。一方が間違っているのに他方は合っているというのも変な話しで、その場合は間違った原因を述べなければならず、又同時に18番を調べた手法の確認も必要です。でもそれはうやむやになっているままなんですね。SNPsパターンは129x1/Sv-CAGホモマウスの特徴がほとんど全染色体に分布してますよね。どうなってるんでしょうかね。
今、アルイミオウジ氏がいくら兄妹交配を重ねても決してホモ化しないSNPsがあるなどというトンデモ主張中ですが、又、テヘで終わるのでしょうかね。そんな現象があるのなら、そもそも報告書のSNPs解析は意味を持ちませんね。
有る特定の129マウスが近交系マウスになったというのは兄妹交配回数が20回を超えて、基本すべての対立遺伝子がホモに揃っているものです。129特異的SNPsというのはその全遺伝子の並びの中で野生型と比較してSNPとして違っている箇所が特定されているということです。近交系マウスの中では全部ホモになっているSNPsです。これは定義なので近交系マウスの中のヘテロなSNPsというのはあり得ないんです。まずヘテロなSNPsになっていたら近交系マウスではない。以前から説明せずに一人よがりする人ですから他所から見て何とも言えませんが、近交系マウスの中でヘテロなローカスというのはいくらでもある。全遺伝子がホモに揃っているというのは実験目的での実用の範囲で言うものです。実際にはヘテロなアレルは残っています。以下が参考になる。
*www.anim.med.kyoto-u.ac.jp/Kuramoto/contents/ExpAnimGenet_1_inbred.pdf
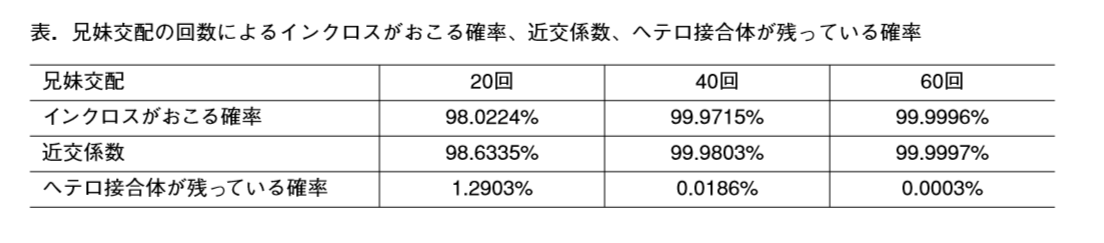
マウスの場合は胎児から成熟して妊娠できるようになるまで20日と50日の合計70日かかる。20回の交配というのは2.8年、40回の交配というのは7.7年、60回の交配は11.5年です。マウスの全塩基数は30億個程度です。60回やってヘテロ残存率が0.0003%になった時の数は9000個です。染色体数を20として一本に450か所ある。20回の交配後既に近交系マウスとして扱われるので、その場合では38,709,000か所残っているので染色体1本につき約2百万か所のヘテロなアレルがあることになる。
このヘテロなアレルは両親の違いがそのまま残っているだけのものです。これを更に兄妹交配して世代を重ねていくとヘテロだったものがどちらかに固定されていくんです。一旦固定されると何度交配してもホモなんですから子供には同じ核酸が分配される。これがインクロスと呼ばれる交配なんです。全部がインクロスになったら完全クローンになりますが、理論的にはこのやり方では無限に繰り返してもヘテロはわずかに残ってゼロに限りなく近づくが完全なゼロにはならない。
この問題と、ある近交系マウスに特異的なSNPsというのは無関係です。これは野生型と違う核酸になっている場所のことで、近交系マウスではほぼすべてのアレルがホモですから、ホモの中で野生型と違っている場所という意味です。ヘテロなSNPsなんて子供を造ったら分かれてしまうのでその近交系マウスに特異的SNPということにならないんです。定義の問題ですね。ホモで且つ野生型でない場所。そして近交系マウスの種類によって核酸の種類も場所も違っているものを特異的SNPsという。
まあ相変わらず一人で分かった気になってまともな説明が無いので端からは何とも言えませんが、この問題の解にはなってない筈です。
アルイミオウジ説の話になったので序に書いて置きますが、彼はntESG1,G2のGをgenerationと解して、最初のntESがG1で、それを使ってキメラマウスを作って更にntES化したのがG2と推測している。そして実際に若山さんはこのやり方で20世まで作っていて別の論文があるわけです。でも太田さんのG1ははっきりしていて、2008年論文で2005年論文で作ったF1のntESを解凍して使った。その解凍した細胞を129B6F1G1と書いているんです。すると解凍する前の状態のラベルはどう書いてあるのかということになる。株分けした時に以前の分に129B6F1G2と書いたとしたらこれはgenerationの意味ではありません。ただ、日経サイエンスに太田さんは自分は2種類の受精卵ESと4種類のntESを作ったと証言している。太田さんに聞けばわかることで、彼には説明義務がありますよね。でも、チューブのラベルと中身が違っていることの説明は彼一人の判断では出来そうもありませんよね。
(残された問題の整理)さて、問題の数も少なくなってきましたね。ただし、解の困難さは増してきました。ここらで残されている問題を整理しておきましょう。
①FLSは「僕のマウス」を渡したと一方的に証拠の提出もなく若山さんが主張しているが、実際の検査で出て来たのはAcr-CAGのB6(岡部マウス)と市販のGFP無しの129X1/SvのF1だという報告書の調査結果となっている。しかし、129側の18番染色体に1コピーのCAG-GFPが入っているのではないかという疑義がでた。なぜなら、SNPs解析結果がこの129は「僕のマウス」の片親であることを示唆しているからだ。最初に放医研がFLSを検査した時、15番にヘテロに4コピーのCAGがあり、18番にはGFPが無かったと発表していたが、後に15番のGFPが否定されたが18番に関しては何も触れられなかった。又間違いの原因もちゃんと説明されていない。内在性アクロシン遺伝子を間違えたのならホモだと思うはずでヘテロに間違うというのは考えにくい。調査自体がとてもあやしい。すると18番に無かったというのは本当なのかという疑義が残るということです。理研のチームはGFPを自分でちゃんと確認したかということです。この説明も無い。
②小保方さんはなぜローザだという説明を受けたのか。彼女は徒手空拳で何の理由もなくローザと書く動機が無い。誰かがローザだといった筈である。
③なぜカルスキメラ子の8番に3番染色体欠失があるのにAcr-GFPがないのか。多分これが我々素人にとっては最大の難問になりそうですね。というのも他のことは誰かが何かをしたとか、し忘れたとかいう可能性を思いつけるが、この問題だけはど素人ではなぜこうなるのかの一つの可能性すら思いつけない。
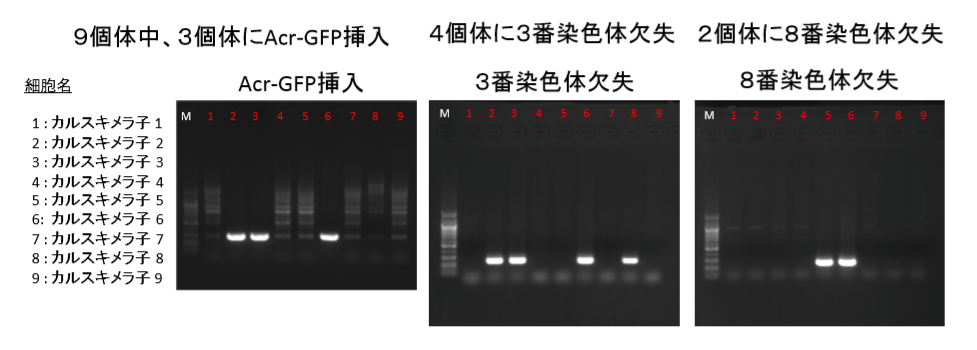
欠失とAcr-GFPはB6の同じ3番染色体上にある。一つの細胞に欠失が起きてそれが分裂して増える。最初は一つの細胞です。ここにAcr-CAGがあったらその後の体細胞分裂でGFPはずっと引き継がれる。欠失は同じなのにGFPが無いということはあり得ないんで、その場合は欠失の種類が違うということしか考えられない。逆にGFPありとGFP無しの細胞に同じ欠失が起こることも無いですね。まあ、せいぜい突然変異でGFPが消えたというなら分かりますけどね。そんな確率で起きることですかね。
この表ではB6に129がコンタミしている場所を指摘していますが、129にB6がコンタミしている場所を提示していませんね。もう一段深めて推定する発想力が無いんですね。そういう発想力があったらマウスコンタミの無いB6と129のデータをコントロールとして並べますよね。客観的たろうという姿勢が無いんです。まあ、科学者としての資質としては相当低い人々でしょうね。一般の会社に入っても役だたずでしょう。こういう奴って悪いことばっかり考えるんだよなあ。ときどき見かけるタイプ。流出させてはいけないデータを羽織ゴロツキとかNHKなんかに流す。

NHKに調査チームが病床の小保方さんの手元から奪い取るように持ち去った実験ノート3冊分の全コピーが流出するという犯罪があった。誰が流出させたのかをNHKが答える義務はない。又理研内での自己点検報告の草稿が作られたとき、それが毎日新聞の記者にリークされた。このリーク者を毎日新聞が公表する義務はない。しかし、二つの公務員としての犯罪が行われたことが明確であるにも関わらず理研がこの事件を警察にとどけることが無かった。これはこれ自体が犯罪ですね。
これは追及されるべき事柄です。日本は法治国家では無いのか。
内部告発も取材源秘匿の自由もちゃんと保証されている。でも違法行為は警察が取り締まるという義務も又課せられているんだとということを忘れてもらっては困る。理研は今からでもNHKに小保方さんの実験ノートを流出させた犯人を捕まえるよう依頼しないといけない法律になっている。無論、その際でも、NHKは警察に対して犯人が誰かを教えなければならないというような義務はありません。取材源の秘匿は法で保護されている。ただし、裁判所の令状があると別ですけどね。裁判所は裁判所で個々のケースで令状を出すか否かは判断します。このケースでは出ませんね。でも警察はそんなもの無くても犯人をすぐ特定しますよ。捜査権限がありますからね。実験ノートに関与した者なんて限られたわずかな人数です。こんなことをそのような人間が調査したとするサンプルの中身に工作しなかったなどと考える方がどうかしてますよね。何れ、その問題は最後の課題となるでしょう。
(①の問題から順番に)SNPs解析結果は129が「僕のマウス」の片割れだと示唆しているにも拘わらず、その18番染色体にCAG-GFPが無いという結論は放医研の出したときのままになっている。そしてB6側の結論が間違っていたと判明した後に理研が129側を確認したか否かも知らされていない。
放医研の調査結果を若山さんが記者会見発表して後に、遠藤氏がそのデータを若山さんから貰って、放医研の出した15番にヘテロに4コピーのCAG-GFPがあると発表した結論は間違いであったと言った。放医研はCAG-GFPを探していたので、CAG-GFPのプロモーターの位置をプライマーで挟んで探していた。その時に、そのプライマーではアクロシン遺伝子の有る場所を検知してしまったのだという。支離滅裂の説明ですね。意味が分からん。

まず不可思議なのはFLSの15番に4コピー入っているとしたPCR産物は何かということです。どういうプライマーで挟んだのかということです。CAG-GFPの挿入された場所を探している。CAG人工遺伝子の並びそのものをプライマーにかけるということになる。
若山さんは自分のマウスはCAGだと説明していますね。だから放医研はCAGを探そうとした。そして順序はともかくとして「僕のマウス」には18番にホモでCAGがあるとした。若山さんは今まで自分のマウスのGFPがどこに入っているのか調べたことがなかったのでしょうかね。岡部マウスはもらった時に3番に入っていると聞いていたわけでしょ。岡部マウスは放医研では調べていませんよね。でも記者会見では3番だと答えている。普通自分のマウスのGFPがどこに入っているかは調べるよね。そもそもマウスの飼育はときどきコンタミが無いかどうかのSNPs検査にも出すものですよね。

FLS1-8の18番にはCAG-GFPが無いが、「僕のマウス」ESにはある。しかもホモで入っていると。
では、15番はどうか。FLS1-8にはある。しかも「僕のマウス」ESには無い。ところがこの15番は間違いだったという。では18番は正しいのか?
これが分からないわけです。桂報告書もGFPが何番染色体に入っているかをリスト上に記載していない。15番を間違えるような奴が18番は間違えなかったとどうしていえるでしょうか。そもそも探したプライマーを明らかにしていない。本当に両方とも同じプライマーで挟んだのかということすら疑われる。18番のCAGが正しいなら15番のCAGも正しいはずだ。アクロシン遺伝子はヘテロに4コピーなんてあり方はしないぞ。これは別のプライマーじゃないのか。
記者会見の画像ではプライマーのところが写されてないものもあって分かりにくいので、片瀬久美子氏のブログのを参考に以下に貼り付けて置こう。
上にどういうプライマーで挟んだのかが図示されている。コンストラクトというのは人工遺伝子のことで、無論これは若山さんがロックフェラー大学でB6にヴィルスヴェクターを使って遺伝子挿入したときのコンストラクトです。若山さんが行ったのですからその遺伝子構成は若山さんが知っていて、これを探すときに若山さんは放医研の知り合いに教えている。で、その末端につけられているポリAのあるあたりの部分にプライマーを設定した。要するに若山さんの人工遺伝子の尻尾を探したわけです。
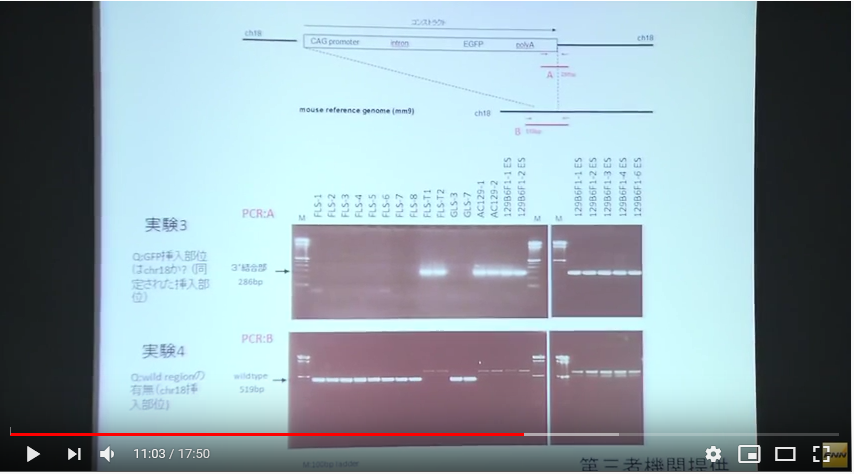
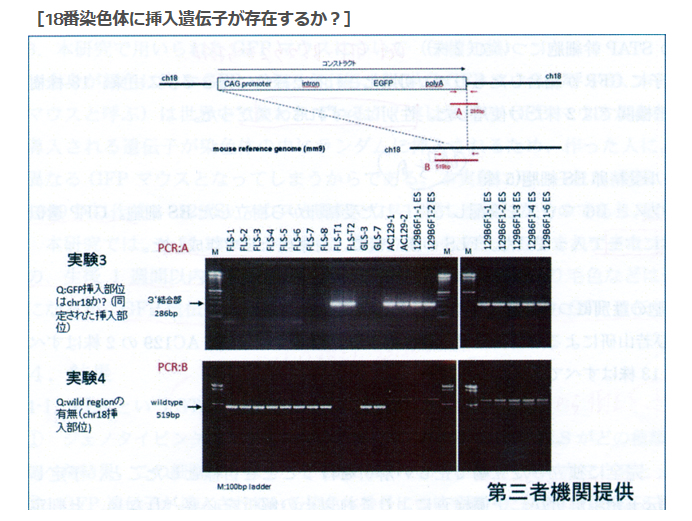
実験3は18番染色体を調べた結果FLS1-8には存在せず、所謂「僕のマウス」シリーズにはあった。従って、若山さんの「僕のマウス」のGFP人工遺伝子は18番に入っていたことが分かったというものです。
このプライマーが分からないわけです。上の図のAの長さは286bpと書かれていて、プライマーの長さは通常20bp程度を前後に二か所設定するわけです。前はポリAのある場所ですが、後ろはコンストラクトの外に表示されている。でも、これは人工遺伝子全体が挿入された場所が分かっていませんからこの後ろのプライマーのDNAの並びは分からない筈ですね。プライマーが分かっていたら既にどこに有るかわかってるということですから調査する必要はない。
ど素人が常識的に若山さんの人工遺伝子を探そうとするとき、コンストラクト全体の遺伝子の並びは分かっていますから、頭の20bpと尻尾の20bpで挟みますね。そして染色体ごとに探すとまずGFPの存在している染色体番号は判明しますよね。もっとも頭の並びはプロモーターの並びなので内在性のプロモーター位置を掴んでしまうかもしれませんが、後ろの20bp以上並ぶポリAはDNAにはないので特定できそうですし、それも何らかの紛らわしいことがあるのなら、中にあるEGFP遺伝子の頭と尻尾に設定してもいいですね。要するに何番染色体に存在しているかということはそんなにむつかしくなさそうに思われる。
ところがこの実験3は妙なプライマー設定だと見受けられる。後ろのプライマーはコンストラクトの外に表示されてませんか。これって挿入位置が既に正確に分かっているコンストラクトの存在を再確認しているだけではないのか。
また、実験4は何をしようとしているのかがど素人には分からない。こちらは全体の長さが519bpです。そして図を見る限りはコンストラクトの少し前の位置から後ろは実験3のプライマーの後ろと同じようです。つまりこの位置の中にコンストラクトがあることがあらかじめ分かっているということです。そしてその区間にFLS1-8ではワイルドタイプの遺伝子が並んでいるのだということなのでしょうが、では「僕のマウス」シリーズではどうかというとバンドが何もない。つまりその中にコンストラクトが挿入されて長くなっているから519bp以上の長さになっているからこのバンドのラインには出て来ないんですね。
これって、どこに入っているかは事前に正確に分かっているということじゃないですか。若山さんは挿入位置を教えて、それがあるか無いかを放医研に確認させただけではないですか。
となると、岡部マウスの情報も分かった上で確認させているのではないかと疑われて来る。
岡部マウスは3番に入っていることは放医研で調べたことにはなってなくて、かつ記者会見では3番と若山さんが言ってますから、あらかじめ岡部さんから教えられている。でも、この調査ではアクロシンなんて思いもよらないという前提で放医研にCAG-GFPの在処を探させているということになっている。
マウスの15番には内在性のアクロシン遺伝子が存在しています。こちらは実験3,4の後です。FLSには18番にGFPが無かった。でも蛍光してますからどこかにはある。で染色体を虱潰しに調べたら15番で引っかかってきたという。変ですよね。これはCAGの遺伝子配列をプライマーに使っている。若山さんのコンストラクト全体の中でプライマー設定されている。15番にはEGFPはありません。因みにGFPとEGFPは同じです。蛍光発色の強化されたものです。
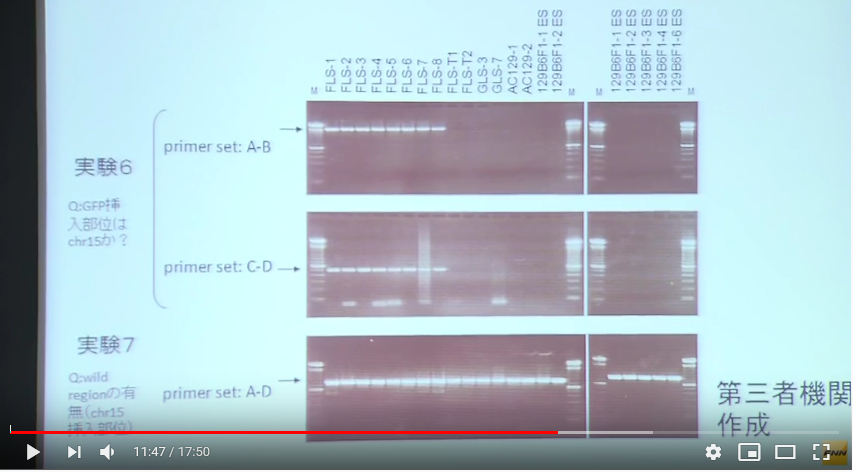
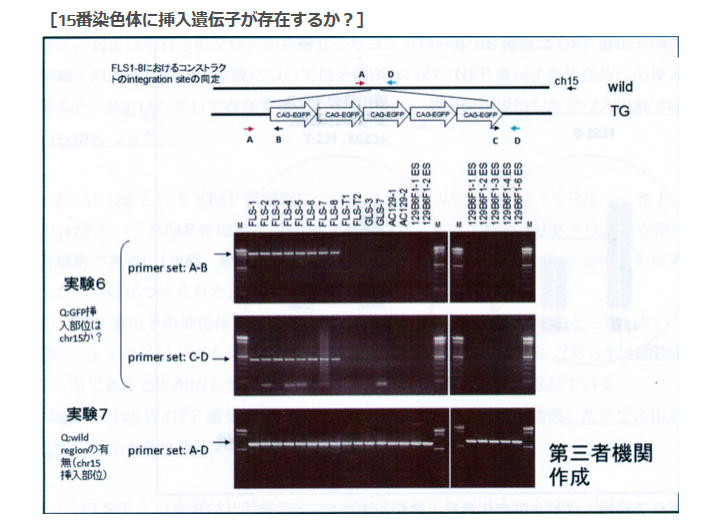
実験6は15番を調べたものと若山さんは記者会見で解説した。このPCR結果はあり得ないものです。GLSに無いのは当たり前ですが、そもそも15番にはGFPはない。それがFLS1-8には出ている。これは何なんでしょう。
そして実験7はA-Dで挟んでいる。これって若山さんのコンストラクトの中です。絶対に出ません。何なんでしょうか。
ひょっとして岡部マウスのコンストラクトを使ってないかと考えてみるとどうなりますかね。頭にアクロシンプロモーターがついている。すると15番にある内在性アクロシン遺伝子のプロモーターを掴むということはあるんですね。そして後ろのDの並びがあったかどうかでしょうね。
(どうしてこんなに分かりにくいのか)まずは実験3と4から確認していきましょう。見やすいように再掲します。

プライマーAはコンストラクトの後ろの方にポリAの並びがあって、その辺りからコンストラクトの末尾の少し後ろで挟んでいる。赤い矢印がそれですが、その長さは286bpと記載されている。そしてプライマーBはコンストラクトの少し前からプライマーAの後ろの位置と同じく、コンストラクトの少し後ろで挟んでいて、その長さは519bpと書かれていて、割り算するとプライマーAの長さは実寸でプライマーBの55%の長さです。この図でも赤の矢印とバーの長さと数値はそれを正しく示している。でも私は最初プライマーAはポリAの付近を挟んでいると思いましたが、他の人はどうなんでしょうかね。プライマーAのバーの長さと上の[CAG promoter---intron---EGFP---polyA]の概念図と比較すると尻尾の部分と誤解しますね。でもプライマーBはコンストラクトの長さを縮小していますよね。破線で縮小が示されている。プライマーの長さは数値で明確に書かれていますから、プライマーAはEGFPまで含んでいるんですね。全体の55% です。どうしてこんな紛らわしい表示にするのでしょうかね。全体を縮小しなかったらいいだけですね。何も面倒でないどころか、その方がシンプルで分かりやすい。縮小している意味が分かりませんよね。人が嘘をつこうとするときこういう姑息な手段を取りがちだということはまあ、警察のようにそれを仕事にしている人々だとか、そうでなくても比較的長く生きて経験豊富な人たちにとってはお見通しでしょうかね。しなくていいことをしている。分かりにくくするため以外には考えられない操作ですね。
コンストラクトの少しでも外側を使わなかったら実験4は出来ないというのは当たり前ですが、少し外側がプライマーとして知られているというのは事前にどこに自分のGFPが入っているかを知っているということです。
プライマーはAGCTの4種の塩基の並びの20桁程度を使います。4の20乗ですから組み合わせ数がほぼ1兆1千億程度になる。その20桁と同じ並びが他の場所にある確率は1兆1千億分の1ということです。マウスの全DNAの桁数は30億塩基程度ですから、余裕で同じ場所は無いと言っていいわけです。
[18番染色体に挿入遺伝子は存在するか?]ということを示している若山さんの記者会見での説明資料にある実験3は二つある前後のプライマーの後ろ側は若山さんのコンストラクトの外側にある。つまり挿入遺伝子は18番に有るということはあらかじめ分かっていて、かつ、どの場所にあるかまで正確にわかっていて、その確認をしているPCR実験です。もし場所が分かっていなくて分かっているのが若山さんのコンストラクトの遺伝子配列情報だけだったとしたら、その外側のプライマーはコンストラクト全体がどの染色体のどの場所に入ったのか分からないのですから作れません。
この実験はすべてが分かった後にその確認のために行われた実験だということになるわけです。[18番染色体に挿入遺伝子は存在するか?]という表題も又ミスリーディングなもので、このPCRで18番に有ることを探し当てたのではありません。既にどこに有るかわかっているものを確認しただけです。
実験4はその明らかな証拠で、プライマーBは若山さんのコントラストを内側に含んだ場所に設定してある。プライマーは業者に依頼して作ってもらうわけですが、どこに入っているか分かって無いと頼めません。これはプライマーの内側にワイルドタイプの遺伝子配列があるものがバンドに出ているものです。若山さんのコンストラクトが入っていたらその分長くなりますから、このバンド位置には出ないということです。これも分かったことを確認しているのであって、記者会見の席上で放医研とやらに調べてもらったなんて大げさなものではありませんね。このくらいは自分のところでできますね。どこに有るか分からないときは業種や専門機関に調べてもらう。高価な機械が必要です。この実験3と4はそのようなものでなく、放医研に頼んだとしたら結論が出た後の確認実験に過ぎません。或いはそもそも自分のマウスのGFP挿入位置なんて作成初期に専門業者に出して調べてもらっているはずのものなのですから、情報を全部与えてただ確認実験だけを第三者の立場だと称して行ってもらっただけのものかもしれない。そもそも放医研は研究所としては正式に依頼を受けたことは無いと言ってる。では依頼されたのは誰なんでしょうね。
次は実験6,7です。これも見やすいように再掲します。

この説明図を見て異様に感じるのは実験3、4では若山さんのコンストラクトを探している、もしくは確認しているはずなのに、ここではそのコンストラクトが示されていないということです。その代わりにCAG-GFPが5つ繋がった概念図が示されている。若山さんのコンストラクトの中のCAG-GFPは1つです。何を探しているのか、又、何を確認しているのでしょうかね。
[18番染色体に挿入遺伝子は存在するか?]というタイトルの実験は実質的には18番染色体のどの位置に有るかということまでが分かっている上での確認実験でした。ところがこの実験は違いますね。[15番染色体に挿入遺伝子は存在するか?]というタイトルで、こちらは本当にあるかないかも分かってない。探しているのは若山さんのコンストラクトではありませんね。検査されているサンプルのGFPがどこにあるのかを探している。若山さんのコンストラクトは「僕のマウス」ESの18番にあったがFLSの18番には無かった。しかし、常時蛍光はしているからどこかに何らかのCAG-GFPは入っている。ではどこにあるのかを調べようとしているんですが、CAG-GFPを探しているのですからCAG-GFPの構造の前後をプライマーに取ればいいんですよね。CAG-GFPの構造は分かっていますからその中でプライマー設定するとそれがPCRにかかってきますからCAGがあるということは分かる。でもこれではどこにあるのかは分からない。そもそも常時蛍光しているんですから有ることは分かっていますからそんな検査は無駄です。これを知るにはある一定区分で区切ったたくさんの種類のプライマーを作って、それをワイルドタイプと比較すればいいとど素人でも思いつきますね。GFPが挿入されている断片は長いので上に出てワイルドタイプは下に出る。二本出たらそこにある。ゲノムは全部読まれていて並びは分かっていますからあとは更に厳密に範囲を狭めて行ったらいいですね。
ゲノムウォークという手法があって、挿入遺伝子の構造は分かっているのですからどちらでも片方のプライマーは設計できるわけです。他方がどこに有るかわかってないんですからここにアダプターを付けて取り敢えずの場所の検討をつけた後にそのアダプターに反応した場所を新たなプライマーとする方法がある。以下はタカラバイオのゲノムウォーキングキッドの宣伝の中の図です。
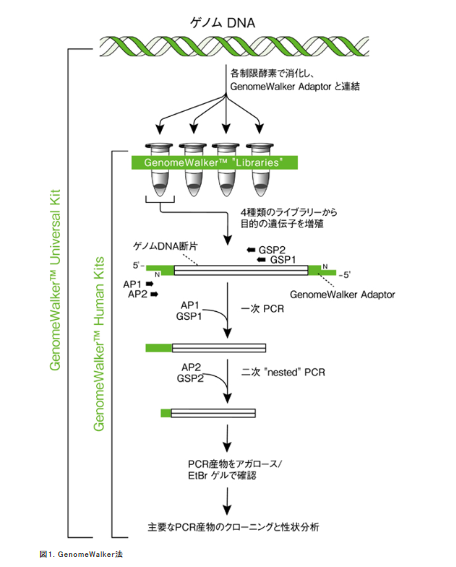
ど素人にはちょっとややこし過ぎて理解できませんが、まあ結論的に言えば場所の特定手法はちゃんとあるということです。業者に頼めば有料でやってもくれる。
ところが、放医研の第三者とやらは間違えたのです。マウスの15番染色体にはCAGは入っていなかったんです。ではこの15番染色体にヘテロで4コピーあったと報告したCAG-GFPは何であったのか。誰でも疑問に思いますね。竹市さんの疑問を貼り付けておきましょう。
>>
2014年7月22 日に訂正版を発表しました。
*ttp://www.riken.jp/pr/topics/2014/20140616_2/
2014年6月16日
CDB に保全されている STAP 関連細胞株に関する検証について
発生・再生科学総合研究センター
センター長 竹市雅俊
背景
STAP 論文では、脾臓から採取された免疫細胞が弱酸性にさらされることにより、多能
性を獲得することを報告した。STAP 細胞から増殖可能な細胞株として STAP 幹細胞が作
製された。STAP 論文に関する疑惑が明らかになった後、山梨大学若山教授は第三者研
究機関に STAP 幹細胞株の DNA 解析を依頼した。その情報は理研と共有され、理研は CDB
に保全されている STAP 関連幹細胞株の解析を進めてきた。
CDB 小保方研 STAP 幹細胞株の検討目的
CDB に保全されている STAP 関連細胞株(STAP 幹細胞、FI-幹細胞株)およびそれらから
作出されたキメラマウスの遺伝子情報を比較解析し、各細胞株間の遺伝子レベルの相違
と起源に関する客観的に検証可能なデータを得ることを目的とする。
CDB で保全されている小保方研細胞株の解析と結果
小保方研細胞株サンプルの遺伝子解析(遺伝的背景および CAG-GFP 挿入部位の確認)
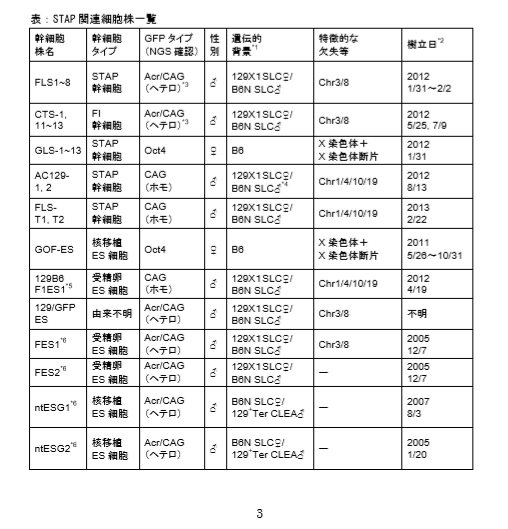
• 小保方研細胞株サンプルのSTAP幹細胞6株に関して、遺伝的背景を6種のSNPマー カーを用いて検査したところ、以下の結果が得られた。GLS-1およびGLS-11につい ては核型の解析も行った。 1. FLS-3およびFLS-4: B6129F1: CAG-GFP, ♂ 2. GLS-1およびGLS-11: B6: oct4-GFP, ♂(核型の解析ではY染色体の一部に欠 失が見られる) 3. AC129-1およびAC129-2: 129B6F1: CAG-GFP ♂ (129 CAG-GFP マウス由来と されたが, 129B6F1由来であることが判明)。
• CDB では、若山氏から、STAP 幹細胞の CAG-GFP 遺伝子挿入位置の情報提供を受け、 上記 STAP 幹細胞株の CAG-GFP 遺伝子挿入部位を検証した。
STAP 幹細胞株 AC129-1 および AC129-2 は、18番染色体 GFP の挿入を持つ若山研 GFP マウスと同じ部位に、 1 コピーの CAG-GFP 遺伝子の挿入を持つことが判明した。かつ、相同染色体の両方に挿入されていることも若山研 GPF マウスと一致した。他方、FLS-3 および FLS-4 に関しては、15番染色体の片方の染色体に GFP 遺伝子が挿入されていることが判明した。また、CAG-GFP 遺伝子は複数コピーがタンデムに並んだ形で挿入されていた。これらの結果は若山研のサンプルの解析結果と一致した。
解析結果に対する見解
1.若山氏が提供されたとされる光るマウス(CAG-GFP 遺伝子保持マウス)から小保方氏が STAP 細胞を作成し、それを若山氏が受け取って樹立した STAP 幹細胞株に関して、保管されていたストックの解析から、CAG-GFP 遺伝子の挿入状況の違いにより、STAP 幹細胞は2種類の異なる遺伝子型のマウス由来であることがわかった。一方は、若山氏が提供した(CAG-GFP を18番染色体にホモで持つ)もの、もう一方は由来不明(CAG-GFP を15番染色体にヘテロで多コピー持つ)のものであった。
2.CAG-GFP を15番染色体にヘテロで持つマウスがどこ由来なのか、そのマウス個体が STAP 細胞から STAP 幹細胞が樹立された時期に若山研(あるいは小保方研)に生存個体として存在していたのかは不明であり、今後、さらなる検証を進める 誰でも15番にどうしてそんなものが見つかったのかを不思議に思いますよね。ここにはCAGはありません。にも関わらずどうして放医研の第三者といわれる人はここにCAGを4コピー見つけたのか。マウスの15番には本来の内在性アクロシン遺伝子があって、そのプロモーター領域もあります。仮に若山さんが岡部マウスのコンストラクトを聞いていてそれを教えていたのだとしましょう。ゲノムウォークするときに既知のプライマーがアクロシンプロモーターになっていた時には15番の内在性遺伝子領域をひっかけた可能性がありますね。でも、ここには4コピーなんて入っていません。4コピーあるのはCAGで3番に入っているのが岡部マウスです。間違えたにしても話がおかしすぎますよね。
(アクロシンプロモーター)放医研の第三者とやらはあくまでもCAGを探していたのだとしましょう。ゲノムウォーキング手法を使うとCAGが5個タンデムに並んだ断片が見つかったのだとしましょう。そして周辺を調べたらそこにアクロシンプロモーターが見つかったので15番だと思ったということですね。ところがそのアクロシンプロモーターは岡部マウスの人工遺伝子につけられていたアクロシンプロモーターだったということですね。
そして後に遠藤氏がそれに気が付いたという話になっているわけです。我々は誰が犯人か知っている。するとこのストーリーが作られたものだということはすぐわかる。その証拠が無いかということですね。
アクロシンマウスは若山さんは自分で維持飼育してますからね。「僕のマウス」B6でなければ、GOFのB6か、岡部マウスB6に決まっている。遠藤さんなんて出てこなくても可能性の中にはすぐ上がってくるものです。そもそも話が白々しいんですよね。あたかも自分は岡部マウスなんて考えもしてなかったなんて、最初のテラトーマからアクロシンが出でいて、このマウスが「僕のマウス」でないことは桂報告書の指摘するところです。FLSの時に「僕のマウス」を渡したということに実験ノートの証拠すらない。
岡部マウスの特徴は以下です。
>>
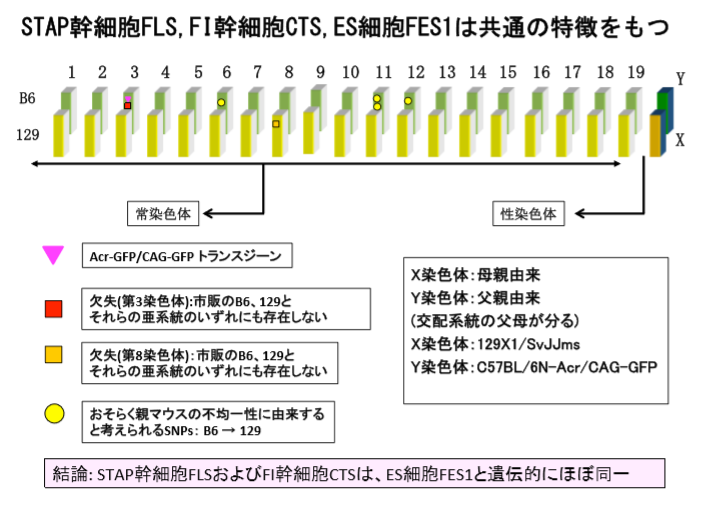
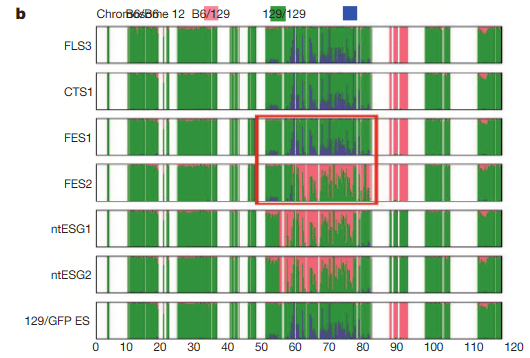
1)Acr-GFP/CAG-GFP 共挿入の位置、コピー数、周囲の塩基配列 表:STAP 幹細胞株一覧に挙げた 12 種類の幹細胞から STAP 幹細胞 FLS-T を除く 11 種 類の幹細胞株、それらの幹細胞が作製された 129 系統、および C57BL/6 系統の NGS によ る全ゲノム解析を行なった。その結果、Acr-GFP/CAG-GFP の共挿入は、STAP 幹細胞 FLS3、 FI 幹細胞 CTS1、そして ES 細胞 FES1 並びに FES2、ntES1 並びに ntES2, および 129/GFP ES の 7 株の第 3 染色体の同一部位に共通に存在することが判明した。また、Acr-GFP が第 3 染色体の片方にのみ挿入されていること(FISH により確認)、Acr-プロモーターの コピー数がどれも約 20 コピーであること、GFP 挿入部位を挟んで第 3 染色体の約 20kb の重複があることと、GFP 挿入部位に隣接して第 4 染色体 20kb 断片の逆向きの挿入があ ることも共通していることが判明した。これらの特徴は、2003 年に CDB 若山研が大阪大 学岡部研より導入した Acr-GFP/CAG-GFP マウスの特徴と完全に一致する。 (4P) ①3番染色体にAcr-GFP/CAG-GFP 共挿入がある。
②アクロシンプロモーターのコピー数は20コピー程度である。
③GFP挿入部位を挟んで約20kbの重複がある。
④GFP 挿入部位に隣接して第 4 染色体 20kb 断片の逆向きの挿入があ る。
⑤この特徴を共有しているのは2003年に岡部研より導入した岡部マウス、FLS3、CTS1、FES1、FES2、ntES-G1、ntES-G2、129/GFP ESである。
無論岡部マウスはホモ、その他はF1なのでヘテロに入っている。
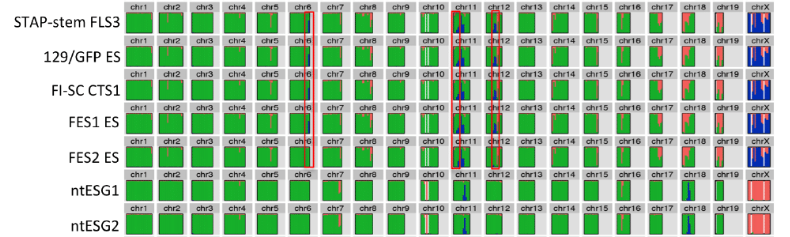
ところで3番染色体には③④の二つの異常がある。しかし以下には欠失だと書かれていて、これが何なのかは上記引用箇所だけでは分からない。何度でも貼り付けましょう。

赤の四角は欠失と書かれていますよね。③④の中腹や交叉乗り換えとは違います。これって何でしょう。
>>
4)第 3 染色体と第 8 染色体の欠失変異
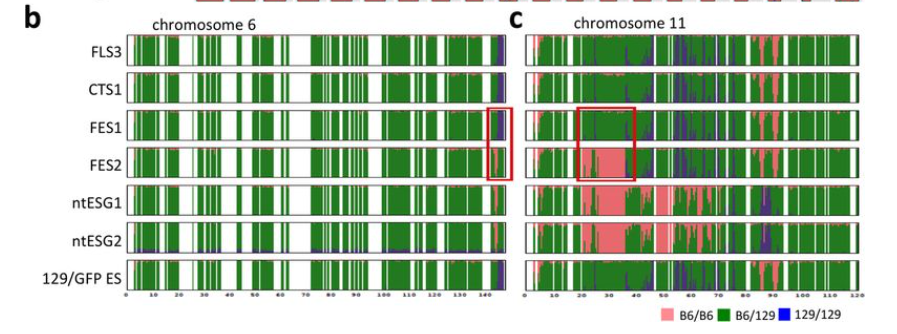
STAP関連11細胞株の全ゲノム解析から、第3染色体の5kbの欠失と第8染色体の17kb の欠失(第8染色体は129系統由来;第3染色体はB6系統由来)が上記STAP幹細胞FLS3、 FI 幹細胞 CTS1、および、ES 細胞 FES1 並びに 129/GFP ES だけに共通に存在することが 判明した。この2箇所の欠失は、STAP 幹細胞 FLS および FI 幹細胞 CTS の全ての株にも 共通に存在することがPCR産物の塩基配列決定により確認された。一方、この両欠失は、 市販の 129 の亜系である 129 x 1/SVJJmsSlc(SLC)と 129+Ter/SvJcl(CLEA)のいずれにも存在しない。また、この第 3 染色体の 5kb の欠失も、市販の B6 の亜系である C57BL/6JJmsSlc (SLC)、C57BL/6NCrSlc (SLC)、C57BL/6J (Charles River)、C57BL/6NCrl (Charles River)、C57BL/6JJcl (CLEA)、C57BL/6NJcl (CLEA)のいずれにも存在しない。 さらに、2010 年に若山研で受精卵凍結された Acr-GFP/CAG-GFP マウスにも存在しなかっ た。 もし、これらの細胞が論文に示されていた(129 x C57BL/6)F1 から作製された株で あるなら、これら 2 個所の欠失の両方、または片方が市販の 129 系統、C57BL/6 系統の いずれかに存在していなければならず、STAP 研究の行なわれた 2 年強という期間でこれ ら 2 個所の欠失が生ずることは考えにくい。従って、この結果は、これら 4 種類の細胞 が、論文に示されていた(129 x C57BL/6)F1 マウスから直接作製された株ではないこ とを明確に示している。(6P)8番は129側ですので今は論じない。B6の3番に別の欠失5kbがあるんですね。そしてそれを共有しているのは今度は、FLS3、CTS1、FES1、129/GFP ESです。岡部マウス、FES2、ntES-G1、ntES-G2には無い。
さて、いよいよ謎のキメラ子です。もう一度貼り付けます。

- 2019/11/07(木) 18:02:54|
- AC129
-
-
| コメント:3
(なぜローザ)清成さんの名前が出たので、少し寄り道しておきましょうかね。
>>
理研CDBの研究室主宰者22名、連名でSTAP論文問題に対する声明を発表
2014年3月14日 19時12分 マイナビニュース
理化学研究所 発生・再生科学総合研究センター(理研CDB)の研究室主宰者22名は3月14日、連名で英科学雑誌「Nature」に掲載されたSTAP細胞に関する論文に関連して生じているさまざまな問題に対する声明を発表した。
今回の声明を出したのは以下の22名の研究室主宰者(声明文記載順)。
・戎家美紀・永樂元次・榎本秀樹・藤原裕展・古田泰秀・花嶋かりな・平谷伊智朗・今井猛・猪股秀彦・北島智也・清成寛・清末優子・倉永英里奈・Raj Ladher・森本充・森下喜弘・西村隆史・Guojun Sheng・柴田達夫・高橋政代・上田泰己・Yu-Chiun Wang
なお、声明の原文は以下のとおり。
今回Nature誌に掲載されたSTAP論文に関連して生じている様々な問題に対し、下記に名を連ねる者は、同じ理研CDBの研究室主宰者として大変深刻に受け止め、憂慮しております。わたしたちは同じ研究者として科学の公正性を回復、担保するためのあらゆる努力を払う所存です。また、理化学研究所における研究活動が社会の信頼無くしては成り立たないことを十分に自覚しております。我々は、社会及び研究者コミュニティーに対して最大限誠実な行動を取ることをお約束すると共に、高い規範の下に研究活動に励み、その成果を社会に還元すべく不断の努力を続けることをここに表明いたします。清成寛さんは、若山さんが協力を断ったために、小保方さんの再現実験でキメラ胚に移植する実験を代わりにしてくれた人です。

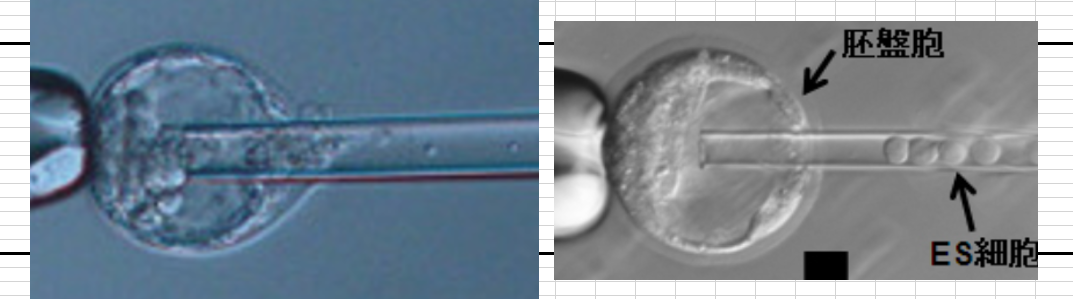
八細胞期胚へのES細胞のインジェクション写真ですね。彼はすべてを分かってると思いますね。彼はES細胞も、そして小保方さんが作ったSTAP酸浴細胞も両方ともインジェクトしたことがあるんです。
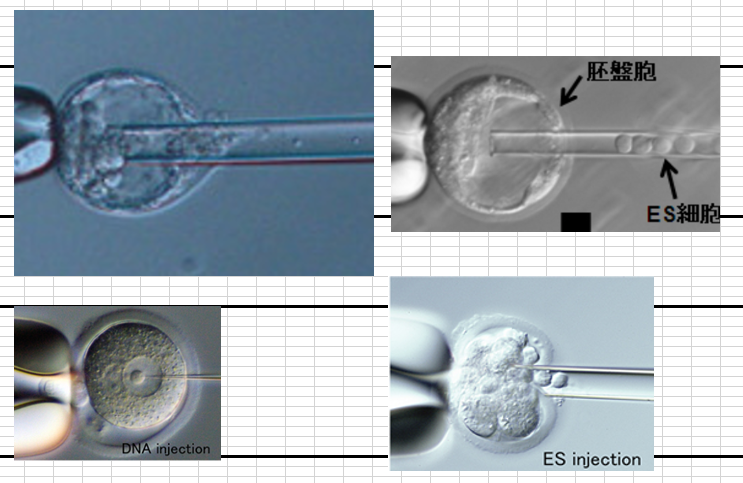
左下のマイクロインジェクションの針は1~2マイクロメーターという細さです。注射する対象物の大きさは移植者が常に意識していることで、ESを混ぜられて分からなかったなどということはありません。笹井さんもそうおっしゃっておられたとおりです。清成さんにES細胞が混ぜられていても分からなかったと思いますかと聞いたら彼はとても困ったでしょうね。記者は誰も聞かなかった。左上の写真はまさに若山さんが小保方さんから渡された細胞をナイフカットしてインジェクトしている当の写真です。細胞塊の中にES細胞が見えますか? 胚盤胞の一番左奥にあるのはリシピエントのインナーセルマスです。ほぼES細胞と同じ大きさですが、そんなものが混じっていて若山さんが気づかないなんてことはありません。この実験は264個の胚盤胞を使って行われているんですよ。清成さんが再現実験で行ったインジェクション操作も244回です。若山さんは64匹のキメラマウスを得たが、清成さんは生まれたマウスの一匹もGFP確認できなかった。
若山さんの作ったキメラマウスは小保方さんの酸浴細胞をナイフカットして移植したキメラ胚から作られたものではありません。彼が小保方酸浴細胞の核を移植したクローン胚から作ったntES由来のキメラなんです。その意味で小保方さんは無実です。キメラは実際にできています。なぜできたのかを説明できなければなりません。小保方さんが出所不明の太田ESと学生にもらったGOF ESとFLSのコントロールとして若山さんから貰っていた「僕のマウス」ESを使って、若山さんを騙してキメラマウスを作らせたというのが桂報告書とBCA報告書の結論です。
出来ているキメラ、幹細胞、テラトーマ=太田ES、GOF ES、「僕のマウス」ESこれが両報告書の結論です。この結論が間違っているのです。重大な間違いなのでこのまま放置してもらっては困る。従ってOoboeさんのパートナー氏が検察に調査依頼をされているのです。GLという名の幹細胞が2011/11/25に培養開始されて樹立されている。この細胞は調べられていませんが、できているのですから、両報告書の論理では学生のntESを混ぜて小保方さんが若山さんに渡したからできたということになる。後のGLSは学生のGOF ESを使ったと言ってるのですから、この時も学生のGOF ESを使っていないとおかしい。ところが2011/12/27に移植したテラトーマからはアクロシンCAGが出たので、両報告書は太田ESが使われていると結論した。テラトーマはGOFマウスがドナーになっている。小保方さんがESを使うなら学生のGOF ESを使うはずなのに、出てきたのは太田ESだという矛盾に両報告書は真摯に向き合わなければならない。
更に、AC129とFLS-Tシリーズの幹細胞から「僕のマウス」ESが出たことは、小保方さんの捏造ではないということが確認されてしまうと、今度は誰が何のために行ったのかという、新たに深刻な問題を発生させる。
①日本細胞生物学会から竹市所長に送られたメールの連名者達
②大隅理事長
③11Jigen
④世界展望
⑤東北大学黒田教授
⑥放医研の協力者
⑦京都大学太田氏
⑧遠藤氏
⑨吉村氏
⑩岡部氏
⑪日経サイエンス記者
⑫毎新聞社記者
⑬理研から小保方さんの実験ノートをNHKに全コピー違法流出させた者
⑭細胞リストを毎日新聞に違法流出させた者
⑮ノフラー氏
⑯李氏
⑰感想氏
⑱小保方バッシングを行い続けてきた書き込み者達(日経サイエンス記者の知り合いと自分で言ってるものもいる)
等々、たくさんの人々が調査対象者になるでしょうね。
最初に小保方さんの高校時代の個人的な悪意ある噂話を流布した週刊誌があったが、誰に頼まれたのかを調査すべきでしょうね。吉村さんはネットに流出していたと称しながらネイチャー査読文を更に拡散した犯人で、しかもTCR再構成の有り方に関して素人に向けてスピン解説をしている。加えて後に遠藤氏論文の中で謝辞の対象者になっている。若山・遠藤・吉村というラインがありますね。一方でNHKには若山さん自らが番組出演している。データを流したのは彼自身ですね。番組にはなぜか山梨大の若山研に大日向氏が出演してPCRの小芝居をしている。いつ山梨に来たのか。若山さんは共同著者なので仮に小保方さんが捏造したのだったら共著者間と理研内で話し合って解決に向かわせないといけない立場です。何をしているのでしょうかね。
一つには理研が大々的に記者会見をして世間に宣伝してしまったということがある。こんな大成功の後ですから引っ込みがつかないというようなこともあるかもしれませんが、そもそも若山さんはただ騙されただけなんでしょう。だったら理研に行って、理研上層部のいる前で、自分は小保方さんに騙されたんだと言えばいいことです。罪は無いではありませんか。そして小保方さんが認めたら、理研はあれは間違いだったと世間に謝ったら済むことです。ところが彼はそうしなかった。マスコミを巻き込みながら小保方さんに騙されたと騒いだのです。そして当の小保方さんは今に至るまで、自分はESを若山さんに渡したことは無いと主張しているままです。どうしてこんなことになったのか。若山さんが理研に行って、皆の居る前で堂々と小保方さんと対峙しなかったからではありませんか。最初の成功時に渡したマウスは何だったのか、「僕のマウス」ではなかったじゃないか。どうしてFLSの時の話から始めなければならないんだい。半分しかGFPが来なかったなんて話は不要でしょ。最初はGFPはヘテロじゃないか。皆の居る前で捏造したと言ってる相手と対峙すべきでしょ。ナイフカットで胚盤胞注入した時にどうしてわからなかったのと笹井さんに聞かれて困るようなことでもあるのか。なんでも本当のことを言って説明すればいいだけじゃないか。君は一体何をしているんだい。
理研も又どうして警察を入れなかったのか。公務員は犯罪を知ったら必ず警察に届けるということになってる。研究倫理の問題ではない。NHKに小保方さんの実験ノートの全コピーが流出した時点で既に犯罪です。公務員は誰でもまず警察に届けなければならない。理研はみなし公務員の義務を怠っている。マスコミ業も惨状のようですね。
お前たちは何をやってるんだということですよ。一般国民は飽きれてるぜ。自動車や工作機械や薬品や発電機やコンピューターを作っている製造業に従事している社員も、ダムやビルや住宅を作っている土建業の人々も、世界各地から物質を調達している商社マンたちも、金融機関に勤めている人々も、卸でも小売りでも流通産業に従事しているものや、JRやANAや宅急便の運輸業に従事している人々も、医者や看護師や弁護士や公認会計や、有りとあらゆる大人の日本国民はお前たちのユルフンに呆れている。無能だね。永遠にぬるま湯に浸かってせいぜいナガイキセイヤ。
(さて、戻って、なぜローザ)
若山研で使われたことのある129マウスは太田氏が論文で使った129/Sv-ter、そして129/Sv-X1、更にロックフェラー大学で作ったB6のGFPホモマウスからGFPだけを移し替えた129/Sv-X1GFPホモがある。
太田氏は129/Sv-terと岡部マウスとのF1を使って2005年の論文の実験を行った。論文の掲載経緯は以下です。
Biology of Reproduction, Volume 73, Issue 3, 1 September 2005, Pages 390–395, https://doi.org/10.1095/biolreprod.105.041673
Published:
1-Sep-05
Article history
Received:
8-Mar-05
Revision Received:
29-Mar-05
Accepted:
26-Apr-05問題になったFES1とFES2の凍結日は2005/12/7です。論文は3月に提出されていますから実験は当然もう終わっています。ntESG2の凍結日は2005/1/20です。番号の若いntESG1の凍結日は2007/8/3ですが、これは2008年の論文があって、ここで2005年時の凍結細胞を解凍してもう一度使っている。番号はこの時に振り直されているのではないかと推定される。2008年論文の掲載経緯は以下です。
Biology of Reproduction, Volume 79, Issue 3, 1 September 2008, Pages 486–492, https://doi.org/10.1095/biolreprod.107.067116
Published:
1-Sep-08
Article history
Received:
12-Dec-07
Revision Received:
9-Jan-08
Accepted:
25-Apr-08この太田氏の二つの論文で作られたntESは129/Sv-ter x 岡部B6なんです。論文にちゃんと書いてあります。岡部B6というのは無論問題のアクロシンGFPの共挿入マウスですね。
ところが、若山氏が京都大学の太田氏から入手して理研にも最終的に提出されたはずのサンプルは論文と同じくラベルも129/Sv-ter x 岡部B6だっんですが、なんと調査した結果は親の雌雄が逆になっていた。つまり岡部B6 x 129/Sv-ter になっていた。
加えて驚くべきことに桂調査チームはこの事実をスルーしたのです。提出されたサンプルの中身が論文とラベルと異なっているにも関わらず、中身の入れ替えを問題にすることなく、同時提出されているFES1とFES2を証拠採用しているのです。
>>
他方、同じ Acr-GFP/CAG-GFP の挿入を持つ ES 細胞 ntESG1、および ntESG2 の X 染色体は C57BL/6 であることが判明したことから、調 査対象の STAP 幹細胞 FLS3、FI 幹細胞 CTS1 と性染色体の構成が異なるため、これ らは比較解析の対照から除外された。 (5P)頭大丈夫か?ユルフンかい?他方、ntESG2は実験終了後早々の2005/1/20には凍結されていて、その後論文の提出作業に入る。FES1とFES2は約1年後の2005/12/7の凍結です。何の目的もなく作ったと太田さんは日経サイエンスに証言している。実験には使っていないから作ってすぐに凍結しているはずです。ところがこの129はterではなく、X1なんです。岡部マウスとのF1の受精卵ESなんです。今まで使っていて飼育しているF1ではなく、又一から交配し直して受精卵ESを作ったという。ntESではありません。何のためにこんなものをつくったのか。本人は何の目的もなく作ったと言っていて、しかも日経サイエンスにはterで作ったと記憶していると語っている。そちらの方が普通でしょう。X1で作るときは交配からやりなおさないといけない。
>>
太田氏によると、このES細胞2株は、核移植ES細胞を作ったのと同じころ、たまたま作ったES細胞だという。結局研究に使うこともなく、2010年3月に他大学に転出する際にすべて持って出たと思っていた。
太田氏の記憶では母マウスはクレア社の茶色い129+Terマウスだったが、実際には129X1の白マウスだった。(53P)GLSの遺伝子異常の件から我々は既に若山さんのサンプルの入れ替えを立証しています。このFESでもサンプルは入れ替えられているのです。薄茶のマウスと白毛のマウスの違いを覚えてないいうことはあり得ないことです。受精卵ESを作ったことは覚えているので、論文の実験では薄茶を使っていたことは明確で、その後新たに白毛を交配させて受精卵ESを作ったのなら覚えていますよ。その記憶がないというのは以前の実験で使っていた維持飼育していたF1を使ったということです。若山さんはそれを取り寄せて、中身をFLSに取り換えた。
BCA報告のSNPs解析結果です。FES1の中身は入れ替えられていてFLS3ですから一致は少しも不思議でない。129GFP ESはそもそも小保方さんが使っていたFLSですからこれも当たり前の結果です。ただし、このサンプルに関して小保方さんも含めて全員が知らないと言ったという証言が確認されていますが、小保方さんに対してどういう聞き方をしたかですね。後にサンプルの中身に関して答えているところでは129GFP ESはFLSのことになります。それを知らないと答えたのはいつの時点での話なのか、又どういう聞き方をしたのかによる。小保方さんは自分がどういう目に遭っているのかを知らないのです。そしてラボに迷惑もかけられないと考えている。しかも桂調査チームもまさかntES化実験だった可能性なんかには気づいてもいないし、若山さんの嘘の動機にも気づいていない。
ntESG1とG2はラベルと中身が違っているものです。そのntESとFES2の一部のSNPs分布が重なっている部分がある。中身の入れ替え時にきれいに洗いだされていないのではないかとも疑われるところです。和モガさんが既に指摘していますね。ただ、この場合は両方が混じるので又違うパターンになるのではないかとも疑義されます。
いずれにせよ、FES1も2もX1ですから太田さんの作ったものではない。またntESG1もG2も太田さんの二本の論文の細胞とは違います。親の雌雄が違っている。むしろラベルの記載が論文通りなのが中身の入れ替えを証していると疑義される。
BCA報告書にはあからさまな間違いが書いてあります。
>>
These STAP cell lines were then compared with four ES cell lines—FES1, FES2, and
two nuclear transfer ES lines (ntESG1 and ntESG2) (ref. 9)—established from crossing the Acr/cag-GFP mouse strain with 129 mice in the Wakayama laboratory in 2005 (Extended Data Fig. 1a and Extended Data Table 1).
(ref. 9)というのは以下ですね。
>>
9. Ohta, H. & Wakayama,T.
Generation of normal progeny by intracytoplasmic sperm injection following grafting of testicular tissue from cloned mice that died postnatally. Biol. Reprod. 73, 390–395 (2005).
この論文で使われているF1はB6N SLC♀/ 129+Ter CLEA♂ではありません。親の雌雄が逆でチューブのラベルに書いてある通りの、ntESG1:129B6F1G1、ntESG2: 129B6F1G2 です。BCA報告は論文もちゃんと読んでない。しかも、ラベルと中身が違ってるのにそれをスルーした。こんな解析データをつけて何の意味があるのでしょうかね。中身は太田氏の細胞ではない、にも関わらず太田氏の細胞だと言ってる。どうしたのだ。アンポンタンなのか。ユルフンなのか?この論文には明確にメスが129だと書いてありますが、今現在コピペできないようにされていますね。以前は山梨大の若山研文献にもありましたが今外されている。このこと自体が若山さん犯人を証明していると言ってもいいでしょう。論文はOoboeさんとパートナー氏、そして相沢さんも持っている。無論、私もです。
日本語訳なら大丈夫かな。
>>
マウス
未成熟雄B6C3F1、成体雌ICRおよび成体雌BDF1マウスは、静岡研究所動物センター(Hamamatsu、Japan)から購入した。メス129 / Sv-terおよびオスICRヌードマウスは、CLEA Japan、Inc.およびCharles River Japan、Inc.からそれぞれ購入した。アクロシン/ eGFP(Acr3-EGFP)[34]およびpCX-eGFP [35]トランスジーン(C57BL / 6TgN(acro / act-EGFP)OsbC3-N01-FJ002 [36])の両方を有する緑色蛍光タンパク質(GFP)トランスジェニックマウスは、 )は、大阪大学(Osaka、Osaka、Japan;すべての菌株の名称は元の研究の名称である)のOkabe博士によって親切に提供された。 GFP導入遺伝子を有する129B6F1マウスを作製するために、メス129 / Sv-terマウスをC57BL / 6 GFPトランスジェニックマウスの雄と交配させ、核移植のためのドナーとしてこれらの交配の子孫を使用した。すべての動物実験は実験動物のケアと使用のガイドラインに準拠しており、理研神戸研究所の実験動物実験機関委員会の承認を受けています。はは。
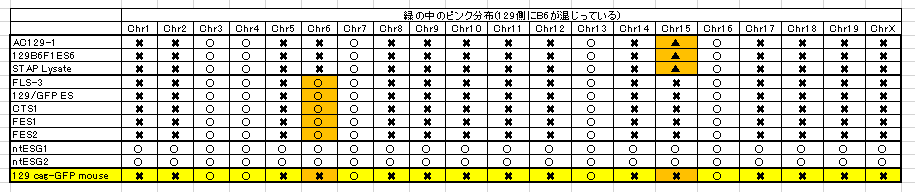
(驚くべき虚偽)以下は桂報告書のスライド版にあるSNPs解析表です。
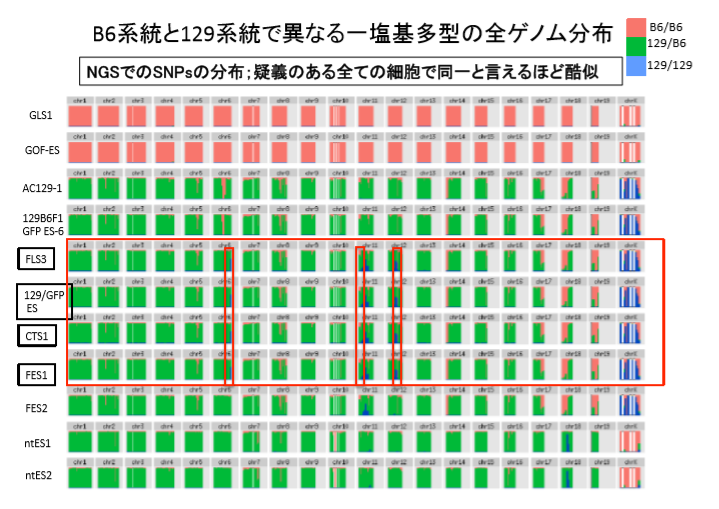
こちらはBCA報告に添付されているSNPs解析表です。
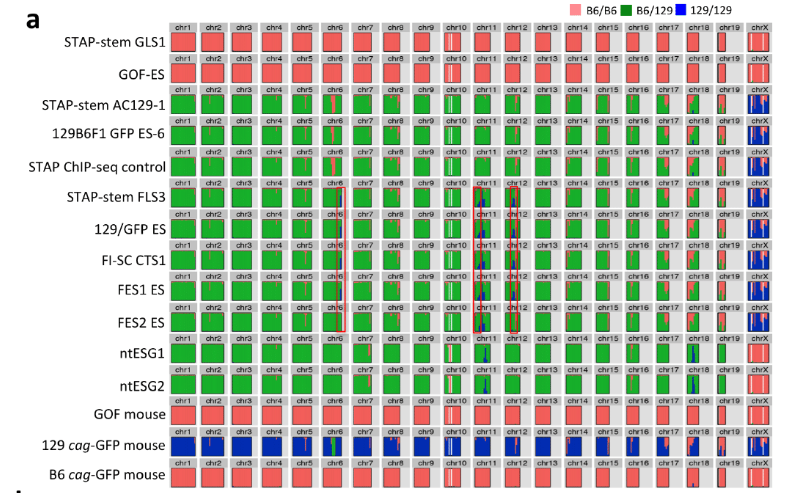
どこが違うか確認されてください。
上部のGOFマウス関係は別として一番下に129 cag-GFP mouseとcag-GFP mouseが置かれている。これは言うまでもありませんが「僕のマウス」の両親です。AC129を巡る問題1で説明したとおり、若山さんはロックフェラー大学でB6のCAGホモマウスを作った。後の調査で18番染色体にGFPが入っていることが分かっています。その後彼は理研に移籍してからこのB6cagホモマウスのGFPをそのまま129/Sv-X1に移し替える実験を長期にわたって行った。一度B6GFPホモマウスと市販の129を交配させてF1を作る。F1ですから今度はGFPがヘテロに入っている。このF1に市販の129をかけ戻す。戻し交配という。この時常にGFPの光っている子供に対してのみ戻し交配を続ける。これを20継代続けるとGFPのみが最初のB6の遺伝子座に残る129の近交系マウスに戻る。その後にヘテロ同士の兄妹交配からホモの子供を作るんです。何度掛け合わせてもすべての子供からGFPが出るとホモになっていることが確認できる。20継代は大体4年位かかる。これでGFP以外は完全に近交系マウスになる。しかし、直前のグラフを見ると129は近交系になってませんよね。
近交系になってないという言い方は誤解が有るかもしれません。129特異的SNPsというのが分かっていて、こういう分析をすると全部ブルーになるマウスがあるというのが前提です。ピンクが混じっていてはおかしいわけです。上の表の若山さんの129はピンクが混じっています。でもこのままで20継代続けたら近交系マウスになります。ただし129/Svマウスとは言えないということです。これは近交系マウスになっていたとしても若山さんマウスということです。ただ、この1件だけでは近交系マウスとして固定しているかどうかは分からない。たくさん抜き出して全部同じパターンになっていたら若山129マウスとして近交系になっているということですが、どうも他の交配結果からして、近交系にもなりきっていませんね。遺伝子配列にヘテロのままになってるいろんなマウスか混じっているようです。つまりマウスコンタミして20継代まで経過していないということです。ここに例示されているパターンのものだけでないということですね。他のパターンのマウスが混じっている。だからこそ129B6129BF1ESも1と6の二種のSNPsパターンに分かれたということです。たまたま使われたマウスが2種だけで実際にはもっと違ったパターンのマウスも入っている可能性がある。
BCA報告は129の全部ブルーの市販のマウスのSNPsパターンを示していません。なぜでしょうね。純粋に青とピンクが半々になったらグリーンで示されることになる。ここにピンクが入っているということは全部ブルーであるはずの129側にピンクが入っているということです。若山さんの129のブルーの中にピンクが入っているパターンがそのまま他の細胞のグリーンの中にピンクで残っているものを表にしてみましょう。
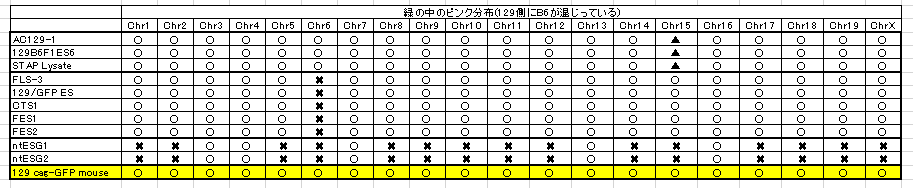
〇は若山マウスと同じB6混入があるという意味です。✖は若山さんのB6混入パターンと違うという意味で、▲は又✖とも違うパターンという意味です。〇という表示はそれが正しいパターンだという意味に取られやすいのですが、若山さんの129は全然正しくないものです。本当は正しいブルーだけのパターンのものをコントロールとしてつけるべきでしょうが、既述しているようにそれがつけられていませんから、代わりにntESG1G2を基準にしてそれを〇表示してみましょう。ntESG1G2は太田さんの論文とチューブラベル表示のものではなくて中身は親の雌雄の違う別の細胞です。でも使われている129+terは市販のもので、若山129マウスではありませんね。報告書が確認しています。こちらは緑の中にほとんどピンクが無いということを確認してください。129+terにはB6のコンタミがほとんどないんです。少しだけあるのはもともとのジャクソン研究所でのコンタミではないかと疑われる。というのもこちらにあるピンクはX1にもありますからね。共通している。以下にntESを〇にした表を示します。

6番と15番染色体以外の染色体に関するB6のコンタミパターンは全部若山129マウスと同じだということに気づけます。市販の129X1/Svにはこういうコンタミは無いはずですね。むしろ129+ter/Svと同じはずです。というのも129X1/Svと129+ter/SvのSNPsパターンがそんなに違っているのなら、こういう比較そのものが意味をなさなくなる。報告書は129とB6のSNPsパターンを比較しているだけです。129の中の亜種で違うSNPsなんて使うわけがありませんね。
上の表は他のF1細胞の129がすべて若山マウスであった可能性を暗示しています。つまり記者会見の情報から推測するに18番に1コピーのCAGが入っているということです。このことは放医研が最初GFP位置を間違えたことに端を発してうやむやになったままで調べられていませんね。
6番と15番のコンタミパターンが違っているというのは若山129マウスの中が不均一な遺伝子構成を持ったマウスでコロニー形成されていた。つまり近交系マウス化できていなかったという原因に求められるのではないでしょうか。
問題はGFPです。若山129マウスはCAG-GFPホモです。F1マウスの3番にヘテロでAcr-CAGが、18番にヘテロでCAGが入っていなければならない。
この問題は一度マクズハラさんのブログでアルイミオウジ氏の指摘として論じられている。
radiation-japan.info/journal/science/stem_sell/stap-cell-case.html

彼もその原因は分かってないから警察に呼び掛けているわけです。
では129のGFPの問題に入る前に、先にB6の方を確認して置きましょう。GOFマウスとGOFESはいいですね。ほぼ全部ピンクです。若山さんがロックフェラー大学で作ったCAGホモのB6マウスもほぼ全部ピンクです。129は混じってませんね。18番にわずかに有りますけどね。ちょっと原因が分かりません。ただもっと大きな問題が6番、11番、12番、そしてこの18番にありますからね。
ここには岡部マウスのSNPsパターンがつけられていない。
①岡部マウスは大阪大学の岡部さんのところにある。
②そしてその後太田さんが大阪大学から岡部マウスを理研若山研に持ち込んだ。そしてすぐに2005年の論文でそのマウスを使いました。そのサンプルは残っていますが、報告書の分析では使われていない。
③2005年の実験1年弱後に何の目的も無く岡部マウスとの受精卵ESが太田さんによって作られた。
④報告書が分析したのは論文とラベルの記載とは異なった親の雌雄の違ったサンプルの中身でした。でも使われているB6は岡部マウスだと分かっている。この時期の岡部マウスが一つあるということです。
管理場所と時期の違った四種類の岡部マウスがあるわけです。129とB6のF1であるからには全てのSNPsパターンは緑が原則です。そこにブルーが入っているということはB6側に129が入っているということです。GOFのマウスとロックフェラー大のCAGホモB6はほぼ全部ピンクでした。緑の中にブルーがはっきり出ている細胞のB6は岡部マウスなんです。これをどうやって調べるか。
まずntESG1,G2の129+terにはB6のコンタミがほとんどありませんでしたね。ほぼ全部ブルーです。ここにその129+terと岡部マウスが掛け合わされた結果がある。④の結果ですね。緑の中にブルーのある部分は④の岡部マウスB6にコンタミしている129のSNPsなんです。なぜなら、129+terが全部ブルーだと分かっているからです。なぜ全部ブルーだと分かったかというと、ntESG1,G2は全部グリーンになっていてB6の混入が無いからです。でもブルーの混入はある。これが④の岡部マウスに混入した129のSNPsなんです。それを基準にしてみましょう。
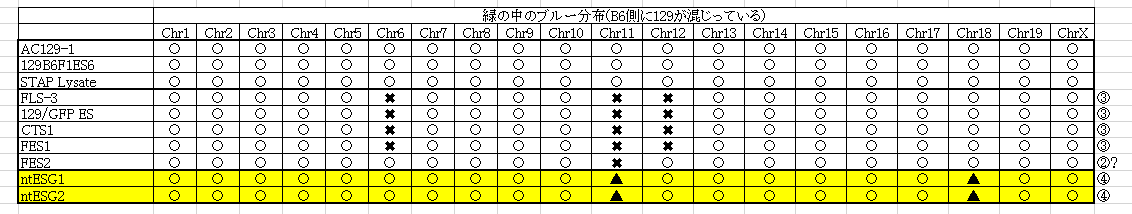
黄色の場所の▲が岡部B6マウスを若山研で自家飼育している過程で入り込んだ129です。AC129-1、129B6F1ES6、STAP Lysateの三つはいいですね。相手のB6はロックフェラー大で作った若山さんのCAGホモマウスで、解析をうけていますね。129のコンタミはありません。問題になっているのはあくまでも岡部マウスなんです。再掲しましょう。
まずntESG1,G2のコンタミ場所は他のいずれとも共通していないということを確認しましょう。この岡部マウスが一つあるということです。④の分ですね。太田さんの論文のものではありませんよ。どこかから出てきたものです。今、Ooboe さんのパートナー氏が検察に調査依頼されています。
次にFLS-3、129/GFP ES、CTS1、FES1の4つの細胞株作成時に使われた岡部マウスがある。この4つは同じ場所に同じパターンのコンタミがあって、同じ岡部マウスからの作製のようですね。そして上記ntESG1,G2とは全く違う場所にあるということを確認しましょう。
そして、最後にFES2に使われた岡部マウスがあって、これは6番、12番にはなくて、11番のみFES1と共通している。時期的にはFES2の岡部マウスが使われたあと更に6番、12番のコンタミが加わっているので、作られた順番はFES2が先で、その後にFES1が作られていることになる。そしてntESG1,G2の岡部マウスは飼育コロニーの違うものとしか考えられませんね。
129も岡部マウスのB6も自家飼育している間にマウスコンタミが発生している。これが遺伝子調査結果にどう反映しているのかはとてもむつかしい問題です。逆にマウスコンタミが無ければおよそ遺伝子解析なんてできなかったでしょうね。でもあったからいろいろと問題が分かったが、そのありようがどうであったか。とても錯綜しています。
そもそも小保方多能性細胞の探求にとって若山研のマウス管理は適合的ではありませんでしたね。Oct4遺伝子を発見するのにGFPを使って大丈夫だなんて酸浴させて死にそうになってる細胞で適合的な実験方法でしょうかね。ジャームライントランスミッション実験でGFPを確認するのにそれだけマウスコンタミのあったコロニーの中で交配するなんて適合的ではないでしょう。でも、それは若山研の責任ではありませんね。若山さんのクローン胚の探求にとって、マウスコンタミは大した問題ではなかったはずですよね。こういうことになったのは偶然に過ぎない。バカンティ氏側がキメラを作ってくれと頼んできた流れです。小保方さんの細胞研究をするためのラボではありません。でもこのラボでOct4-GFPが大量に光った。誰が悪いのでしょうかね。
リクルートの話とはこれは別なんですね。多分科学的にはこちらが問題だった。
(そもそもAC129の問題とは何なのか)AC129は129-CAG-GFPマウスから作られたと言われているのにこの幹細胞からは「僕のマウス」ESが出てきた。これは中身の入れ替えで説明できる。でもSTAP lysateはどうなのか。これも中身は「僕のマウス」ESだった。桂報告書はサンプルを調整してGRASに提出したのは小保方さんだったと主張している。でも一つのことを忘れている。若山さんはリトラクション理由に何と書いているか。
>>
(4) In Fig. 4b of the Letter, STAP cell and ES cell are wrongly labelled in a reverse manner.
 これが本当ならStap Lysateと書かれているサンプルはESなんですね。こちらを分析して「僕のマウス」ESが出るのは当たり前です。まさかそんなことしてないですよね。
これが本当ならStap Lysateと書かれているサンプルはESなんですね。こちらを分析して「僕のマウス」ESが出るのは当たり前です。まさかそんなことしてないですよね。
大丈夫か?
>>
3)STAP 細胞由来 ChIP-seq (input)サンプルは 129B6 F1ES1 から取得された
(調査結果) 小保方氏が CDB ゲノム資源解析ユニット(以下「GRAS」という)に持参し残されてい た STAP 細胞由来 ChIP-seq (input)サンプルを再度 NGS 解析した結果、STAP 細胞由来と される ChIP-seq input データは CAG-GFP の挿入を持つ 129xB6 へテロ系統由来の細胞か ら取得されたものと判明した。さらに SNPs の解析、特異的な欠失変異の解析により(2 -3-1-1(d)参照のこと)CAG-GFP が挿入された 129B6 F1ES1 とほぼ同一細胞由来の データであることが明らかとなった。 ES Lysateの方は調べたのか? リトラクション日付は (03 July 2014)よ。両方調べて初めて若山さんのリトラクション理由が本当か嘘かが分かる。又両方の結果を記載して初めて報告書がどちらのサンプルが「僕のマウス」ESだったとしているのかが分かる。このままだと、どちらが「僕のマウス」ESだったのかが分からない。その可能性として4つある。
①STAP LysateとラベルされているデータとES Lysateとラベルされているデータのどちらも「僕のマウス」ESだった。
②STAP Lysateとラベルされているデータだけを調べてそれが「僕のマウス」ESだった。
③リトラクション理由書に取り違えがあると書かれていたのでES Lysateと記載されている方がSTAP Lysateだと見做してそれのみを分析したら「僕のマウス」ESだった。
④そもそもSTAP Lysateとラベルされているデータしかなかった。
①ならそう書くことによって129/Sv-CAG-GFPのデータは無いので Fig.ure 4-b は捏造であると報告しなければならない。
②の場合はリトラクション理由書に書かれている通りES Lysateが129/Sv-CAG-GFPである可能性があるので、それを調べないままにSTAP Lysateが「僕のマウス」ESだと書いた桂報告書自体が捏造だとされなければならない。
③の場合は、ではSTAP Lysate を調べてないことによってリトラクション理由書が虚偽記載されているということを確認しないまま報告書を書いたということになり、これも捏造報告書だということになる。
④の場合はリトラクション理由書に取り違えがあると書かれていることに気づいていない杜撰な報告書だということになる。
調査報告に瑕疵があるということは明白ですね。取り下げてもらうか、訂正してもらわないといけない。我々は若山さんが犯人だと考えているので、リトラクション理由には強い疑義を抱いている。どうしてあんなに似ている Figure 4-b の取り違えに気づけるのか。手伝った人が居るのではないかと。また、取り違えが本当なら、リトラクション理由書はむしろ桂調査の結果を否定することになるのはどうしてかと。
リトラクション理由書が先に提出されている。その後にGRASに残されていたデータが再解析された。若山さんはデータが残されていたことは予期してなかったと思われる。しかし、STAP Lysateを記載通りに調べたら、中身がESであったということになると、取り違えは本当であったということになる。ところが、ではどうして調査チームはES表記されている方を調べなかったのか。そもそも我々は試料が残されていたということ自体を疑うもので、調査チームの中に犯罪者が居るのではないかと疑義している。資料が残されていたということは記者会見の席上で伊藤氏が証言している。その時の彼の表情については既述しているとおりである。
AC129を巡る問題1で述べたことを再掲しましょう。
**********
>>
(4) In Fig. 4b of the Letter, STAP cell and ES cell are wrongly labelled in a reverse manner.
Letter Figure 4-bは以下です。よくこの程度の粗雑な簡略図でSTAPとESの取り違えに気づけたものです。誰かラボ内に手伝った人でもいて、元データを持っていた人がいたことが推察されます。

**********
2年前の生データがSTAP細胞だけ冷凍されて残っているというのも変で、残っているならこの時の試料の全てのはずですが、記者会見では伊藤氏が口頭で記者の質問に答えて、この時の分析の試料がGRASにたまたま残っていたのだと証言しただけで、他の試料の有無に関しての更なる追及も無かった。又その保管に関して文書で追加説明があったということもない。伊藤さんはBCA報告の著者の一人です。
>>
Daijiro Konno, Takeya Kasukawa, Kosuke Hashimoto, Takehiko Itoh, Taeko Suetsugu, Ikuo Miura, Shigeharu Wakana, Piero Carninci & Fumio Matsuzaki
桂会見での古田女史の質問に対する答えは以下でした。言い終わった後の彼のしぐさと表情は精神分析学の研究対象に好例かもしれない。(*ttps://www.youtube.com/watch?v=M9oJGioHvIQ 1:15:00から)
>>
ただ、唯一やったのが、あ、GRASの方に残っていたChIP seq のinput 、しかもSTAP細胞のインプットはDNAとして残っていたので、それが10ページバワーポイントの、えーと、ところに書いてある、3番のアイテマイズして書いてあるところですけれども、これに関しては、えーと、30倍になるだけ、他のゲノムと同量となるまで読んでいます。そして、読んだ結果、この、同一であるということを、ま、認定したということで、それ以外に、それ以外に関しては、ま、残っていませんのでやってない、やってません。
なぜ、たくさんある中の、唯一、STAP ChIP lysateだけが残されていたのか、どういう形で唯一残されていたのか、誰かの恣意で残されていたのか、それともGRASには残し方に関する規則は無いのか。裁判だったらまず最初に検討されるもので、ここにちゃんとした答えが無いと証拠採用はされない。
**********
①唯一やったのが
②STAP細胞のインプットはDNAとして残っていた
③読んだ結果、この、同一であるということを、ま、認定したということ
④それ以外に、それ以外に関しては、ま、残っていませんのでやってない、やってません。
つまり、STAP Lysateと書いてあるDNAのインプット試料だけがあったということです。そしてこの記載事項は2014/7/3に提出さけているリトラクション理由書の記載が本当なら、小保方さんが論文の図表として採用するときに間違えたか、そもそもGRASに提出するときに既に記載を間違えているかのどちらかです。ただ、リトラクション理由書は試料が残っていたということを知らなかった可能性が高い。若山さんは何をもって取り違えに気づいたのか。あのレター論文の図だけを見て取り違えに気づけるというのは難しいのではないか。何か、別の根拠があるはずです。
図だけを見て気づけるのはNanogの発現量です。ただこれがどちらがどうといえるためには元の解析データを若山さんが持っているのでなければならない。この場合は元のデータには提出時の記載事項通りに結果が知らされていることになる。若山さんは小保方さんの図表とGRASの結果を比較してNanogの発現量から図表は結果を取り違えていると主張したことになる。この場合、GRASのSTAP Lysateと書かれているデータが図表のESだということになる。Nanogの発現量の小さい方です。これが「僕のマウス」ESだったということになる。
もう一つの可能性はそもそも小保方さん自身がSTAP細胞を作ったのちにGRASにES細胞と一緒に提出するときに記載を間違えた場合です。この場合はGRASは記載されたとおりにデータを渡すので、そもそものデータが逆になっていることになるが、若山さんが図表とGRASの間での取り違えに気づくことも無いでしょう。
明確なのは小保方さんがSTAP細胞と記載してGRASに提出した細胞は「僕のマウス」ESだということです。少なくともSTAP細胞は小保方さんが作るので、これが「僕のマウス」ESであるためには提出記載の取り違えか、或は、渡されたマウスが「僕のマウス」だったかしかありません。取り違えはES記載された試料が残ってないので確認できない。渡されたマウスが「僕のマウス」だったのなら129/Sv-CAG-GFPマウスの実験なんて無かったということになる。
我々は若山さんが嘘をついていることを知っている。AC129の問題は彼がここで何をしたかを解明することです。
取り敢えず、 Fig.ure 4-bに関して登録されているデータを書きに提示しましょう。
ChIP-Seq
①SRR1171553 小保方 CD45 positive Cells:ChIPSeq.H3K27me3 derived from spleen Oct3/4::gfp C57BL/6 GOF
②SRR1171554 小保方 CD45 positive Cells:ChIPSeq.H3K4me3 derived from spleen Oct3/4::gfp C57BL/6 GOF
③SRR1171555 小保方 CD45 positive Cells:ChIPSeq.input derived from spleen Oct3/4::gfp C57BL/6 GOF
④SRR1171562 若山 Embryonic Stem Cells:ChIPSeq.H3K27me3 C57BL/6x129/Sv
⑤SRR1171563 若山 Embryonic Stem Cells:ChIPSeq.H3K4me3 C57BL/6x129/Sv
⑥SRR1171564 若山 Embryonic Stem Cells:ChIPSeq.input C57BL/6x129/Sv
⑦SRR1171567 若山 FI-SC(Fgf Induced Stem Cells):ChIPSeq.H3K27me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-Acr-CAG
⑧SRR1171568 若山 FI-SC(Fgf Induced Stem Cells):ChIPSeq.H3K4me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-Acr-CAG
⑨SRR1171569 若山 FI-SC(Fgf Induced Stem Cells):ChIPSeq.input Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-Acr-CAG
⑩SRR1171582 小保方 Low pH treated CD45 positive Cells:ChIPSeq.H3K27me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑪SRR1171583 小保方 Low pH treated CD45 positive Cells:ChIPSeq.H3K4me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑫SRR1171584 小保方 Low pH treated CD45 positive Cells:ChIPSeq.input Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑬SRR1171587 若山 STAP-SC(STAP derived Stem Cells):ChIPSeq.H3K27me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑭SRR1171588 若山 STAP-SC(STAP derived Stem Cells):ChIPSeq.H3K4me3 Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑮SRR1171589 若山 STAP-SC(STAP derived Stem Cells):ChIPSeq.input Oct3/4 expressing cells C57BL/6x129/Sv 129xB6-CAGヘテロ
⑯SRR1171592 丹羽研 Trophoblast Stem Cells:ChIPSeq.H3K27me3 CD1
⑰SRR1171593 丹羽研 Trophoblast Stem Cells:ChIPSeq.H3K4me3 CD1
⑱SRR1171594 丹羽研 Trophoblast Stem Cells:ChIPSeq.input CD1
伊藤さんが試料そのものが残っていたと言っているのは⑫のことです。生試料があったと言ってる。その他は残って無かったと。
>>
ただ、唯一やったのが、あ、GRASの方に残っていたChIP seq のinput 、しかもSTAP細胞のインプットはDNAとして残っていたので、それが10ページバワーポイントの、えーと、ところに書いてある、3番のアイテマイズして書いてあるところですけれども、これに関しては、えーと、30倍になるだけ、他のゲノムと同量となるまで読んでいます。そして、読んだ結果、この、同一であるということを、ま、認定したということで、それ以外に、それ以外に関しては、ま、残っていませんのでやってない、やってません。
本当に残ってなかったかな。私の直感は以下のようなものでしたけどね。今でも見れますね。
>>
桂会見での古田女史の質問に対する答えは以下でした。言い終わった後の彼のしぐさと表情は精神分析学の研究対象に好例かもしれない。(*ttps://www.youtube.com/watch?v=M9oJGioHvIQ 1:15:00から)
若山さんがリトラクション理由に挙げているのは⑤と⑪が逆になっているということです。そしてレターの図を見て誰にでも違いの気づけるところはNanogのグラフですが、これはTs.Marker さんが解析図を作ってくれています。

上が⑤で下が⑪です。これを見ると確かに小保方さんはグラフを間違えていますね。Nanogの多く発現している方がESです。これは論文のグラフを作ったときの間違いです。そしてリトラクション理由書の指摘事実は正しいということです。でもこれを以てリトラクトする必要はありませんよね。間違いは訂正したらいいだけです。共同著者の若山さんの行為はそういうものではない。論文そのものを全否定しているんです。自分で行った実験であるにも関わらず。他の共著者には説明も無い。話し合いもせずに一方的にリトラクトだと一人で騒いだ。何をしたのだということですね。そして小保方さんに騙されてES細胞でキメラを作らされてしまったのだというストーリーをマスコミを通して拡散した。そんなことマスコミを通すことか。共著者間で批判しあうべきことでしょう。共著者間の話し合いのテーブルにつけない理由があるわけです。我々は既にその理由を知ってるということです。
でも、今我々が解明しようとしているのはそのことではない。以下のように公共データ登録されている細胞⑪が「僕のマウス」ESであったという事実です。
>>
SRR1171583 Low pH treated CD45 positive Cells:ChIPSeq.H3K4me3 SAMN02393447 SRS559100 C57BL/6x129/Sv Oct3/4 expressing cells 2013/11/5 H.Obokata PRJNA238286 RIKEN 2014/2/13
小保方さんは2013/11/5に理研にデータを提出していて、理研はそれを2014/2/13に NCBI に登録したんです。その時の事務の担当者が誰かは分かっていません。
ここで小保方さんはこの細胞のマウス背景をC57BL/6x129/Svと書いています。まず最初に気づかなければならないことはこの細胞はAC129を作成したSTAP細胞ではないということです。雌雄の書き分けは小保方さんの間違いなのか事務担当者の間違いなのかは分かりませんが、AC129の実験で使われたマウスは「僕のマウス」の片親である129X1/Sv-CAG-GFPホモマウスだと報告書に書かれている。でも小保方さんが登録したのはF1マウスのSTAP細胞です。小保方さんはこのマウスをAC129作成時のSTAP細胞だなんて書いていませんよね。それどころかF1のSTAP細胞だと書いている。
そして検査の結果事実F1のマウス背景結果が出た。小保方さんがF1と書いているものからF1の結論が出て何がおかしいのだ。
報告書はAC129の背景を調べたらF1だったと分かったが、このAC129は129マウスで行ったものなのだからここからF1結果が出てきたらおかしいと言ってるわけです。AC129を作ったのは若山さんでしょ。自分で白毛の129/Svを小保方さんに渡してそのSTAP細胞からAC129を作ったんでしょ。そしたら中身がF1だったから小保方さんが自分にESを渡したのだと騒いでいるわけですけど、自分で中身をESに詰め替えたんでしょ。そうでないという証明が何もないじゃないか。何を一方的な嘘をついているんだよ。
それに関しては桂報告書も以下のような嘘を書いている。
>>
STAP 幹細胞 AC129 とされる細胞は 2012 年 8 月 13 日に作製されていることから、この 細胞はこれ以降に実施された実験に用いられたと判断した。公開データ再解析の結果に よれば、論文に記載された実験の中では Letter Fig.4 に使われた可能性が高く、また Letter 論文 Fig. 2i にも使われた可能性がある。しかし実験記録の不備から使用実験を 特定するには至らなかった。なお、Article のメソッドに、129/Sv carrying Rosa26-gfp からキメラ寄与能を有する STAP 幹細胞が樹立された、との記述があるが、129/Sv carrying Rosa26-gfp マウスは理研 CDB に導入された記録や飼育記録はないことから、 これは誤記と考えられ、若山氏の説明によればここで言及された STAP 幹細胞は AC129 であった可能性が高い。 「論文に記載された実験の中では Letter Fig.4 に使われた可能性が高く」などという推測は小保方さんが提出してる記載事実を見たらあり得ないと分かるでしょうに。小保方さんはF1だと書いているではないか。
これが本当に2012年の8月に提出されているSTAP細胞だったら必ず若山さんはF1マウスを小保方さんに渡しています。STAP細胞は継代保存ができない。維持培養期間は出来てからせいぜい1週間程度です。昔の細胞を提出するなんてことはできません。ChIPSeqに出した日付はGRASに残っているでしょう。生試料まであったというのだから、分析日付くらい分かっているはずだ。この頃小保方さんはたくさんのSTAP細胞を作らされたと証言していることを伊藤さんが記者会見の席で説明しましたね。他の人の実験のために使われたと言ってると。この細胞はAC129の実験のものだけではないでしょうよ。F1でも作らされている。それを小保方さんは提出しているんです。若山さんは実験ノートをつけていませんよね。よくもまあ、小保方さんのことが言えたものです。伊藤さんは小保方さんがかき集めたSTAP細胞が129/Svだったとでも思っていたんですかね。その実験は一回で終わってるでしょうに。
AC129作成時のSTAP細胞が「 Letter Fig.4 に使われた可能性」なんて全くありません。
- 2019/10/31(木) 22:32:20|
- AC129
-
-
| コメント:3